
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 14,821,008 – 14,821,139 |
| Length | 131 |
| Max. P | 0.999344 |

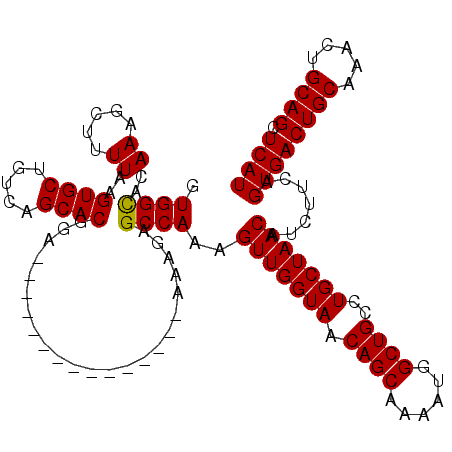
| Location | 14,821,008 – 14,821,113 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.64 |
| Mean single sequence MFE | -34.38 |
| Consensus MFE | -28.77 |
| Energy contribution | -28.63 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.69 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.52 |
| SVM RNA-class probability | 0.960599 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
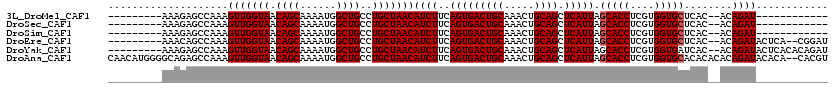
>3L_DroMel_CAF1 14821008 105 + 23771897 GUGGCAGAAAGCUUUUAAGUGCUGUCAGCACGGA---------------AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAU .((((((....(((((..((((.....))))..)---------------))))(((((..((((....))))....)))))..)))))).........((((((((.....)))).)))) ( -35.60) >DroSec_CAF1 55819 105 + 1 GUGGCACAAAGCUUUUAAGUGCUGUCAGCACGGA---------------AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAU .((((......(((((..((((.....))))..)---------------)))).))))..(((((((.((((......))))..))))))).......((((((((.....)))).)))) ( -34.90) >DroSim_CAF1 49008 105 + 1 GUGGCACAAAGCUUUUAAGUGCUGUCAGCACGGA---------------AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAU .((((......(((((..((((.....))))..)---------------)))).))))..(((((((.((((......))))..))))))).......((((((((.....)))).)))) ( -34.90) >DroEre_CAF1 56239 105 + 1 GUGGCACAAAGCUUUUAAGUGCUGUCAGCACGGA---------------AAACAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAU ..(((((...........)))))((.((((.((.---------------...((((((..((((....))))....)))))))))))).)).......((((((((.....)))).)))) ( -32.10) >DroYak_CAF1 59080 105 + 1 GUGGCACAAAGCUUUUAAGUGCUGUCAGCACGGA---------------AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAU .((((......(((((..((((.....))))..)---------------)))).))))..(((((((.((((......))))..))))))).......((((((((.....)))).)))) ( -34.90) >DroAna_CAF1 53264 120 + 1 GUGGUACAAAGCUUUUAAGUGCUGUCAGCACGAACGGCAACAACAUGGGGCAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAU .((((.(...(((((...((((.....))))....(....).....))))).).))))..(((((((.((((......))))..))))))).......((((((((.....)))).)))) ( -33.90) >consensus GUGGCACAAAGCUUUUAAGUGCUGUCAGCACGGA_______________AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAU .((((..((.....))..((((.....)))).......................))))..(((((((.((((......))))..))))))).......((((((((.....)))).)))) (-28.77 = -28.63 + -0.14)

| Location | 14,821,008 – 14,821,113 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.64 |
| Mean single sequence MFE | -29.87 |
| Consensus MFE | -27.35 |
| Energy contribution | -27.68 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.07 |
| Structure conservation index | 0.92 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.611238 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14821008 105 - 23771897 AUGAGCUGCAGUUUGCAGUCACUGAAGAUGUUAGCAGGCAGCCAUUUUGCUGUUACCAACUUUGGCUCUUU---------------UCCGUGCUGACAGCACUUAAAAGCUUUCUGCCAC .(((.((((.....)))))))..((((.((.((((((.(((.....))))))))).)).))))(((.((((---------------(..((((.....))))..)))))......))).. ( -28.30) >DroSec_CAF1 55819 105 - 1 AUGAGCUGCAGUUUGCAGUCACUGAAGAUGUUAGCAGGCAGCCAUUUUGCUGUUACCAACUUUGGCUCUUU---------------UCCGUGCUGACAGCACUUAAAAGCUUUGUGCCAC .(((.((((.....))))))).......((((((((((((((......)))))).(((....)))......---------------....))))))))((((...........))))... ( -29.30) >DroSim_CAF1 49008 105 - 1 AUGAGCUGCAGUUUGCAGUCACUGAAGAUGUUAGCAGGCAGCCAUUUUGCUGUUACCAACUUUGGCUCUUU---------------UCCGUGCUGACAGCACUUAAAAGCUUUGUGCCAC .(((.((((.....))))))).......((((((((((((((......)))))).(((....)))......---------------....))))))))((((...........))))... ( -29.30) >DroEre_CAF1 56239 105 - 1 AUGAGCUGCAGUUUGCAGUCACUGAAGAUGUUAGCAGGCAGCCAUUUUGCUGUUACCAACUUUGGCUGUUU---------------UCCGUGCUGACAGCACUUAAAAGCUUUGUGCCAC .(((.((((.....))))))).......((((((((((((((((..(((.......)))...))))))...---------------.)).))))))))((((...........))))... ( -31.70) >DroYak_CAF1 59080 105 - 1 AUGAGCUGCAGUUUGCAGUCACUGAAGAUGUUAGCAGGCAGCCAUUUUGCUGUUACCAACUUUGGCUCUUU---------------UCCGUGCUGACAGCACUUAAAAGCUUUGUGCCAC .(((.((((.....))))))).......((((((((((((((......)))))).(((....)))......---------------....))))))))((((...........))))... ( -29.30) >DroAna_CAF1 53264 120 - 1 AUGAGCUGCAGUUUGCAGUCACUGAAGAUGUUAGCAGGCAGCCAUUUUGCUGUUACCAACUUUGGCUCUGCCCCAUGUUGUUGCCGUUCGUGCUGACAGCACUUAAAAGCUUUGUACCAC ..(((((.......((((...(..(((.((.((((((.(((.....))))))))).)).)))..)..))))....((((((((((....).)).)))))))......)))))........ ( -31.30) >consensus AUGAGCUGCAGUUUGCAGUCACUGAAGAUGUUAGCAGGCAGCCAUUUUGCUGUUACCAACUUUGGCUCUUU_______________UCCGUGCUGACAGCACUUAAAAGCUUUGUGCCAC .(((.((((.....))))))).......((((((((((((((......)))))).(((....))).........................))))))))((((...........))))... (-27.35 = -27.68 + 0.33)



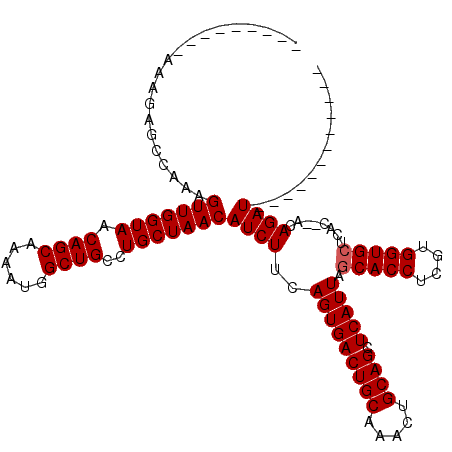
| Location | 14,821,042 – 14,821,139 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.54 |
| Mean single sequence MFE | -32.98 |
| Consensus MFE | -30.78 |
| Energy contribution | -30.95 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.18 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.53 |
| SVM RNA-class probability | 0.999344 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14821042 97 + 23771897 ---------AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAUUAGCACCUCGUGGUGCUCAC--ACAGAU------------ ---------...........(((((((.((((......))))..)))))))((((..(((((((((.....)))).)))))((((((....))))))...--..))))------------ ( -31.90) >DroSec_CAF1 55853 97 + 1 ---------AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAUUAGCACCUCGUGGUGCUCAC--ACAGAU------------ ---------...........(((((((.((((......))))..)))))))((((..(((((((((.....)))).)))))((((((....))))))...--..))))------------ ( -31.90) >DroSim_CAF1 49042 97 + 1 ---------AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAUUAGCACCUCGUGGUGCUCAC--ACAGAU------------ ---------...........(((((((.((((......))))..)))))))((((..(((((((((.....)))).)))))((((((....))))))...--..))))------------ ( -31.90) >DroEre_CAF1 56273 107 + 1 ---------AAACAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAUUAGCACCUCGUGGUGCUCAC--ACAGAUACUCA--CGGAU ---------......((...(((((((.((((......))))..)))))))((((..(((((((((.....)))).)))))((((((....))))))...--..)))).....--.)).. ( -34.60) >DroYak_CAF1 59114 109 + 1 ---------AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAUUAGCACCUCGUGGUGAUCAC--ACAGAUACUCACACAGAU ---------...(((.....(((((((.((((......))))..)))))))((((..(((((((((.....)))).)))))..((((....)))).....--..)))).)))........ ( -31.30) >DroAna_CAF1 53304 118 + 1 CAACAUGGGGCAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAUUAGCACCUCGUGGUGCACACACACAGAUACACA--CACGU ..((.((.((((((((((..((((....))))....)))))..)))))...((((..(((((((((.....)))).))))).(((((....)))))........)))).....--)).)) ( -36.30) >consensus _________AAAGAGCCAAAGUUGGUAACAGCAAAAUGGCUGCCUGCUAACAUCUUCAGUGACUGCAAACUGCAGCUCAUUAGCACCUCGUGGUGCUCAC__ACAGAU____________ ....................(((((((.((((......))))..)))))))((((..(((((((((.....)))).))))).(((((....)))))........))))............ (-30.78 = -30.95 + 0.17)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:49:30 2006