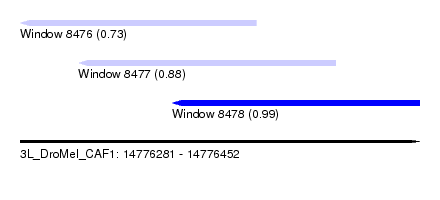
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 14,776,281 – 14,776,452 |
| Length | 171 |
| Max. P | 0.992090 |

| Location | 14,776,281 – 14,776,382 |
|---|---|
| Length | 101 |
| Sequences | 4 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 76.97 |
| Mean single sequence MFE | -22.05 |
| Consensus MFE | -15.80 |
| Energy contribution | -15.43 |
| Covariance contribution | -0.38 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.728409 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14776281 101 - 23771897 GG----CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGUGCAAAAAAAAAAAAGGAGGAAAACAUAGCAGGG--GUAAAGCGAA---AA .(----((.(((.((((((((.......))))((..(((((((.....)))))((.....))............))..))........)))).--))).)))...---.. ( -20.80) >DroSec_CAF1 19167 99 - 1 GG----CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGUGCAAAAAA-AAAAAGGAGGAAAACGUAGUAGGG--GAAAAGCGAA---A- .(----((.((((((((((((.......))))((((....))))....))))))))((((........-.............)))).......--....)))...---.- ( -19.50) >DroYak_CAF1 19268 104 - 1 GGCUGGCUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGUGCAAA------AAAGGAGGGGUACACAGUGGGGGAAAAAAGCGAGCGGAA .((((..((((((.....))))))..))))((((.((((((((.....))))).....((((...------.........))))...............))))))).... ( -28.80) >DroAna_CAF1 18823 91 - 1 AG----GUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCAGACAAACAGACGUAACGUGCAAAAAG-------AUACGAAAUACAGUAGGA--------CGGGCGGAA ..----...((((((((........(((((.....)))))........)))))))).(((.(......-------.(((........)))...--------.).)))... ( -19.09) >consensus GG____CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGUGCAAAAAA___AAAGGAGGAAAACACAGUAGGG__GAAAAGCGAA___AA .........((((((((........(((((.....)))))........)))))))).(.(((.........................))).).................. (-15.80 = -15.43 + -0.38)



| Location | 14,776,306 – 14,776,416 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 73.29 |
| Mean single sequence MFE | -27.91 |
| Consensus MFE | -14.99 |
| Energy contribution | -14.99 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.93 |
| SVM RNA-class probability | 0.884631 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14776306 110 - 23771897 AUCUACAUAUAUGUGG-C---GAUG--UACUGGGCUAUAUGG----CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGUGCAAAAAAAAAAAAGGAGGA .(((.(((....)))(-(---.(((--(.((((((....((.----.((((((.....))))))..))..))))))..(((((.....)))))...))))))............)))... ( -27.30) >DroSec_CAF1 19191 101 - 1 --------AUAUGUGG-G---GCUG--UACUGGGCUAUAUGG----CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGUGCAAAAAA-AAAAAGGAGGA --------.(((((..-(---((((--(((((((((....))----))(((((.....)))))...))))))..))))(((((.....)))))...))))).......-........... ( -25.90) >DroEre_CAF1 18702 95 - 1 ------------GCGG-GGUAGCUG--UAUAUGGCUAUAUGG----CUAUAUGUCUGGCAUACAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGCGGAAA------GCAGGAGGG ------------(((.-.(((((((--((((....)))))))----))))(((((((.....((.(((((.....))))).)).....)))))))..))).....------......... ( -30.20) >DroYak_CAF1 19298 102 - 1 ------------GCGGGGGUAGCUAGCUAUACGGCUAUAUGGCUGGCUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGUGCAAA------AAAGGAGGG ------------(((.(.(((((((((((((......)))))))))))))(((((((((((.......))))((((....))))....)))))))..).)))...------......... ( -35.80) >DroAna_CAF1 18845 91 - 1 --------AUAUA--------GCUU--UAUGGAUGUGGGUAG----GUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCAGACAAACAGACGUAACGUGCAAAAAG-------AUACG --------.....--------.(((--(.((.(((((.((..----((...(((((((((.................))))))))).))..)).).)))).)).))))-------..... ( -20.33) >consensus ________AUAUGCGG_G___GCUG__UAUAGGGCUAUAUGG____CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGUCUGACAAACAGACGUAACGUGCAAAAA____AAAGGAGGG .................................................((((((((........(((((.....)))))........))))))))........................ (-14.99 = -14.99 + 0.00)



| Location | 14,776,346 – 14,776,452 |
|---|---|
| Length | 106 |
| Sequences | 4 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 81.08 |
| Mean single sequence MFE | -34.58 |
| Consensus MFE | -19.39 |
| Energy contribution | -19.70 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.56 |
| Structure conservation index | 0.56 |
| SVM decision value | 2.31 |
| SVM RNA-class probability | 0.992090 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14776346 106 - 23771897 CAGA-GUUAUAUGCGUA--UAUAUAUCCGGGAUAUAUGUAUCUACAUAUAUGUGG-C---GAUG--UACUGGGCUAUAUGG----CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGU ....-.(((((((((((--((((((((...)))))))))))...((.((((((((-(---.(((--((......))))).)----)))))))).))))))))))(((((.....))))) ( -35.50) >DroSec_CAF1 19230 98 - 1 CAGACGAUAUAUGCGUA--UAUAUAUCCG-GAUAUAUGU--------AUAUGUGG-G---GCUG--UACUGGGCUAUAUGG----CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGU (((.(.(((((((((((--(((.......-.))))))))--------)))))).)-.---.)))--..((((((....((.----.((((((.....))))))..))..)))))).... ( -33.20) >DroEre_CAF1 18736 96 - 1 CAGA-GAUAUAUGCGUA--UAUAAGUCCG-GAUAUAUGU------------GCGG-GGUAGCUG--UAUAUGGCUAUAUGG----CUAUAUGUCUGGCAUACAAACAGUGCUCAGCUGU ....-.........(((--(....(.(((-((((((((.------------((.(-.((((((.--.....)))))).).)----)))))))))))))))))..(((((.....))))) ( -28.70) >DroYak_CAF1 19332 105 - 1 CAGA-GAUAUAUGCGUACAUAUAUAUCCG-GAUAUAUGU------------GCGGGGGUAGCUAGCUAUACGGCUAUAUGGCUGGCUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGU ....-..(((((((((((((((((.....-.))))))))------------))(((.(((((((((((((......)))))))))))))...))).))))))).(((((.....))))) ( -40.90) >consensus CAGA_GAUAUAUGCGUA__UAUAUAUCCG_GAUAUAUGU____________GCGG_G___GCUG__UACAGGGCUAUAUGG____CUAUAUGUCUGGCAUAUAAACAGUGCUCAGCUGU .......(((((((.....((((((((...)))))))).................................(((.((((((....)))))))))..))))))).(((((.....))))) (-19.39 = -19.70 + 0.31)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:48:19 2006