
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 14,714,331 – 14,714,465 |
| Length | 134 |
| Max. P | 0.924783 |

| Location | 14,714,331 – 14,714,445 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 80.56 |
| Mean single sequence MFE | -41.52 |
| Consensus MFE | -27.66 |
| Energy contribution | -28.42 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.35 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.558107 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14714331 114 + 23771897 GUUGUGG---CUUGCUGAUUUCGAACAAUAUGACCGGCGGCAGUGACAUUGGUCACGCUGGAGCGCUGAUUUGUGCGCGUCAAGGUGACCGUCACCUCGGAACCCAGUCCGGCAAAC (((...(---((.((((.((((((......((((...((((.(((((....)))))))))..((((........)))))))).((((.....))))))))))..))))..))).))) ( -40.70) >DroPse_CAF1 49911 111 + 1 GCU---G---AUUGUUGGUUCCGUACAAUAUGACCAGCGGCGGUGACACUGGUGACGCUGGCGCGCUGAUUGGUUCGCGUCAACGUAACCGUCACCUCAGAGCCCAAACCGGCAAAC .((---(---((((((((((...........))))))))).((((((...(((.(((.(((((((((....))..))))))).))).))))))))))))).(((......))).... ( -41.70) >DroGri_CAF1 51386 111 + 1 A---GGG---UUUGCUGAUUCCUAACAAUAUGACCGGCGGCAGUCACAUUUGUGACGCUGGCACGCUGAUUGGUGCGCGUCAAUGUAACUGUCACCUCAGACCCAAGGCCGGCAAAG .---...---(((((((...(((.......(((..((.((((((.((((...((((((..((((........)))))))))))))).)))))).)))))......))).))))))). ( -38.62) >DroWil_CAF1 219262 117 + 1 GAUGUGGAUGAUUGCUGAUUCCGUACAAUAUGACCAGAGGCAGUGACAUUGGUUACCCUGUCGCGUUGAUUGGUACGCGUGAGAGUAACGGUCACCUCAGAGCCUAGACCGGCAAAG ...........((((((....((((...)))).....((((.((((((..((....))))))))(.((((((.(((.(....).))).)))))).).....))))....)))))).. ( -31.50) >DroMoj_CAF1 58071 111 + 1 A---CGG---UUUGCUGAUUCCUAACAAUAUGACCGGCGGCUGUGACAUUGGUUACGCUGGCGCGCUGAUUGGUGCGCGUCAGAGUAACUGUCACCUCAGAGCCAAGGCCAGCAAAG .---...---(((((((...(((..(.....)...(((.(..((((((...(((((.(((((((((........))))))))).)))))))))))..)...))).))).))))))). ( -45.60) >DroAna_CAF1 20271 112 + 1 --UGUGG---CUUGCUGAUUCCUUACAAUAUGACCGGCGGCUGUGACACUCGUCACGCUGGCACGCUGAUUGGUGCGUGUCAGGGUGACGGUCACCUCGGAGCCGAGGCCAGCAAAU --(((((---(((((((.................))))(((((((((...((((((.(((((((((........))))))))).))))))))))).....)))).)))))).))... ( -51.03) >consensus G_U_UGG___AUUGCUGAUUCCGAACAAUAUGACCGGCGGCAGUGACAUUGGUCACGCUGGCGCGCUGAUUGGUGCGCGUCAAAGUAACCGUCACCUCAGAGCCAAGGCCGGCAAAG ...........((((((........(.....)......(((.(((((...((((((..((((((((........))))))))..)))))))))))......))).....)))))).. (-27.66 = -28.42 + 0.75)

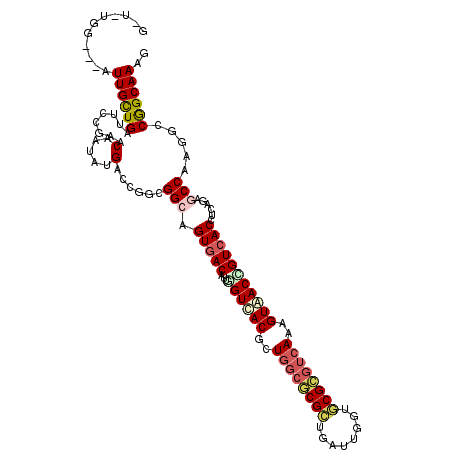
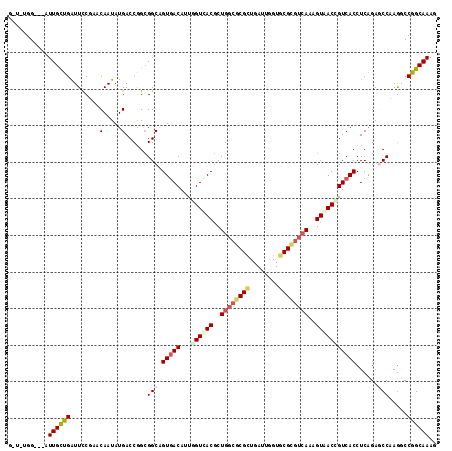
| Location | 14,714,331 – 14,714,445 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 80.56 |
| Mean single sequence MFE | -39.83 |
| Consensus MFE | -30.85 |
| Energy contribution | -31.00 |
| Covariance contribution | 0.15 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.858934 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14714331 114 - 23771897 GUUUGCCGGACUGGGUUCCGAGGUGACGGUCACCUUGACGCGCACAAAUCAGCGCUCCAGCGUGACCAAUGUCACUGCCGCCGGUCAUAUUGUUCGAAAUCAGCAAG---CCACAAC ((((((..((((((....((.((((((((((((((.((.((((........)))))).)).))))))...))))))..)))))))).........(....).)))))---)...... ( -42.00) >DroPse_CAF1 49911 111 - 1 GUUUGCCGGUUUGGGCUCUGAGGUGACGGUUACGUUGACGCGAACCAAUCAGCGCGCCAGCGUCACCAGUGUCACCGCCGCUGGUCAUAUUGUACGGAACCAACAAU---C---AGC ....((.((((..((.((((.(((((((((.((((((.((((..........)))).)))))).)))...))))))(((...))).........)))).))...)))---)---.)) ( -37.10) >DroGri_CAF1 51386 111 - 1 CUUUGCCGGCCUUGGGUCUGAGGUGACAGUUACAUUGACGCGCACCAAUCAGCGUGCCAGCGUCACAAAUGUGACUGCCGCCGGUCAUAUUGUUAGGAAUCAGCAAA---CCC---U .............((((((((((((.((((((((((((((((((((.....).))))..))))))...))))))))).))))..((.((....)).)).))))...)---)))---. ( -38.90) >DroWil_CAF1 219262 117 - 1 CUUUGCCGGUCUAGGCUCUGAGGUGACCGUUACUCUCACGCGUACCAAUCAACGCGACAGGGUAACCAAUGUCACUGCCUCUGGUCAUAUUGUACGGAAUCAGCAAUCAUCCACAUC ....(((((...((((.....((((((.((((((((..(((((........)))))..))))))))....)))))))))))))))..........(((...........)))..... ( -33.70) >DroMoj_CAF1 58071 111 - 1 CUUUGCUGGCCUUGGCUCUGAGGUGACAGUUACUCUGACGCGCACCAAUCAGCGCGCCAGCGUAACCAAUGUCACAGCCGCCGGUCAUAUUGUUAGGAAUCAGCAAA---CCG---U .(((((((.(((((((...(..(((((((((((.(((.(((((........))))).))).)))))...))))))..).))))...........)))...)))))))---...---. ( -40.70) >DroAna_CAF1 20271 112 - 1 AUUUGCUGGCCUCGGCUCCGAGGUGACCGUCACCCUGACACGCACCAAUCAGCGUGCCAGCGUGACGAGUGUCACAGCCGCCGGUCAUAUUGUAAGGAAUCAGCAAG---CCACA-- .(((((((((((((....))))))(((((((((.(((.(((((........))))).))).)))))(.(((.......))))))))..............)))))))---.....-- ( -46.60) >consensus CUUUGCCGGCCUGGGCUCUGAGGUGACAGUUACCCUGACGCGCACCAAUCAGCGCGCCAGCGUAACCAAUGUCACUGCCGCCGGUCAUAUUGUAAGGAAUCAGCAAA___CCA_A_C ....((((((...(((.....((((((((((((.(((.(((((........))))).))).)))))...))))))))))))))))....((((.........))))........... (-30.85 = -31.00 + 0.15)

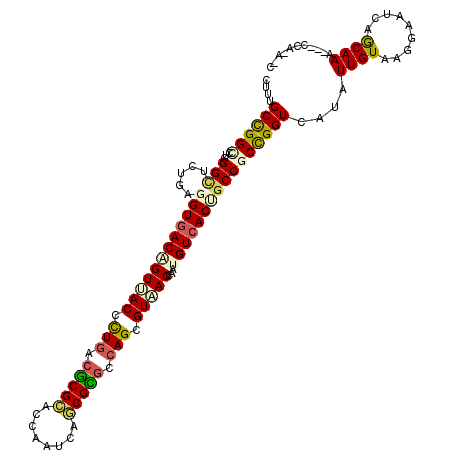

| Location | 14,714,365 – 14,714,465 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 79.80 |
| Mean single sequence MFE | -41.82 |
| Consensus MFE | -24.75 |
| Energy contribution | -25.76 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.591701 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14714365 100 + 23771897 CGGCAGUGACAUUGGUCACGCUGGAGCGCUGAUUUGUGCGCGUCAAGGUGACCGUCACCUCGGAACCCAGUCCGGCAAACGAAACAGCUGGCG---UGGA---UGG (((..(((((...((((((..((..((((........))))..))..))))))))))).)))....(((..(((((..........)))))..---))).---... ( -37.70) >DroVir_CAF1 50542 100 + 1 CGGCAGUGACAUUGGUUACGCUGGCGCGCUGAUUGGUGCGCGUCAAGGUAACUGUCACCUCAGAGCCAAGGCCAGCAAAGGAGACAGAUGGUG---UGGU---UGC ..((.((((((...(((((..((((((((........))))))))..))))))))))).....(((((..((((.....(....)...)))).---))))---))) ( -39.50) >DroGri_CAF1 51417 100 + 1 CGGCAGUCACAUUUGUGACGCUGGCACGCUGAUUGGUGCGCGUCAAUGUAACUGUCACCUCAGACCCAAGGCCGGCAAAGGAGACGGACGGUG---UUGU---UGU .((((((.((((...((((((..((((........)))))))))))))).))))))..........(((.((((.(...(....).).)))).---))).---... ( -36.70) >DroWil_CAF1 219299 99 + 1 AGGCAGUGACAUUGGUUACCCUGUCGCGUUGAUUGGUACGCGUGAGAGUAACGGUCACCUCAGAGCCUAGACCGGCAAAGGAGACAGCUGGGG---GAGU----CG .(((.((((((..((....))))))))..((((((.(((.(....).))).))))))((((((.(((......)))...(....)..))))))---..))----). ( -36.70) >DroMoj_CAF1 58102 100 + 1 CGGCUGUGACAUUGGUUACGCUGGCGCGCUGAUUGGUGCGCGUCAGAGUAACUGUCACCUCAGAGCCAAGGCCAGCAAAGGACACAGACGGCG---UCGU---UGC .((((((((((...(((((.(((((((((........))))))))).))))))))))).....)))).......((((..(((.(.....).)---)).)---))) ( -42.30) >DroAna_CAF1 20303 106 + 1 CGGCUGUGACACUCGUCACGCUGGCACGCUGAUUGGUGCGUGUCAGGGUGACGGUCACCUCGGAGCCGAGGCCAGCAAAUGAGACGGCCGGCGGUGUGGCCGGUGG ..((((((((...((((((.(((((((((........))))))))).))))))))))(((((....))))).))))........(.((((((......)))))).) ( -58.00) >consensus CGGCAGUGACAUUGGUUACGCUGGCGCGCUGAUUGGUGCGCGUCAAGGUAACUGUCACCUCAGAGCCAAGGCCAGCAAAGGAGACAGACGGCG___UGGU___UGG .(((.(((((...((((((..((((((((........))))))))..)))))))))))......)))...((((..............)))).............. (-24.75 = -25.76 + 1.00)



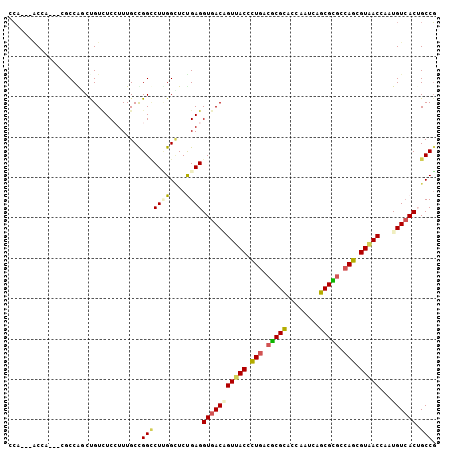
| Location | 14,714,365 – 14,714,465 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 79.80 |
| Mean single sequence MFE | -37.47 |
| Consensus MFE | -26.99 |
| Energy contribution | -26.77 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.924783 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14714365 100 - 23771897 CCA---UCCA---CGCCAGCUGUUUCGUUUGCCGGACUGGGUUCCGAGGUGACGGUCACCUUGACGCGCACAAAUCAGCGCUCCAGCGUGACCAAUGUCACUGCCG (((---(((.---.((.(((......))).)).))).)))....((.((((((((((((((.((.((((........)))))).)).))))))...))))))..)) ( -36.50) >DroVir_CAF1 50542 100 - 1 GCA---ACCA---CACCAUCUGUCUCCUUUGCUGGCCUUGGCUCUGAGGUGACAGUUACCUUGACGCGCACCAAUCAGCGCGCCAGCGUAACCAAUGUCACUGCCG (((---...(---((.....((((.((((....(((....)))..)))).))))(((((.(((.(((((........))))).))).)))))...)))...))).. ( -32.40) >DroGri_CAF1 51417 100 - 1 ACA---ACAA---CACCGUCCGUCUCCUUUGCCGGCCUUGGGUCUGAGGUGACAGUUACAUUGACGCGCACCAAUCAGCGUGCCAGCGUCACAAAUGUGACUGCCG ...---....---........(((.((((.(((.......)))..)))).)))(((((((((((((((((((.....).))))..))))))...)))))))).... ( -33.40) >DroWil_CAF1 219299 99 - 1 CG----ACUC---CCCCAGCUGUCUCCUUUGCCGGUCUAGGCUCUGAGGUGACCGUUACUCUCACGCGUACCAAUCAACGCGACAGGGUAACCAAUGUCACUGCCU .(----((..---........(((.((((.(((......)))...)))).))).((((((((..(((((........)))))..))))))))....)))....... ( -33.10) >DroMoj_CAF1 58102 100 - 1 GCA---ACGA---CGCCGUCUGUGUCCUUUGCUGGCCUUGGCUCUGAGGUGACAGUUACUCUGACGCGCACCAAUCAGCGCGCCAGCGUAACCAAUGUCACAGCCG (((---(.((---(((.....)))))..)))).((((((......)))(((((((((((.(((.(((((........))))).))).)))))...)))))).))). ( -40.30) >DroAna_CAF1 20303 106 - 1 CCACCGGCCACACCGCCGGCCGUCUCAUUUGCUGGCCUCGGCUCCGAGGUGACCGUCACCCUGACACGCACCAAUCAGCGUGCCAGCGUGACGAGUGUCACAGCCG ....(((((........)))))........((((((((((....))))))(((((((((.(((.(((((........))))).))).))))))...))).)))).. ( -49.10) >consensus CCA___ACCA___CGCCAGCUGUCUCCUUUGCCGGCCUUGGCUCUGAGGUGACAGUUACCCUGACGCGCACCAAUCAGCGCGCCAGCGUAACCAAUGUCACUGCCG .................................(((((((....))))(((((((((((.(((.(((((........))))).))).)))))...)))))).))). (-26.99 = -26.77 + -0.22)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:47:31 2006