
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 14,509,382 – 14,509,515 |
| Length | 133 |
| Max. P | 0.653681 |

| Location | 14,509,382 – 14,509,488 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 75.93 |
| Mean single sequence MFE | -38.29 |
| Consensus MFE | -22.09 |
| Energy contribution | -24.37 |
| Covariance contribution | 2.28 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.58 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 14509382 106 - 23771897 -----UGUCGGUAGUCGGUAGUCGUUACGAUCCGAUUCUGGCCGAGUCGGGUGGUACGAUCUGCCAUCUCAGAUACACCAU---CACCAU--CACCGAUCUCUGGCAAUACGGCUG -----((((((..((((((((((((.((.(((((((((.....))))))))).)))))).)))))......(((.....))---).....--....)))..))))))......... ( -38.70) >DroSec_CAF1 5984 105 - 1 ------GUCGGUAGUCGGUAGUGGUUACGAUCCGAUUCUGGCUGAGUCGAGUGGUACGAUCUGCGAUCUCCGAUACACCAU---CACCAC--CACCGAUCUCUGGCAAUACGGCCG ------(((((..((((((.(((((...((((((((((.....)))))).(..(......)..)))))...(((.....))---))))))--.))))))..))))).......... ( -37.70) >DroSim_CAF1 6158 107 - 1 ------GUCGGUAGUCGGUAGUCGUUACGAUCCGAUUCUGGCUGAGUCGGGUGGUACGAUCAGCGAUCUCCGAUACACCAU---CACCACAUCACCGAUCUCUGGCAAUACGGCCG ------(((((..((((.(.(((((.((.(((((((((.....))))))))).))))))).).))))..))))).......---...................(((......))). ( -39.80) >DroEre_CAF1 6299 109 - 1 -----UGUCGGUAGUCGGUAGUCGAUACGAUUCGAUUCUGGCUGAGUCGGGUGGUGCGAUCUGCCAUCUGCGAUCUGCAAUCUACACCAG--CAACGAUCUCUGGCAAUCCGGCCG -----.(((((..(((((((((((.(((.(((((((((.....))))))))).)))))).)))))......(((((((...........)--))..))))...)))...))))).. ( -40.50) >DroYak_CAF1 6000 112 - 1 GUCGAUGUCGGUAGUCGGUAGUCGUUACGAUCCGAUUCUGGCUGAGUCGAGUGGUGCGAUCUGCGAUCUGCGAUCUCCUA--UACACCAG--CAGCGAUCUCUGGCAAUACGGCCG ((((.((((((..((((((((((((.((.(..((((((.....))))))..).)))))).))))...((((.........--.......)--)))))))..))))))...)))).. ( -38.69) >DroAna_CAF1 5871 90 - 1 ---------GAUAGUCGUU--UCGAUCCGAUCCGAAUCGGGUCUGGCCGGGUGGGGCGAUCUCCG-UAUAAAAAACCCCAA-----------UACCCAG--CCGGCUGUU-UGCCG ---------(((((((...--..((((((((....)))))))).(((.((((((((.........-.........))))..-----------.)))).)--)))))))))-..... ( -34.37) >consensus ______GUCGGUAGUCGGUAGUCGUUACGAUCCGAUUCUGGCUGAGUCGGGUGGUACGAUCUGCGAUCUCCGAUACACCAU___CACCAC__CACCGAUCUCUGGCAAUACGGCCG ......(((((..(((((((((((.(((.(((((((((.....))))))))).)))))).)))).(((...))).....................))))..))))).......... (-22.09 = -24.37 + 2.28)

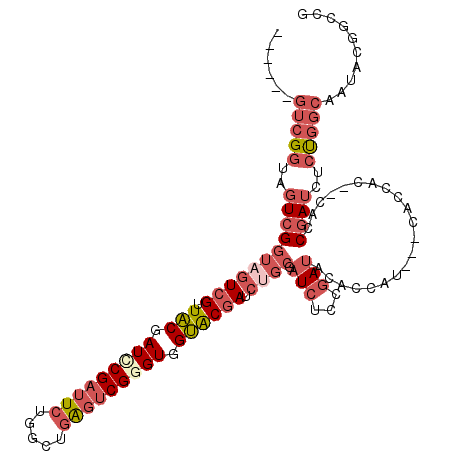
| Location | 14,509,414 – 14,509,515 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 76.34 |
| Mean single sequence MFE | -37.18 |
| Consensus MFE | -22.03 |
| Energy contribution | -23.78 |
| Covariance contribution | 1.75 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.653681 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
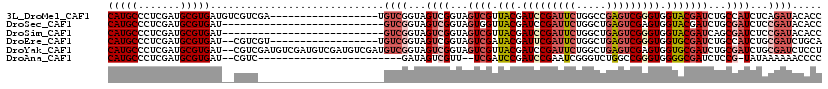
>3L_DroMel_CAF1 14509414 101 - 23771897 CAUGCCCUCGAUGCGUGAUGUCGUCGA------------------UGUCGGUAGUCGGUAGUCGUUACGAUCCGAUUCUGGCCGAGUCGGGUGGUACGAUCUGCCAUCUCAGAUACACC .......(((((((.....).))))))------------------((((.(.((..(((((((((.((.(((((((((.....))))))))).)))))).)))))..))).)))).... ( -36.90) >DroSec_CAF1 6016 92 - 1 CAUGCCCUCGAUGCGUGAU---------------------------GUCGGUAGUCGGUAGUGGUUACGAUCCGAUUCUGGCUGAGUCGAGUGGUACGAUCUGCGAUCUCCGAUACACC (((((.......))))).(---------------------------(((((..((((.((((.((.((.(..((((((.....))))))..).)))).).)))))))..)))))).... ( -30.90) >DroSim_CAF1 6192 92 - 1 CAUGCCCUCGAUGCGUGAU---------------------------GUCGGUAGUCGGUAGUCGUUACGAUCCGAUUCUGGCUGAGUCGGGUGGUACGAUCAGCGAUCUCCGAUACACC (((((.......))))).(---------------------------(((((..((((.(.(((((.((.(((((((((.....))))))))).))))))).).))))..)))))).... ( -40.80) >DroEre_CAF1 6334 99 - 1 CAUGCCCUCGAUGCGUGAU--CGUCGU------------------UGUCGGUAGUCGGUAGUCGAUACGAUUCGAUUCUGGCUGAGUCGGGUGGUGCGAUCUGCCAUCUGCGAUCUGCA ...........((((.(((--(..((.------------------...))((((..((((((((.(((.(((((((((.....))))))))).)))))).)))))..)))))))))))) ( -39.30) >DroYak_CAF1 6033 117 - 1 CAUGCCCUCGAUGCGUGAU--CGUCGAUGUCGAUGUCGAUGUCGAUGUCGGUAGUCGGUAGUCGUUACGAUCCGAUUCUGGCUGAGUCGAGUGGUGCGAUCUGCGAUCUGCGAUCUCCU ...(((((((((...(((.--(((((((((((....)))))))))))))).(((((((.(((((........)))))))))))).)))))).)))..((((.((.....)))))).... ( -45.60) >DroAna_CAF1 5894 90 - 1 CAUGCCCUCGAUGCGUGAU--CGUC------------------------GAUAGUCGUU--UCGAUCCGAUCCGAAUCGGGUCUGGCCGGGUGGGGCGAUCUCCG-UAUAAAAAACCCC ..........(((((.(((--((((------------------------..((.(((.(--..((((((((....))))))))..).))).)).)))))))..))-))).......... ( -29.60) >consensus CAUGCCCUCGAUGCGUGAU__CGUC_____________________GUCGGUAGUCGGUAGUCGUUACGAUCCGAUUCUGGCUGAGUCGGGUGGUACGAUCUGCGAUCUCCGAUACACC (((((.......))))).............................((((...((((...((((.(((.(((((((((.....))))))))).)))))))...))))...))))..... (-22.03 = -23.78 + 1.75)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:45:34 2006