
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 1,522,704 – 1,522,820 |
| Length | 116 |
| Max. P | 0.758177 |

| Location | 1,522,704 – 1,522,820 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 79.49 |
| Mean single sequence MFE | -32.50 |
| Consensus MFE | -24.21 |
| Energy contribution | -23.97 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.37 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.758177 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
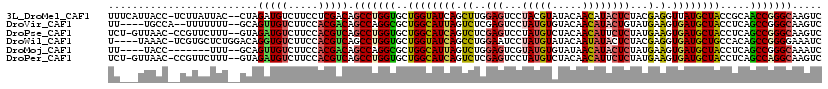
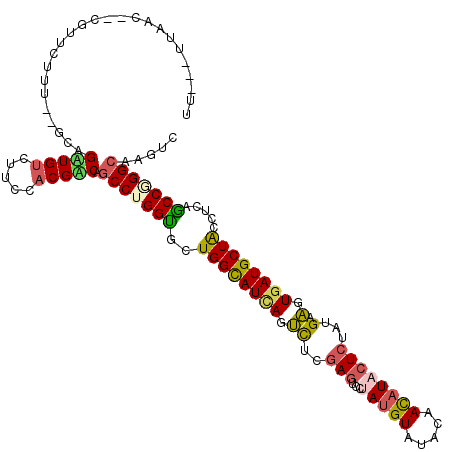
>3L_DroMel_CAF1 1522704 116 + 23771897 UUUCAUUACC-UCUUAUUAC--CUAGAUGUCUUCCUCGACAGCCUGGUGCUGGUAUCAGCUUGGAGUCCUACGUAUACAACAUACUCUACGAGGUUAUGCUACCGCAACCGGGCAAGUC ..........-.........--.....((((......))))(((((((((.(((...(((....((.(((.((((............)))))))))..)))))))).)))))))..... ( -28.90) >DroVir_CAF1 3808 111 + 1 UU----UGCCA--UUUUUUU--GCAGUUGUCUUCCACGACAGCCAGGCGCUGGCAUUAGUCUCGAGUCCUAUGUGUACAACAUACUGUAUGAAGUGAUGCUACCUCAGCCGGGCAAGUC ((----((((.--.......--...((((((......))))))..(((..((((((((.(.(((..(..(((((.....)))))..)..)))).)))))))).....))).)))))).. ( -31.90) >DroPse_CAF1 1249 115 + 1 UCU-GUUAAC-CCGUUCUUU--GUAGAUGUCUUCCACGUCAGCCUGGUGCUGGCAUCAGUCUCGAGUCCUAUGUCUACAACAUUCUCUAUGAAGUGAUGCUACCUCAGCCGGGCAAGUC ...-......-.........--...(((((.....))))).(((((((..((((((((.((..(((....((((.....)))).)))...))..)))))))).....)))))))..... ( -32.30) >DroWil_CAF1 1659 114 + 1 U----UAAAC-UCGUGCUCUGGACAGGUGUCUUCCACGUCAGCCUGGUGCUGGUAUCAGCCUGGAAUCCUAUGUAUACAAUAUACUCUACGAGGUGAUGCUGCCACAGCCGGGGAAAUC .----....(-(((.(((.(((...((((((..((.((((((.((((((....)))))).))).........(((((...)))))...))).)).)))))).))).)))))))...... ( -35.00) >DroMoj_CAF1 2637 106 + 1 UU----UACC-------UUU--GCAGUUGUCUUCCACGACAGCCAGGCGCUGGCAUUAGUCUGGAGUCGUAUGUGUAUAACAUACUCUAUGAAGUGAUGCUACCUCAGCCGGGCAAAUC ..----....-------(((--((.((((((......))))))..(((..((((((((.((((((((...((((.....)))))))))).))..)))))))).....)))..))))).. ( -33.90) >DroPer_CAF1 1248 115 + 1 UCU-GUUAAC-CCGUUCUUU--GUAGAUGUCUUCCACGUCAGCCUGGUGCUGGCAUCAGUCUCGAGUCCUAUGUCUACAACAUUCUCUAUGAAGUGAUGCUACCUCAGCCAGGCAAGUC ...-......-.........--...(((((.....))))).(((((((..((((((((.((..(((....((((.....)))).)))...))..)))))))).....)))))))..... ( -33.00) >consensus UU___UUAAC__CGUUCUUU__GCAGAUGUCUUCCACGACAGCCUGGUGCUGGCAUCAGUCUCGAGUCCUAUGUAUACAACAUACUCUAUGAAGUGAUGCUACCUCAGCCGGGCAAGUC .........................(((((.....))))).(((((((..((((((((.((..(((...(((((.....))))))))...).).)))))))).....)))))))..... (-24.21 = -23.97 + -0.24)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:43:59 2006