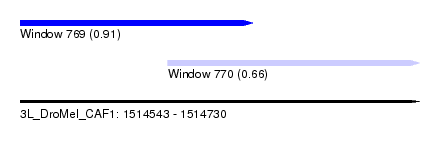
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 1,514,543 – 1,514,730 |
| Length | 187 |
| Max. P | 0.912658 |

| Location | 1,514,543 – 1,514,652 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 82.08 |
| Mean single sequence MFE | -45.28 |
| Consensus MFE | -31.10 |
| Energy contribution | -32.05 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.36 |
| Mean z-score | -3.07 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.08 |
| SVM RNA-class probability | 0.912658 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
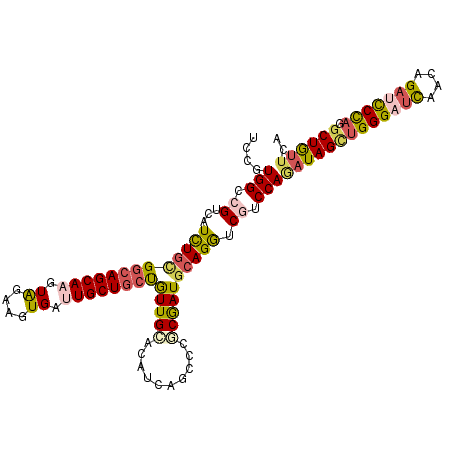
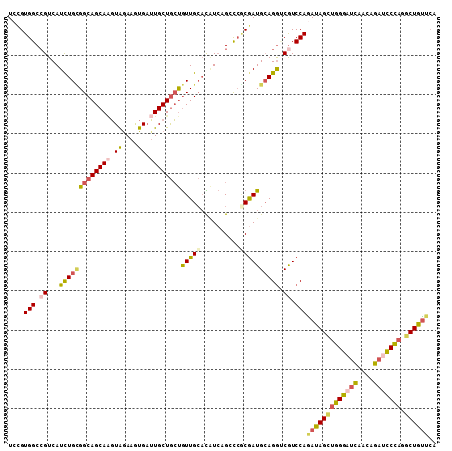
>3L_DroMel_CAF1 1514543 109 + 23771897 UCCGUGGCCGUCAUCUGCGGCAGCAUGUAGAAGUGAUUGCUGCUGUUGCACAUCAGGCCGCGAUGCAGAUCGUCCAGAUAGCUGGGAUCAACAGAUCCCAGGCUGUUCA ..(((((((...((.((((((((((.((((......)))))))))))))).))..)))))))..(((((((.....)))..((((((((....)))))))).))))... ( -49.60) >DroPse_CAF1 24288 109 + 1 UCUGUGGCCGUCAUUUGGGGCAGCAGUUGGAAGUGCAUGCUAGCAUUGGCUCUAAGCUGCCGAUGAAGGCCAUCCAGAUAGUCGGGGCUGAUGGAGUCUCUGCUAUUGA ...((((((.(((((...((((((..((((((((((......)))))...)))))))))))))))).))))))...((((((.((((((.....)))))).)))))).. ( -44.70) >DroSec_CAF1 19581 109 + 1 UCCGUGGCCGUCAUCUGCGGCAGCAAGUAGAAAUGAUUGCUGCUGUUGCACAUCAGCCCGCGAUGCAGGUCGUCCAAAUAGCUGGGAUCAACAGAUCCCACGCUGUUCA ....(((.((..((((((((((((((.((....)).))))))))(((((..........))))))))))))).)))(((((((((((((....))))))).)))))).. ( -46.00) >DroSim_CAF1 20087 109 + 1 UCCGUGGCCGUCAUCUGCGGCAGCAAGUAAAAGUGAUUGCUGCUGUUGCACAUCAGCCCGCGAUGCAGGUCGUCCAAAUAGCUGGGAUCAACAGAUCCCACGCUGUUCA ..(((((.(...((.((((((((((.((((......)))))))))))))).))..).))))).....((....)).(((((((((((((....))))))).)))))).. ( -43.90) >DroEre_CAF1 21133 109 + 1 UCGGUGGCCGUCAUCUGUGGCAGCAAGUAGAAGUGAUUGCUGCUGUUGCACAUCAGGCCACAAUGCAGGGCGUCCAGAUAGCUGGGAUCAACAGAUCCUAGACUGUGCA ...((((((...((.((..((((((.((((......))))))))))..)).))..))))))..(((.((....))..((((((((((((....)))))))).))))))) ( -46.70) >DroYak_CAF1 19316 109 + 1 UCCGUGGCAGUCAUCUGCGGCAGCAAGUAGAAGUGAUUGCUGCUGUUGCAUGUCAGUCCACGAUGCAGGGCGUCCAGAUAGCUGGGUUCAACAGAUCCCAGGCUGCUCA ...(((((.(.((((.((((((((((..........)))))))))).).))).).).))))(((((...)))))..((((((((((.((....)).)))).)))).)). ( -40.80) >consensus UCCGUGGCCGUCAUCUGCGGCAGCAAGUAGAAGUGAUUGCUGCUGUUGCACAUCAGCCCGCGAUGCAGGUCGUCCAGAUAGCUGGGAUCAACAGAUCCCAGGCUGUUCA ....(((.((...(((((((((((((..........))))))))(((((..........)))))))))).)).)))(((((((((((((....))))))).)))))).. (-31.10 = -32.05 + 0.95)

| Location | 1,514,612 – 1,514,730 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 80.96 |
| Mean single sequence MFE | -41.55 |
| Consensus MFE | -24.84 |
| Energy contribution | -25.60 |
| Covariance contribution | 0.76 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.662398 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
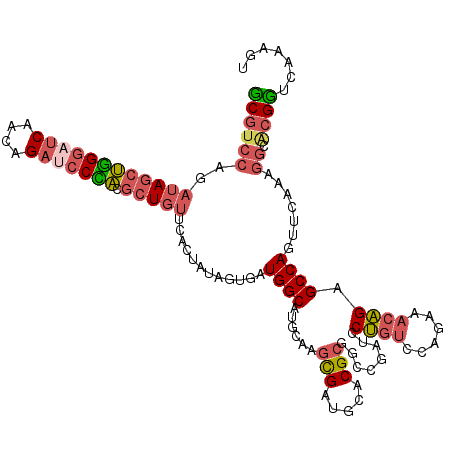
>3L_DroMel_CAF1 1514612 118 + 23771897 UCGUCCAGAUAGCUGGGAUCAACAGAUCCCAGGCUGUUCACUAUAGUGAUGGCAUGCAAGCGAUGCACGCGGCCGAUCCUGUCCAGAAACAGAGCCAGCUCGAAAGCGCGGUCAAAGU (((.((..((((((((((((....))))))).)))))((((....))))..((.((((.....)))).)))).)))..((((......)))).(((.((.(....).)))))...... ( -40.80) >DroVir_CAF1 21747 116 + 1 GCAUCCAGAAAUCCUGCACCAGUGGCGCCGGCGCUGUGCACUAUGCUGAUGGCCUGCAGCUGAUGGACGCGUCCGAUGCGUGCCAAGAACUGCGCCAUAUUC--GUGCCGUUCACAGC (((.(((...((..(((((.((((.(...).)))))))))..)).....)))..))).((((.(((((((((...)))))).))).((((.((((.......--)))).)))).)))) ( -39.80) >DroSec_CAF1 19650 118 + 1 UCGUCCAAAUAGCUGGGAUCAACAGAUCCCACGCUGUUCACUAUAGUAAUGGCAUGCAAGCGAUGCACGCGGCCGAUCCUGUCCAGAAACAGAGCCAGUUCAAAGGCACGGUCAAAGU .......(((((((((((((....))))))).)))))).............((.((((.....)))).))(((((...((((......)))).(((........))).)))))..... ( -39.60) >DroSim_CAF1 20156 118 + 1 UCGUCCAAAUAGCUGGGAUCAACAGAUCCCACGCUGUUCACUAUAGUAAUGGCAUGCAAGCGAUGCACGCGGCCGAUCCUGUCCAAAAACAGAGCCAGUUCAAAGGCACGGUCAAAGU .......(((((((((((((....))))))).)))))).............((.((((.....)))).))(((((...((((......)))).(((........))).)))))..... ( -39.60) >DroEre_CAF1 21202 118 + 1 GCGUCCAGAUAGCUGGGAUCAACAGAUCCUAGACUGUGCACUAUAGUGAUGGCAUGCAAGCGAUGCACGCGGCCAAUCCUGUUCAGAAACAGAGCCAGUUCAUUGGGACGCGCAAAGU ((((((..((((((((((((....)))))))).))))......((((((((((..((..(((.....))).)).....(((((....))))).))))..))))))))))))....... ( -45.00) >DroYak_CAF1 19385 118 + 1 GCGUCCAGAUAGCUGGGUUCAACAGAUCCCAGGCUGCUCACUAUAGUGAUGGCAUUCAAGCGAUGCACGCGGCCGAUCCUGUCCAGAAACAGAGCCAGUUCAUGGGGACGCUCAAAGC ((((((..((((((((........((((...((((((((((....))))..(((((.....)))))..))))))))))((((......))))..)))))).))..))))))....... ( -44.50) >consensus GCGUCCAGAUAGCUGGGAUCAACAGAUCCCACGCUGUUCACUAUAGUGAUGGCAUGCAAGCGAUGCACGCGGCCGAUCCUGUCCAGAAACAGAGCCAGUUCAAAGGCACGGUCAAAGU ((((((..((((((((((((....))))))).)))))............((((......(((.....)))........((((......)))).)))).......)).))))....... (-24.84 = -25.60 + 0.76)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:43:57 2006