
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,980,585 – 13,980,742 |
| Length | 157 |
| Max. P | 0.993754 |

| Location | 13,980,585 – 13,980,705 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 73.29 |
| Mean single sequence MFE | -42.92 |
| Consensus MFE | -22.30 |
| Energy contribution | -24.47 |
| Covariance contribution | 2.17 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.52 |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.965377 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13980585 120 + 23771897 AAAAGGAGCCAACAAAGGUGGAACCCAAGCCCGGCGAGUCUCUGCUGCGCUCGACAGCGACUGUAACUGCAACACCUACUGCGGCAACAAUCGCUGCGACAACUCUGCUCGAUGUGAGCA .......((..(((..((..((......((.(((((......))))).))(((.((((((.(((..(((((........)))))..))).)))))))))....))..).)..)))..)). ( -39.00) >DroPse_CAF1 19401 106 + 1 AGGUGGUGCAGCCGAAAGUGGAGCCCAAGCCAGGGGAAUCUCUGCUGCGCAGCACAGCUACAGCUGCAGCCACAUCCACAGCCACAGCGGUAGCACCAGCAC-----CAGG--------- .(.((((((.(((....(((((((...(((.((((....)))))))))(((((.........))))).......))))).((....))))).)))))).)..-----....--------- ( -41.50) >DroSec_CAF1 17813 120 + 1 AAAAGGAGCCAACAAAGGUGGAACCCAAGCCGGGCGAGUCUCUGCUGCGCACGACAGCGACUGUAACUGCAACAACUACUGCAGCAGCAAUCGCUGCGACAACUCUGCUCGACGUGAGCA .......((..((...((......))..(.(((((((((...(((...)))((.((((((.(((..(((((........)))))..))).))))))))...)))).))))).)))..)). ( -37.80) >DroEre_CAF1 18326 120 + 1 AAAAGGAGCCCACGAAGGUGGAGCCCAAACCGGGCGAGUCUCUGCUACGCACUACAGCAACUGCAACUGCAUCAACAACUGCAGCAGCAGUCGCUGCGACAACUCUGCUCGAUGUGAGCA ....(((((((((....)))).((((.....))))..).))))(((.((((...((((.(((((..(((((........)))))..))))).))))(((((....)).))).))))))). ( -47.90) >DroYak_CAF1 18284 120 + 1 AAAAGGAGCCCACGAAGGUGGAGCCCAAGCCGGGCGAGUCUUUGCUGCGCACGACAGCAACUGUAACUGCAACACCAACUGCAGCAGCAGUCGCUGCGGCCACGCUGCUCGAUGUUAGCA ....((.((((((....)))).))))..((..(.(((((....((((.((.((.((((.(((((..(((((........)))))..))))).)))))))))).)).)))))...)..)). ( -50.00) >DroPer_CAF1 26081 106 + 1 AGGUGGUGCAGCCGAAAGUGGAGCCCAAGCCAGGAGAGUCUCUGCUGCGCAGCACAGCUACAGCUGCAGCCACAUCCACAGCCACAGCGGUAGCACCAGCAC-----CAGG--------- .(.((((((.(((....(((((......(((((.((.....)).))).)).((.((((....))))..))....))))).((....))))).)))))).)..-----....--------- ( -41.30) >consensus AAAAGGAGCCAACGAAGGUGGAGCCCAAGCCGGGCGAGUCUCUGCUGCGCACGACAGCAACUGCAACUGCAACAACAACUGCAGCAGCAGUCGCUGCGACAACUCUGCUCGAUGUGAGCA .....((((.......((..((((((......)).)...)))..))........((((((((((..(((((........)))))..))))))))))..........)))).......... (-22.30 = -24.47 + 2.17)

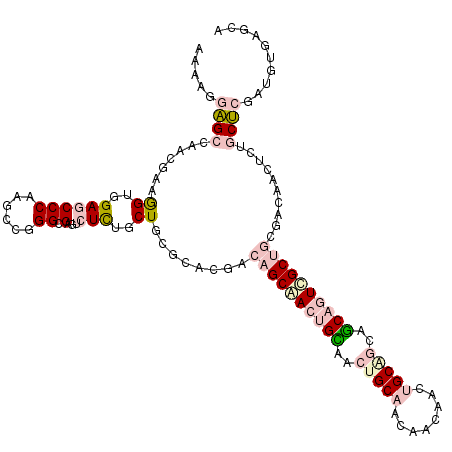
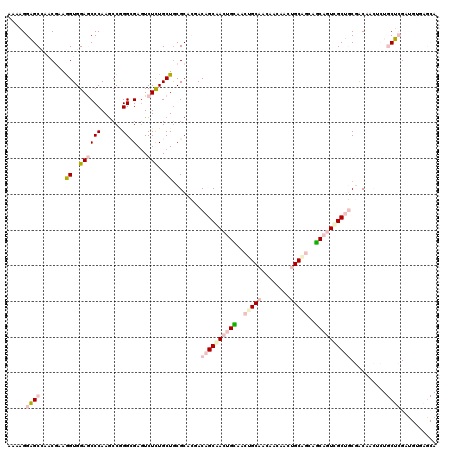
| Location | 13,980,585 – 13,980,705 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 73.29 |
| Mean single sequence MFE | -49.05 |
| Consensus MFE | -31.01 |
| Energy contribution | -29.85 |
| Covariance contribution | -1.16 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.63 |
| SVM decision value | 1.98 |
| SVM RNA-class probability | 0.984535 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13980585 120 - 23771897 UGCUCACAUCGAGCAGAGUUGUCGCAGCGAUUGUUGCCGCAGUAGGUGUUGCAGUUACAGUCGCUGUCGAGCGCAGCAGAGACUCGCCGGGCUUGGGUUCCACCUUUGUUGGCUCCUUUU (((((.....)))))(((((.(((((((((((((.((.((((......)))).)).)))))))))).)))((...))...)))))(((((.(..(((.....)))..))))))....... ( -46.90) >DroPse_CAF1 19401 106 - 1 ---------CCUG-----GUGCUGGUGCUACCGCUGUGGCUGUGGAUGUGGCUGCAGCUGUAGCUGUGCUGCGCAGCAGAGAUUCCCCUGGCUUGGGCUCCACUUUCGGCUGCACCACCU ---------..((-----((((.((((((((.((((..((..(....)..))..)))).))))).(((..((.((((..((......)).)).)).))..))).....)))))))))... ( -45.50) >DroSec_CAF1 17813 120 - 1 UGCUCACGUCGAGCAGAGUUGUCGCAGCGAUUGCUGCUGCAGUAGUUGUUGCAGUUACAGUCGCUGUCGUGCGCAGCAGAGACUCGCCCGGCUUGGGUUCCACCUUUGUUGGCUCCUUUU (((((.....)))))((((.(((((((((((((..(((((((......)))))))..))))))))).)).)).((((((((....((((.....)))).....))))))))))))..... ( -50.60) >DroEre_CAF1 18326 120 - 1 UGCUCACAUCGAGCAGAGUUGUCGCAGCGACUGCUGCUGCAGUUGUUGAUGCAGUUGCAGUUGCUGUAGUGCGUAGCAGAGACUCGCCCGGUUUGGGCUCCACCUUCGUGGGCUCCUUUU .(((((.((((.((.(((((.(((((((((((((.((((((........)))))).)))))))))))..(((...)))))))))))).)))).)))))(((((....)))))........ ( -54.60) >DroYak_CAF1 18284 120 - 1 UGCUAACAUCGAGCAGCGUGGCCGCAGCGACUGCUGCUGCAGUUGGUGUUGCAGUUACAGUUGCUGUCGUGCGCAGCAAAGACUCGCCCGGCUUGGGCUCCACCUUCGUGGGCUCCUUUU ..........((((.((.(((((((((((((((..(((((((......)))))))..))))))))).)).)).))))........((((.....)))).((((....))))))))..... ( -50.60) >DroPer_CAF1 26081 106 - 1 ---------CCUG-----GUGCUGGUGCUACCGCUGUGGCUGUGGAUGUGGCUGCAGCUGUAGCUGUGCUGCGCAGCAGAGACUCUCCUGGCUUGGGCUCCACUUUCGGCUGCACCACCU ---------..((-----((((.((((((((.((((..((..(....)..))..)))).))))).(((..((.((((.(((...)))...)).)).))..))).....)))))))))... ( -46.10) >consensus UGCUCACAUCGAGCAGAGUUGCCGCAGCGACUGCUGCUGCAGUAGAUGUUGCAGUUACAGUAGCUGUCGUGCGCAGCAGAGACUCGCCCGGCUUGGGCUCCACCUUCGUCGGCUCCUUUU ..........((((......((.(((((((((((.(((((((......))))))).)))))))))))...))(.(((.(((.((.....)))))..))).)..........))))..... (-31.01 = -29.85 + -1.16)

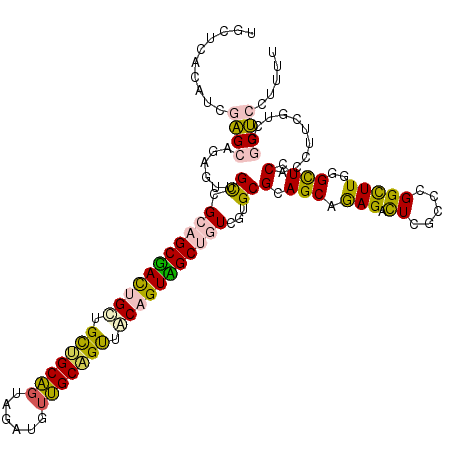
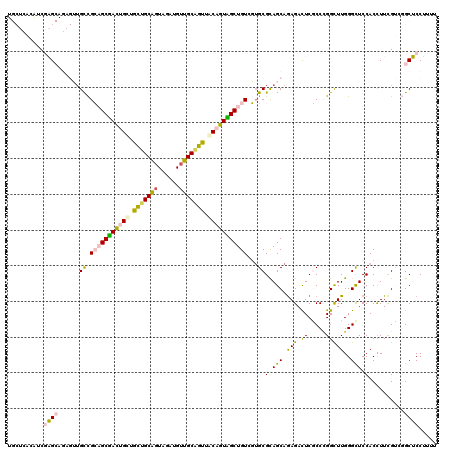
| Location | 13,980,625 – 13,980,742 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 69.04 |
| Mean single sequence MFE | -35.19 |
| Consensus MFE | -18.96 |
| Energy contribution | -22.27 |
| Covariance contribution | 3.31 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.54 |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.975471 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13980625 117 + 23771897 UCUGCUGCGCUCGACAGCGACUGUAACUGCAACACCUACUGCGGCAACAAUCGCUGCGACAACUCUGCUCGAUGUGAGCAAAGUGGCAUUCAAGACUCG---ACCACCUCUCAAACUGGA ..(((..((.(((.((((((.(((..(((((........)))))..))).)))))))))).....(((((.....)))))..)..)))...........---.(((..........))). ( -35.80) >DroPse_CAF1 19441 105 + 1 UCUGCUGCGCAGCACAGCUACAGCUGCAGCCACAUCCACAGCCACAGCGGUAGCACCAGCAC-----CAGG----------AGGUGCAAUCAAUUCCCGUCCGCUAACCCUCAAGAUGAA ..((((....))))..(((((.((((..((..........))..)))).)))))....((((-----(...----------.)))))..........((((.............)))).. ( -27.92) >DroSec_CAF1 17853 117 + 1 UCUGCUGCGCACGACAGCGACUGUAACUGCAACAACUACUGCAGCAGCAAUCGCUGCGACAACUCUGCUCGACGUGAGCAAAGUGGCAGUCAAGACUCG---ACCACCACUCAAGCUGGA ((((((((.((((.((((((.(((..(((((........)))))..))).))))))(((((....)).))).)))).))..(((((..(((.......)---))..)))))..))).))) ( -41.10) >DroEre_CAF1 18366 117 + 1 UCUGCUACGCACUACAGCAACUGCAACUGCAUCAACAACUGCAGCAGCAGUCGCUGCGACAACUCUGCUCGAUGUGAGCAAAGUGGCAGUCAAGACUAA---ACCCCCUCUUAAGCUGGA .(((((((......((((.(((((..(((((........)))))..))))).)))).........(((((.....)))))..)))))))..........---.((..((....))..)). ( -37.70) >DroYak_CAF1 18324 117 + 1 UUUGCUGCGCACGACAGCAACUGUAACUGCAACACCAACUGCAGCAGCAGUCGCUGCGGCCACGCUGCUCGAUGUUAGCAAAGCGGCAGUGAAGACUAA---ACCACCUGUUAAACUGGA ((..((((((.((.((((.(((((..(((((........)))))..))))).))))))))..(((((((.......)))..))))))))..))......---....((.(.....).)). ( -40.70) >DroPer_CAF1 26121 105 + 1 UCUGCUGCGCAGCACAGCUACAGCUGCAGCCACAUCCACAGCCACAGCGGUAGCACCAGCAC-----CAGG----------AGGUGCAAUCAAUUCCCGUCCGCUAACCCUCAAGAUGAA ..((((....))))..(((((.((((..((..........))..)))).)))))....((((-----(...----------.)))))..........((((.............)))).. ( -27.92) >consensus UCUGCUGCGCACGACAGCAACUGCAACUGCAACAACAACUGCAGCAGCAGUCGCUGCGACAACUCUGCUCGAUGUGAGCAAAGUGGCAGUCAAGACUCG___ACCACCUCUCAAGCUGGA ..((((((......((((((((((..(((((........)))))..)))))))))).........((((.......))))..))))))................................ (-18.96 = -22.27 + 3.31)

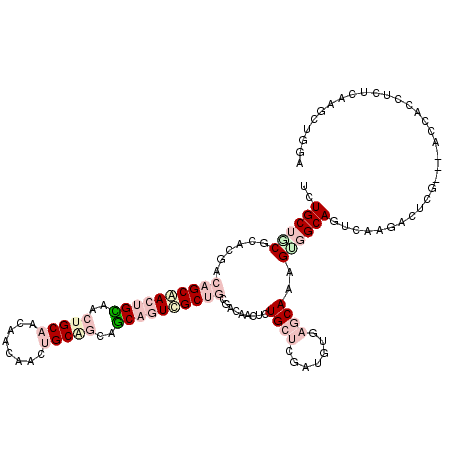
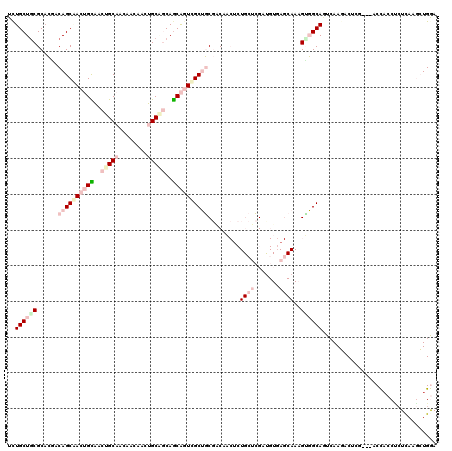
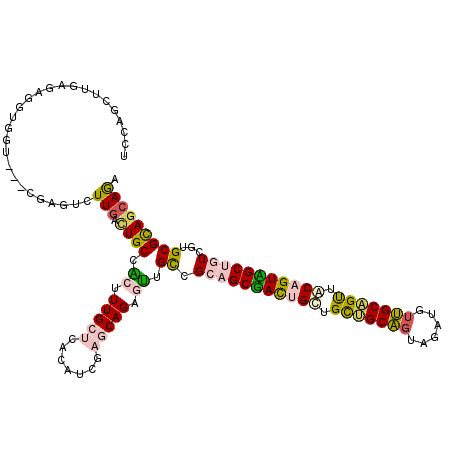
| Location | 13,980,625 – 13,980,742 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 69.04 |
| Mean single sequence MFE | -46.10 |
| Consensus MFE | -28.87 |
| Energy contribution | -28.73 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.63 |
| SVM decision value | 2.42 |
| SVM RNA-class probability | 0.993754 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13980625 117 - 23771897 UCCAGUUUGAGAGGUGGU---CGAGUCUUGAAUGCCACUUUGCUCACAUCGAGCAGAGUUGUCGCAGCGAUUGUUGCCGCAGUAGGUGUUGCAGUUACAGUCGCUGUCGAGCGCAGCAGA .....((((....(((.(---(((.........((.(((((((((.....))))))))).))..((((((((((.((.((((......)))).)).)))))))))))))).)))..)))) ( -45.20) >DroPse_CAF1 19441 105 - 1 UUCAUCUUGAGGGUUAGCGGACGGGAAUUGAUUGCACCU----------CCUG-----GUGCUGGUGCUACCGCUGUGGCUGUGGAUGUGGCUGCAGCUGUAGCUGUGCUGCGCAGCAGA ............(((.(((((((..........(((((.----------...)-----))))....(((((.((((..((..(....)..))..)))).)))))))).))))..)))... ( -40.30) >DroSec_CAF1 17853 117 - 1 UCCAGCUUGAGUGGUGGU---CGAGUCUUGACUGCCACUUUGCUCACGUCGAGCAGAGUUGUCGCAGCGAUUGCUGCUGCAGUAGUUGUUGCAGUUACAGUCGCUGUCGUGCGCAGCAGA ..((((((((((((..((---(((...)))))..))))))(((((.....))))))))))).(((((((((((..(((((((......)))))))..))))))))).))(((...))).. ( -54.40) >DroEre_CAF1 18366 117 - 1 UCCAGCUUAAGAGGGGGU---UUAGUCUUGACUGCCACUUUGCUCACAUCGAGCAGAGUUGUCGCAGCGACUGCUGCUGCAGUUGUUGAUGCAGUUGCAGUUGCUGUAGUGCGUAGCAGA ....(((...((((.(((---(.......)))).))(((((((((.....)))))))))..))(((((((((((.((((((........)))))).))))))))))).......)))... ( -51.20) >DroYak_CAF1 18324 117 - 1 UCCAGUUUAACAGGUGGU---UUAGUCUUCACUGCCGCUUUGCUAACAUCGAGCAGCGUGGCCGCAGCGACUGCUGCUGCAGUUGGUGUUGCAGUUACAGUUGCUGUCGUGCGCAGCAAA ....(((....(((((((---..(((....))))))))))....))).....((.(((((.(.((((((((((..(((((((......)))))))..)))))))))).)))))).))... ( -45.20) >DroPer_CAF1 26121 105 - 1 UUCAUCUUGAGGGUUAGCGGACGGGAAUUGAUUGCACCU----------CCUG-----GUGCUGGUGCUACCGCUGUGGCUGUGGAUGUGGCUGCAGCUGUAGCUGUGCUGCGCAGCAGA ............(((.(((((((..........(((((.----------...)-----))))....(((((.((((..((..(....)..))..)))).)))))))).))))..)))... ( -40.30) >consensus UCCAGCUUGAGAGGUGGU___CGAGUCUUGACUGCCACUUUGCUCACAUCGAGCAGAGUUGCCGCAGCGACUGCUGCUGCAGUAGAUGUUGCAGUUACAGUAGCUGUCGUGCGCAGCAGA ...........................(((.((((.((.(((((.......))))).)).((.(((((((((((.(((((((......))))))).)))))))))))...))))))))). (-28.87 = -28.73 + -0.13)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:41:57 2006