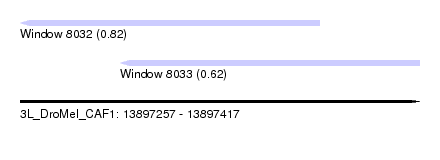
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,897,257 – 13,897,417 |
| Length | 160 |
| Max. P | 0.817899 |

| Location | 13,897,257 – 13,897,377 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.50 |
| Mean single sequence MFE | -45.78 |
| Consensus MFE | -26.05 |
| Energy contribution | -25.87 |
| Covariance contribution | -0.18 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.67 |
| SVM RNA-class probability | 0.817899 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13897257 120 - 23771897 UCAAACUGGGAUCCUGCAACGACUCCUUUUCGUUGCUAGAAUUGCGAUCCCUUAACUCAGUUAGUUGGUCGUUUAGCUUCUGUACCAGUUCCCUAAACUUAACGAUAGUUUGGUCGCGAU ...((((((......(((((((.......)))))))(((((..((((((..(((((...)))))..)))))).....)))))..))))))..(((((((.......)))))))....... ( -31.50) >DroVir_CAF1 13355 120 - 1 UAAGGCUGGGAUCCUGCAAUGACUUUUGGUCGCUGCUCGAGCUGCGCUCGCGCAGUUCAAUUAGCUGCUCGUUCAGCUUCUGGACGAGUUCGCGGAACUUGGUGAUGGUCUGGUCGCGAU ...((((((......(((.((((.....)))).)))..((((((((....))))))))..))))))((((((((((...))))))))))((((((.(((.......)))....)))))). ( -47.80) >DroGri_CAF1 6072 120 - 1 UGAGGCUGGGAUCCUGUAGAGAUUUCAGGUCAUUAUUGGAGCUGCGCUCGCGCAGCUCGAUUAGCUGUUCGUUCAGCUUUUGGACGAGCUCACGGAACUUCGUGAUGGUCUGGUCGCGAU .....(((..((((....).)))..)))(((.......((((((((....))))))))((((((((((((((((((...))))))))))((((((....)))))).)).))))))..))) ( -48.80) >DroMoj_CAF1 7069 120 - 1 UGAGGCUGGGAUCCUGCAGUGACUUUUGAUCAUUGUUAGAGCUGCGUUCGCGCAGUUCGAUCAGCUGUUCGUUCAGCUUCUGGACGAGCUCACGGAACUUGGUGAUGGUCUGGUCGCGAU ....((.((...)).)).((((((...((((((..(((((((((((....))))))))..((....((((((((((...)))))))))).....))...)))..)))))).))))))... ( -48.50) >DroAna_CAF1 5770 120 - 1 UGAGGCUCGGGUCCUGCAGCGACUCCUUGUCGUUGCUGGAGCUGCGCUCCCUCAGUUCGGUCAACUGGUCGUUCAGCUUCUGCACCAGUUCCCUGAACUUGGUGAUGGUCUGGUCGCGAU ((((((.(((.(((.((((((((.....)))))))).))).))).))...))))((((((..(((((((.((.........)))))))))..))))))...(((((......)))))... ( -50.70) >DroPer_CAF1 5922 120 - 1 UGAGGCUAGGGUCCUGCAGGGACUUGUCCUCAUUGCUGGACGUCCUCUCGCGCAGCUCCGUCAGCUGUUCGUUCAGCUUCUGGACCAGCUCCCGGAACUUGGUGAUGGUCUGAUCGCGAU .((((...(.((((.(((((((....))))...))).)))).)))))((((((((..((((((((((......))))((((((........)))))).....)))))).)))..))))). ( -47.40) >consensus UGAGGCUGGGAUCCUGCAGCGACUUCUGGUCAUUGCUGGAGCUGCGCUCGCGCAGCUCGAUCAGCUGUUCGUUCAGCUUCUGGACCAGCUCCCGGAACUUGGUGAUGGUCUGGUCGCGAU ...((((((......(((((((.......)))))))..((((((((....))))))))..))))))..((((((((((........))))....))))...(((((......))))))). (-26.05 = -25.87 + -0.18)


| Location | 13,897,297 – 13,897,417 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.94 |
| Mean single sequence MFE | -41.25 |
| Consensus MFE | -27.01 |
| Energy contribution | -24.85 |
| Covariance contribution | -2.16 |
| Combinations/Pair | 1.53 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.621336 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13897297 120 - 23771897 GCGAACAUUUGUUUGUAGUCGAUGGUUUCGGUGACCAUUUUCAAACUGGGAUCCUGCAACGACUCCUUUUCGUUGCUAGAAUUGCGAUCCCUUAACUCAGUUAGUUGGUCGUUUAGCUUC ((((((....(((((.....(((((((.....)))))))..))))).(((((((((((((((.......))))))).))......))))))((((((.....))))))..)))).))... ( -32.40) >DroVir_CAF1 13395 120 - 1 GCAAACAUUUGCUUGUAGUCGAUGGUCUCCGUGGCCAGUUUAAGGCUGGGAUCCUGCAAUGACUUUUGGUCGCUGCUCGAGCUGCGCUCGCGCAGUUCAAUUAGCUGCUCGUUCAGCUUC ((((....))))..(((((..((((((((...((((.......))))))))))..(((.((((.....)))).)))..((((((((....)))))))).))..)))))............ ( -40.60) >DroGri_CAF1 6112 120 - 1 GCGAACAUUUGCUUGUAGUCGAUCGUCUCGGUGGCCAGCUUGAGGCUGGGAUCCUGUAGAGAUUUCAGGUCAUUAUUGGAGCUGCGCUCGCGCAGCUCGAUUAGCUGUUCGUUCAGCUUU (((((((...(((...(((((((((((((...(((((((.....)))))...))....)))))....)))).......((((((((....))))))))))))))))))))))........ ( -45.70) >DroEre_CAF1 6081 120 - 1 GCGAACAUCUGCUUGUAGUCGAUGGUUUCGGUGACCAUUUUCAAGCUUGGGUCCUGCAGCGACUCCUUUUCGUUGCUGGAGCUGCGAUCCCUCAAAUCAGUUAGUUGAUCGUUCAGCUUC (((((((((.(((((.....(((((((.....)))))))..)))))..(((((((.(((((((........))))))).))....)))))................))).)))).))... ( -36.80) >DroMoj_CAF1 7109 120 - 1 GCGAACAUCUGCUUGUAGUCGAUGGUCUCGGUGGCCAGCUUGAGGCUGGGAUCCUGCAGUGACUUUUGAUCAUUGUUAGAGCUGCGUUCGCGCAGUUCGAUCAGCUGUUCGUUCAGCUUC .(((.((((.((.....)).))))...)))....(((((.....)))))((((..((((((((....).)))))))..((((((((....))))))))))))(((((......))))).. ( -45.20) >DroAna_CAF1 5810 120 - 1 GCAAACAUCUGCUUGUAGUCGAUGGUCUCUGUGGCCAGCUUGAGGCUCGGGUCCUGCAGCGACUCCUUGUCGUUGCUGGAGCUGCGCUCCCUCAGUUCGGUCAACUGGUCGUUCAGCUUC (((......)))....(((.((((..(....(((((((((.(((((.(((.(((.((((((((.....)))))))).))).))).))...))))))).)))))...)..))))..))).. ( -46.80) >consensus GCGAACAUCUGCUUGUAGUCGAUGGUCUCGGUGGCCAGCUUGAGGCUGGGAUCCUGCAGCGACUCCUGGUCGUUGCUGGAGCUGCGCUCCCGCAGUUCAAUUAGCUGGUCGUUCAGCUUC ((((((.......((((((...(((((.....)))))..............((..(((((((.......)))))))..)))))))).....((((((.....))))))..)))).))... (-27.01 = -24.85 + -2.16)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:41:13 2006