
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,418,360 – 13,418,520 |
| Length | 160 |
| Max. P | 0.822390 |

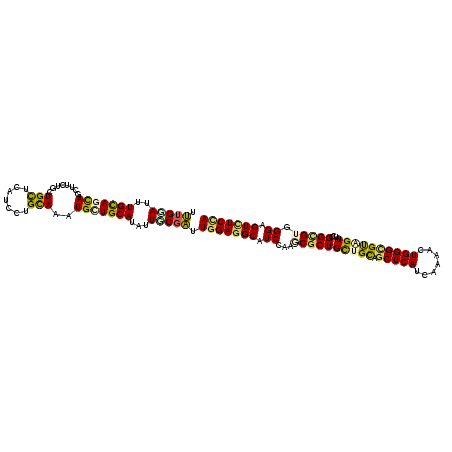
| Location | 13,418,360 – 13,418,480 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.72 |
| Mean single sequence MFE | -43.70 |
| Consensus MFE | -27.33 |
| Energy contribution | -26.67 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.822390 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13418360 120 - 23771897 UUUGGAUUUGUAACAGCUUCGCCUGUUCGUCGUGCAGGUGCUGCAUGUUCCUAUUGGAGGCUUCCAAGCGCUGCUGCAACUCAUCGAAGCUGGGCGUGGUAUCGGGCGUGGGCGUUUCCA ..((((..(((..(((((((((((((.......))))))((.(((..((((....))))(((....)))..))).))........))))))).((.((....)).))....)))..)))) ( -40.90) >DroVir_CAF1 17042 120 - 1 UUUGUAUUUGCAGCAACUUUUGCUGCUCAUCCUGCAAAUGCUGCAUAUUGCGAUUUGAGGCAUCCAAGCGCUGCUGCAGCUCAUCAAAACUGGGUGUAGUAUCCGGCGUUGGUGUCUCCA .(((((..(((((((...(((((..........))))))))))))...)))))...((((((.((((.((((((((((.((((.......))))))))))....)))))))))))))).. ( -44.50) >DroGri_CAF1 16327 120 - 1 UUUGGAUUUGCAGCAACUUUUGCUGCUCAUCAUGUAAAUGCUGCAUGUUUCGAUUGGAGGCAUCCAAGCGCUGCUGCAGCUCAUCGAAACUGGGCGUAGUAUCCGGCGUGGGCGUCUCCA .(((((..(((((((...(((((..........))))))))))))...))))).(((((((..((..((((((((((.(((((.......))))))))))....)))))))..))))))) ( -45.50) >DroWil_CAF1 17983 120 - 1 UCUGUAUUUGUAGCAGCUUCUGUUGCUCAUCCUGCAAAUGUUGCAUAUUCCGGUUGGAGGCCUCCAAACGCUGUUGCAGCUCAUCGAAAGUGGGCGCUGUAUCUGGUGUGGGAGUAUCUA ..((((..((.((((((....))))))))...))))........(((((((..(((((....)))))(((((..((((((.(..(....)...).))))))...))))).)))))))... ( -38.60) >DroMoj_CAF1 17282 120 - 1 UUUGUAUUUGUAGCAGCUUCUGCUGCUCAUCCUGCAAGUGUUGCAUAUUGCGAUUGGAGGCAUCUAGCCGCUGUUGUAGCUCAUCAAAACUGGGCGUUGUAUCUGGUGUUGGUGUCUCCA ((((((..((.((((((....))))))))...)))))).(((((.....)))))((((((((((..((((........(((((.......)))))........))))...)))))))))) ( -43.39) >DroAna_CAF1 19234 120 - 1 UCUGGAUUUGCAAUAGUUUGGCCUGCUCAUCUUGCAGAUGCUGCAUGUUGCGGUUGGAGGCCUCCAGCCUCUGCUGCAGCUCAUCAAAGGUGGGUGCAGUGUCGGGCGUAGGAGUUUCCA ...(((....(....((((((((((((((((((...((.((((((.((.(.(((((((....))))))).).))))))))))....)))))))..)))).)))))))....)....))). ( -49.30) >consensus UUUGGAUUUGCAGCAGCUUCUGCUGCUCAUCCUGCAAAUGCUGCAUAUUGCGAUUGGAGGCAUCCAAGCGCUGCUGCAGCUCAUCAAAACUGGGCGUAGUAUCCGGCGUGGGAGUCUCCA .(((((..(((((((........(((.......)))..)))))))...))))).(((((((.((...((((((((((.(((((.......))))))))))....))))).)).))))))) (-27.33 = -26.67 + -0.66)

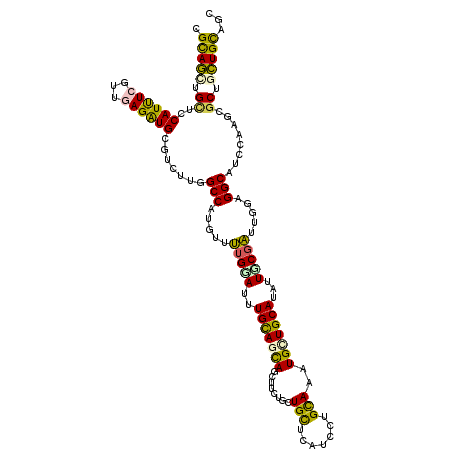
| Location | 13,418,400 – 13,418,520 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.33 |
| Mean single sequence MFE | -39.38 |
| Consensus MFE | -26.20 |
| Energy contribution | -25.07 |
| Covariance contribution | -1.13 |
| Combinations/Pair | 1.48 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.812303 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13418400 120 - 23771897 CGCAGCCGCUCCAUCUCGUUAAGGUGAGUCUUGGCCAAAUUUUGGAUUUGUAACAGCUUCGCCUGUUCGUCGUGCAGGUGCUGCAUGUUCCUAUUGGAGGCUUCCAAGCGCUGCUGCAAC .(((((.((.(((.((((......))))...)))......((((((...((....((..(((((((.......)))))))..))...((((....)))))).)))))).)).)))))... ( -40.70) >DroVir_CAF1 17082 120 - 1 CGCAGAUGUUCCAUUUCGUUGAGAUGUGUCUUAGCCAUGUUUUGUAUUUGCAGCAACUUUUGCUGCUCAUCCUGCAAAUGCUGCAUAUUGCGAUUUGAGGCAUCCAAGCGCUGCUGCAGC .((.(((((..((..(((((((((....)))))))........(((..(((((((...(((((..........))))))))))))...)))))..))..)))))...))((((...)))) ( -38.00) >DroGri_CAF1 16367 120 - 1 CGUAGUUGCUCCAUUUCGUUGAGAUGUGUUUUGGCCAUGUUUUGGAUUUGCAGCAACUUUUGCUGCUCAUCAUGUAAAUGCUGCAUGUUUCGAUUGGAGGCAUCCAAGCGCUGCUGCAGC .(((((.((((((....((((((((((((...(((.((......(((..((((((.....))))))..)))......))))))))))))))))))))).((......)))).)))))... ( -39.20) >DroWil_CAF1 18023 120 - 1 CGCAAAUGUUCCAUUUCAUUUAGAUGCGAUUUAGCCAUAUUCUGUAUUUGUAGCAGCUUCUGUUGCUCAUCCUGCAAAUGUUGCAUAUUCCGGUUGGAGGCCUCCAAACGCUGUUGCAGC .(((((((........)))))...((((((...((.(((((.((((..((.((((((....))))))))...))))))))).))......((.(((((....))))).))..)))))))) ( -30.20) >DroMoj_CAF1 17322 120 - 1 CGCAACUGCUCCAUUUCGUUGAGAUGCGUUUUGGCCAUGUUUUGUAUUUGUAGCAGCUUCUGCUGCUCAUCCUGCAAGUGUUGCAUAUUGCGAUUGGAGGCAUCUAGCCGCUGUUGUAGC .(((((.((((((..((((....((((((....)).....((((((..((.((((((....))))))))...))))))....))))...)))).))))(((.....))))).)))))... ( -40.00) >DroAna_CAF1 19274 120 - 1 CGCAGCUGUUCCAUCUCGCUGAGAUGCGACUUGGCCAGGUUCUGGAUUUGCAAUAGUUUGGCCUGCUCAUCUUGCAGAUGCUGCAUGUUGCGGUUGGAGGCCUCCAGCCUCUGCUGCAGC .(((((......((((.((.((((((.(....(((((..((.((......))..))..)))))..).)))))))))))))))))..((((((((.((((((.....)))))))))))))) ( -48.20) >consensus CGCAGCUGCUCCAUUUCGUUGAGAUGCGUCUUGGCCAUGUUUUGGAUUUGCAGCAGCUUCUGCUGCUCAUCCUGCAAAUGCUGCAUAUUGCGAUUGGAGGCAUCCAAGCGCUGCUGCAGC .(((((.((..((((((...)))))).......(((.....(((((..(((((((........(((.......)))..)))))))...))))).....)))........)).)))))... (-26.20 = -25.07 + -1.13)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:33:16 2006