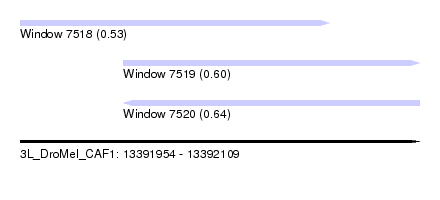
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,391,954 – 13,392,109 |
| Length | 155 |
| Max. P | 0.636139 |

| Location | 13,391,954 – 13,392,074 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.72 |
| Mean single sequence MFE | -54.13 |
| Consensus MFE | -33.83 |
| Energy contribution | -35.70 |
| Covariance contribution | 1.87 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.63 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.530401 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13391954 120 + 23771897 CAUAGAGCUUCGAGUUGGACGGCGGCUUGAUGGGAGCGGUGGCCAGCGACUUGUCGCGGUCGGCGCUCAGCGAGAGCAGAUUGGGCUCCGACUUGGCGCUGACCAGCGCCAUUCCGCCGG ...................((((((((..((..(((((.(((((.((((....))))))))).))))).((....))..))..)))...((..(((((((....))))))).))))))). ( -58.30) >DroPse_CAF1 9456 120 + 1 CAUAGAGCUUUGAGUUGGACGGCGGCUUGAUGGGGGUAGAGGCCAGCUGCUUCUCGCGGUCGGCGCUUAGGGAGAGCAGAUUUGGCUCCGAUUUGGCGCUGACCAGAGCCAUGCCUCCGG .........(..(((((.....)))))..)((((((((..((((.((........))((((((((((.((...((((.......))))...)).)))))))))).).))).)))))))). ( -49.40) >DroSec_CAF1 7774 120 + 1 CAUAAAGCUUGGAGUUGGACGGCGGCUUGAUGGGUGCGGUGGCCAGCGAUUUGUCGCGGUCGGCGCUCAGCGAGAGCAGAUUGGGCUCCGACUUGGCGCUGACCAGCGCCAUUCCGCCGG (((.(((((....(((....)))))))).)))((((..((((((.((((....))))((((((((((.((((.((((.......)))))).)).)))))))))).).)))))..)))).. ( -56.70) >DroMoj_CAF1 4731 101 + 1 CAUAGAGCUUGGAGUUGGAUGGCGGCUUGCUGGGCGU------CACCAUCUUC------UCGGCGCUCAGCGACAGCAGAUUCGGCUCCGACUUGGUGCUGAUCAAUU------GGCCG- ....(((.((((((((((((.((...(((((((((((------(.........------..))))))))))))..))..))))))))))))))).(.((..(....).------.))).- ( -42.40) >DroAna_CAF1 13864 120 + 1 CAUAGAGCUUCGAGUUGGAGGGCGGCUUGAUGGCGGCGGGCGGUAGCGGCUUGUCGCGGUCGGCGCUCAGUGAGAGCAGGUUCGGCUCUGACUUGGCGCUGACCAACGCCAUUCCGCCAG .........((((((((.....))))))))((((((..((((...((((....))))((((((((((.(((.(((((.......))))).))).))))))))))..))))...)))))). ( -66.20) >DroPer_CAF1 9597 120 + 1 CAUAGAGCUUGGAGUUGGACGGCGGCUUGAUGGGGGUGGAGGCCAGCUGCUUCUCGCGGUCGGCGCUUAGGGAGAGCAGAUUUGGCUCCGAUUUGGCGCUGACCAGAGCCAUGCCUCCGG ....((((.(((..((((.(((((.(..(((.(((((.((((((.(((((.....))))).)))((((.....))))...))).))))).))).).))))).))))..))).).)))... ( -51.80) >consensus CAUAGAGCUUGGAGUUGGACGGCGGCUUGAUGGGGGCGGAGGCCAGCGGCUUCUCGCGGUCGGCGCUCAGCGAGAGCAGAUUCGGCUCCGACUUGGCGCUGACCAGCGCCAUUCCGCCGG (((.(((((....(((....)))))))).)))....(((((((..((........))((((((((((.((((.((((.......)))))).)).))))))))))........))))))). (-33.83 = -35.70 + 1.87)

| Location | 13,391,994 – 13,392,109 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.86 |
| Mean single sequence MFE | -57.47 |
| Consensus MFE | -44.35 |
| Energy contribution | -46.10 |
| Covariance contribution | 1.75 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.598017 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
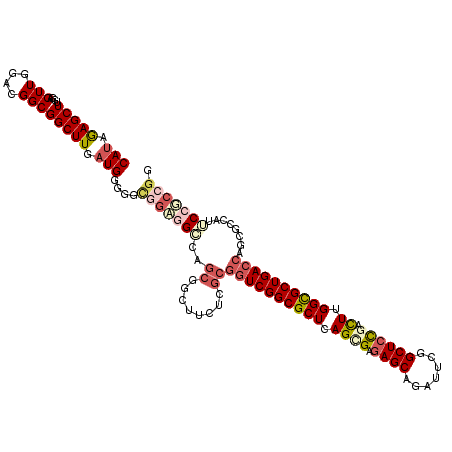
>3L_DroMel_CAF1 13391994 115 + 23771897 GGCCAGCGACUUGUCGCGGUCGGCGCUCAGCGAGAGCAGAUUGGGCUCCGACUUGGCGCUGACCAGCGCCAUUCCGCCGGCGGCCGCUGCCGCUGCU---GCUGCCGCCUCCCGCUGA-- ...(((((.......((((((((.(((((((....))....))))).))))).(((((((....)))))))...))).((((((.((.((....)).---)).))))))...))))).-- ( -61.70) >DroSec_CAF1 7814 115 + 1 GGCCAGCGAUUUGUCGCGGUCGGCGCUCAGCGAGAGCAGAUUGGGCUCCGACUUGGCGCUGACCAGCGCCAUUCCGCCGGCGGCCGCUGCCGCUGCU---GCUGCCGCUUCCCGCUGA-- ...(((((.......((((((((.(((((((....))....))))).))))).(((((((....)))))))...)))(((((((.((.......)).---))))))).....))))).-- ( -59.00) >DroSim_CAF1 4733 115 + 1 GGCCAGCGACUUGUCGCGGUCGGCGCUCAGCGAGAGCAGAUUGGGCUCCGACUUGGCGCUGACCAGCGCCAUUCCGCCGGCGGCCGCUGCCGCUGCU---GCUGCCGCUUCCCGCUGA-- ...(((((.......((((((((.(((((((....))....))))).))))).(((((((....)))))))...)))(((((((.((.......)).---))))))).....))))).-- ( -59.00) >DroEre_CAF1 5115 115 + 1 GGCCAGCGACUUGUCGCGGUCGGCGCUCAGCGAGAGCAGAUUGGGCUCCGACUUGGCGCUGACCAGCGCCAUUCCGCCGGCGGCCGCUGCCGCUGCU---GCUGCCGCUUCCCGCUGA-- ...(((((.......((((((((.(((((((....))....))))).))))).(((((((....)))))))...)))(((((((.((.......)).---))))))).....))))).-- ( -59.00) >DroMoj_CAF1 4768 96 + 1 ---CACCAUCUUC------UCGGCGCUCAGCGACAGCAGAUUCGGCUCCGACUUGGUGCUGAUCAAUU------GGCCG------GCCGCAGCAGCA---GCCGCCGCCUCGCGCUGGCA ---..((......------..)).((.(((((...((.(((.((((.((.....)).)))))))....------(((.(------((.((....)).---))))))))....))))))). ( -36.80) >DroAna_CAF1 13904 118 + 1 CGGUAGCGGCUUGUCGCGGUCGGCGCUCAGUGAGAGCAGGUUCGGCUCUGACUUGGCGCUGACCAACGCCAUUCCGCCAGCGGCCGCCGCUGCUGCCGCGGCAGCCGCCUCCCGCUGG-- .((..((((((.(((((((((((((((.(((.(((((.......))))).))).))))))))))...........(.(((((((....))))))).))))))))))))..))......-- ( -69.30) >consensus GGCCAGCGACUUGUCGCGGUCGGCGCUCAGCGAGAGCAGAUUGGGCUCCGACUUGGCGCUGACCAGCGCCAUUCCGCCGGCGGCCGCUGCCGCUGCU___GCUGCCGCCUCCCGCUGA__ (((..((((....))))((((((((((.((((.((((.......)))))).)).))))))))))...))).......((((((..((.((.((.......)).)).))...))))))... (-44.35 = -46.10 + 1.75)
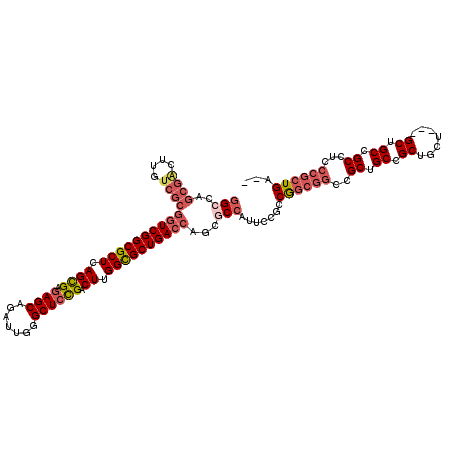
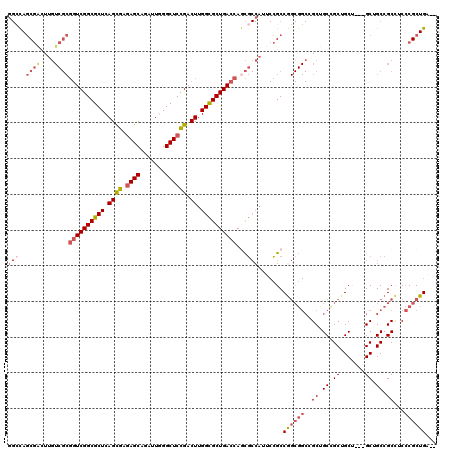
| Location | 13,391,994 – 13,392,109 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.86 |
| Mean single sequence MFE | -58.07 |
| Consensus MFE | -44.07 |
| Energy contribution | -45.88 |
| Covariance contribution | 1.81 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.21 |
| SVM RNA-class probability | 0.636139 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13391994 115 - 23771897 --UCAGCGGGAGGCGGCAGC---AGCAGCGGCAGCGGCCGCCGGCGGAAUGGCGCUGGUCAGCGCCAAGUCGGAGCCCAAUCUGCUCUCGCUGAGCGCCGACCGCGACAAGUCGCUGGCC --(((((((((((((((.((---.((....)).)).)))))).(((((.(((((((....)))))))....((...))..))))))))))))))..((((...((((....)))))))). ( -61.90) >DroSec_CAF1 7814 115 - 1 --UCAGCGGGAAGCGGCAGC---AGCAGCGGCAGCGGCCGCCGGCGGAAUGGCGCUGGUCAGCGCCAAGUCGGAGCCCAAUCUGCUCUCGCUGAGCGCCGACCGCGACAAAUCGCUGGCC --...((.....))(((.((---.((....)).)).)))((((((((..((.(((.((((.((((..(((.(((((.......))))).)))..)))).))))))).))..)))))))). ( -57.20) >DroSim_CAF1 4733 115 - 1 --UCAGCGGGAAGCGGCAGC---AGCAGCGGCAGCGGCCGCCGGCGGAAUGGCGCUGGUCAGCGCCAAGUCGGAGCCCAAUCUGCUCUCGCUGAGCGCCGACCGCGACAAGUCGCUGGCC --...((.....))(((.((---.((....)).)).)))((((((((..((.(((.((((.((((..(((.(((((.......))))).)))..)))).))))))).))..)))))))). ( -58.50) >DroEre_CAF1 5115 115 - 1 --UCAGCGGGAAGCGGCAGC---AGCAGCGGCAGCGGCCGCCGGCGGAAUGGCGCUGGUCAGCGCCAAGUCGGAGCCCAAUCUGCUCUCGCUGAGCGCCGACCGCGACAAGUCGCUGGCC --...((.....))(((.((---.((....)).)).)))((((((((..((.(((.((((.((((..(((.(((((.......))))).)))..)))).))))))).))..)))))))). ( -58.50) >DroMoj_CAF1 4768 96 - 1 UGCCAGCGCGAGGCGGCGGC---UGCUGCUGCGGC------CGGCC------AAUUGAUCAGCACCAAGUCGGAGCCGAAUCUGCUGUCGCUGAGCGCCGA------GAAGAUGGUG--- .((((((((..(((((((((---(((....)))))------).)))------....((.(((((.....(((....)))...))))))))))..))))(..------...).)))).--- ( -43.40) >DroAna_CAF1 13904 118 - 1 --CCAGCGGGAGGCGGCUGCCGCGGCAGCAGCGGCGGCCGCUGGCGGAAUGGCGUUGGUCAGCGCCAAGUCAGAGCCGAACCUGCUCUCACUGAGCGCCGACCGCGACAAGCCGCUACCG --....(((..((((((((((((.......))))))))))))(((((..((.(((.((((.((((..(((.(((((.......))))).)))..)))).))))))).))..))))).))) ( -68.90) >consensus __UCAGCGGGAAGCGGCAGC___AGCAGCGGCAGCGGCCGCCGGCGGAAUGGCGCUGGUCAGCGCCAAGUCGGAGCCCAAUCUGCUCUCGCUGAGCGCCGACCGCGACAAGUCGCUGGCC ........((..((.((.((.......)).)).))..))((((((((..((.(((.((((.((((..(((.(((((.......))))).)))..)))).))))))).))..)))))))). (-44.07 = -45.88 + 1.81)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:32:57 2006