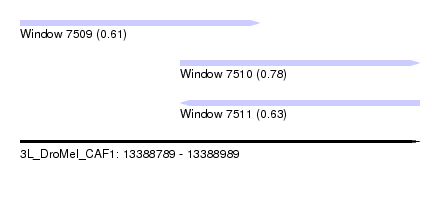
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,388,789 – 13,388,989 |
| Length | 200 |
| Max. P | 0.776521 |

| Location | 13,388,789 – 13,388,909 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.83 |
| Mean single sequence MFE | -49.15 |
| Consensus MFE | -32.77 |
| Energy contribution | -32.25 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.606377 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13388789 120 + 23771897 UGGGCGAAUUUGGAUCUCCUGCCCACAAGGGCGGCGUUUACGCAUUGGCCUGGAAACCAGACAGCACCCAACUGCUAACCUGCUCCGGCGACAAGACGUGCCGCCUUUGGACAGUGGAGU ((((((.....((....))))))))((((((((((....(((..(((.((((((...(((..((((......))))...))).))))).).)))..)))))))))))))........... ( -43.50) >DroVir_CAF1 1996 120 + 1 UGGGCGAAUUUGGUUCGCCAGCACACAAGGGUGGCGUUUAUGCGGUCGCAUGGAAGCCAGACGGCACACAGCUACUUACCUGCUCGGGUGAUAAGACCUGCCGUCUCUGGCAGGUUGAGU ..(((((((...)))))))....(((..(((((((.(((((((....))))))).(((....))).....))))))).((.....)))))....(((((((((....))))))))).... ( -47.40) >DroGri_CAF1 1851 120 + 1 UGGGUGAAUUUGGUUCACCGGCGCACAAGGGUGGCGUCUAUGCGGUCGCCUGGAAGCCAGAUGGCACUCAGCUGUUGACCUGCUCCGGUGACAAGACCUGCCGCCUGUGGCAGGUUGAGU ..(((((((...)))))))(((((((....)).)))))......((((((.(((.((..((((((.....)))))).....)))))))))))..(((((((((....))))))))).... ( -50.90) >DroWil_CAF1 2755 120 + 1 UUGGUGAAUUUGGUUCACCAGCCCAUAAGGGUGGCGUCUAUGCUGUGGCAUGGAAGCCAGAUGGCACACAAUUGUUAACCUGCUCGGGCGAUAAAACUUGCCGCUUGUGGCAAGUGGAGA .((((((((...))))))))((((....(((((((.(((((((....))))))).)))...(((((......)))))))))....))))......((((((((....))))))))..... ( -51.10) >DroMoj_CAF1 1846 120 + 1 UGGGCGAAUUCGGCUCGCCAGCGCACAAGGGUGGCGUCUAUGCGGUCGCAUGGAAGCCGGACAGCACUCAGCUGCUCACCUGCUCGGGCGACAAGACCUGCCGUCUCUGGCAGGUUGAGU ..(((((.......))))).((.(....(((((((.(((((((....))))))).)))(..((((.....))))..)))))....).)).((..(((((((((....)))))))))..)) ( -54.10) >DroAna_CAF1 9831 120 + 1 UGGGCGAAUUUGGGUCGCCAGCUCAUAAAGGCGGCGUCUAUGCCUUGGCCUGGAAGCCAGACAGUACCCAGCUGCUGACCUGCUCCGGCGAUAAGACCUGUCGCUUGUGGCAGGUGGAAU ..(((...((..(((((((..........)))((((....))))..))))..)).)))...(((((......)))))((((((..((((((((.....)))))).))..))))))..... ( -47.90) >consensus UGGGCGAAUUUGGUUCGCCAGCCCACAAGGGUGGCGUCUAUGCGGUCGCAUGGAAGCCAGACAGCACACAGCUGCUAACCUGCUCCGGCGACAAGACCUGCCGCCUGUGGCAGGUGGAGU ..(((((.......))))).((......(((((((.((((.((....)).)))).)))....((((......)))).))))......))......((((((((....))))))))..... (-32.77 = -32.25 + -0.52)


| Location | 13,388,869 – 13,388,989 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.67 |
| Mean single sequence MFE | -50.28 |
| Consensus MFE | -28.02 |
| Energy contribution | -27.25 |
| Covariance contribution | -0.77 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.33 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.776521 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13388869 120 + 23771897 UGCUCCGGCGACAAGACGUGCCGCCUUUGGACAGUGGAGUCGCGCGAGCUGGUUAGCGAGUUUGUGAUGGGCACCACUGUGGACGACCAGCAGGUGUCCUGCUUGUGGCAAGGAGACAAC (((((((((.((.....)))))(((...(((((((((.(((.((((((((.(....).))))))))...))).)))))))((((.(((....)))))))..))...)))..)))).)).. ( -53.20) >DroVir_CAF1 2076 120 + 1 UGCUCGGGUGAUAAGACCUGCCGUCUCUGGCAGGUUGAGUCUCGUGAGCUGAUCAGCGAGUUUGUUAUGGGCAGCACCGUGGAUGAUCAGCAGGUGUCGUGUCUGUGGCAAGGGGAGCAU .(((((.(.(((..(((((((((....)))))))))..))).).)))))......((...((((((((((((((((((((.(.....).)).)))))..)))))))))))))....)).. ( -50.40) >DroGri_CAF1 1931 120 + 1 UGCUCCGGUGACAAGACCUGCCGCCUGUGGCAGGUUGAGUCCCGUGAGCUGAUCAGUGAAUUUGUAAUGGGCACCACUGUGGAUGAUCAGCAGGUGUCGUGCCUGUGGCAGGGAGAGCAU .(((((((.(((..(((((((((....)))))))))..)))))).))))......((...(((((.((((((((((((((.(.....).))).)))..)))))))).)))))....)).. ( -52.90) >DroWil_CAF1 2835 120 + 1 UGCUCGGGCGAUAAAACUUGCCGCUUGUGGCAAGUGGAGACACGUGAAUUGAUCUCGGAAUUUGUUAUGGGAAACACCGUUGACGAUCAGCAGGUGUCGUGUUUGUGGCAAGGAGAACAC ..(((..((.(((((((((((((....))))))))...(((((.((...(((((((((....((((......))))...)))).))))).)).)))))...))))).))...)))..... ( -40.40) >DroMoj_CAF1 1926 120 + 1 UGCUCGGGCGACAAGACCUGCCGUCUCUGGCAGGUUGAGUCGCGCGAACUGAUCAGCGAGUUUGUCAUGGGCAAUACCGUGGAUGAUCAGCAGGUGUCGUGCCUGUGGCAGGGGGAGCAC ((((((.(((((..(((((((((....)))))))))..))))).)...............((((((((((((((((((((.(.....).)).)))))..)))))))))))))..))))). ( -56.40) >DroAna_CAF1 9911 120 + 1 UGCUCCGGCGAUAAGACCUGUCGCUUGUGGCAGGUGGAAUCGCGUGAACUUGUUGCAGAGUUCGUAAUGGGAGCCACCGUGGACGAUCAGCAAGUGUCCUGCCUGUGGCAGGGAGACAAC ((((((.(((((...((((((((....))))))))...)))))....((((((((......((((.((((......))))..)))).))))))))...(((((...))))))))).)).. ( -48.40) >consensus UGCUCCGGCGACAAGACCUGCCGCCUGUGGCAGGUGGAGUCGCGUGAACUGAUCAGCGAGUUUGUAAUGGGCACCACCGUGGACGAUCAGCAGGUGUCGUGCCUGUGGCAAGGAGAGCAC ..(((....(((...((((((((....))))))))...)))...................(((((.(..((((.((((((.(.....).)).))))...))))..).))))))))..... (-28.02 = -27.25 + -0.77)



| Location | 13,388,869 – 13,388,989 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.67 |
| Mean single sequence MFE | -37.74 |
| Consensus MFE | -22.40 |
| Energy contribution | -23.82 |
| Covariance contribution | 1.42 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.626380 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13388869 120 - 23771897 GUUGUCUCCUUGCCACAAGCAGGACACCUGCUGGUCGUCCACAGUGGUGCCCAUCACAAACUCGCUAACCAGCUCGCGCGACUCCACUGUCCAAAGGCGGCACGUCUUGUCGCCGGAGCA ..((.((((.(((.....)))(((((..((..((((((.(...(((((....)))))......(((....)))..).)))))).)).)))))...(((((((.....))))))))))))) ( -40.10) >DroVir_CAF1 2076 120 - 1 AUGCUCCCCUUGCCACAGACACGACACCUGCUGAUCAUCCACGGUGCUGCCCAUAACAAACUCGCUGAUCAGCUCACGAGACUCAACCUGCCAGAGACGGCAGGUCUUAUCACCCGAGCA .(((((.......................((((((((....((((..((.......)).)).)).))))))))......((....(((((((......)))))))....))....))))) ( -31.50) >DroGri_CAF1 1931 120 - 1 AUGCUCUCCCUGCCACAGGCACGACACCUGCUGAUCAUCCACAGUGGUGCCCAUUACAAAUUCACUGAUCAGCUCACGGGACUCAACCUGCCACAGGCGGCAGGUCUUGUCACCGGAGCA .(((((..(.((((...)))).)......((((((((......(((((....)))))........))))))))...((((((...(((((((......)))))))...))).)))))))) ( -44.44) >DroWil_CAF1 2835 120 - 1 GUGUUCUCCUUGCCACAAACACGACACCUGCUGAUCGUCAACGGUGUUUCCCAUAACAAAUUCCGAGAUCAAUUCACGUGUCUCCACUUGCCACAAGCGGCAAGUUUUAUCGCCCGAGCA .(((((................(((((.((.(((((.....(((.((((........)))).))).)))))...)).)))))...(((((((......)))))))..........))))) ( -29.60) >DroMoj_CAF1 1926 120 - 1 GUGCUCCCCCUGCCACAGGCACGACACCUGCUGAUCAUCCACGGUAUUGCCCAUGACAAACUCGCUGAUCAGUUCGCGCGACUCAACCUGCCAGAGACGGCAGGUCUUGUCGCCCGAGCA .(((((....((((...))))(((.....((((((((....((((.(((.......))))).)).))))))))))).(((((...(((((((......)))))))...)))))..))))) ( -41.80) >DroAna_CAF1 9911 120 - 1 GUUGUCUCCCUGCCACAGGCAGGACACUUGCUGAUCGUCCACGGUGGCUCCCAUUACGAACUCUGCAACAAGUUCACGCGAUUCCACCUGCCACAAGCGACAGGUCUUAUCGCCGGAGCA ........((((((...)))))).((((.(.((......)))))))(((((......(((((........)))))..(((((...(((((.(......).)))))...))))).))))). ( -39.00) >consensus GUGCUCUCCCUGCCACAGGCACGACACCUGCUGAUCAUCCACGGUGGUGCCCAUAACAAACUCGCUGAUCAGCUCACGCGACUCAACCUGCCACAGGCGGCAGGUCUUAUCGCCCGAGCA .(((((.......................((((((((.....(((..............)))...))))))))....(((((...(((((((......)))))))...)))))..))))) (-22.40 = -23.82 + 1.42)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:32:47 2006