
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,297,483 – 13,297,627 |
| Length | 144 |
| Max. P | 0.556829 |

| Location | 13,297,483 – 13,297,592 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.31 |
| Mean single sequence MFE | -30.55 |
| Consensus MFE | -19.02 |
| Energy contribution | -19.55 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.62 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.513826 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
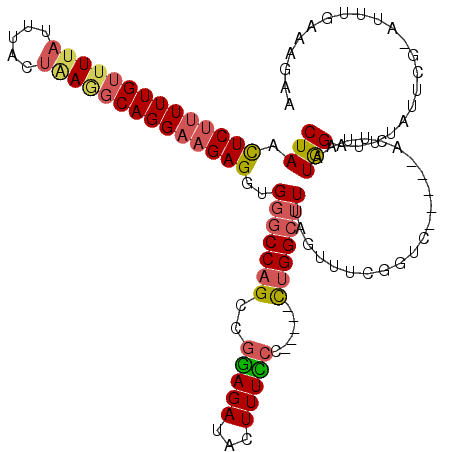
>3L_DroMel_CAF1 13297483 109 - 23771897 CUAACUCUUUUUGUUUUAUUUACUAAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUUCC-----CUGGCUUUAGUUUCGGUC-----ACUUAAUGGAUUUAUUCG-AUUUGAAAGAA (((.((((((((((((((.....)))))))))))))).)))(((((..(((((....)))))-----)))))......(((..((-----(...(((.((.....)).-))))))..))) ( -32.60) >DroSec_CAF1 59602 109 - 1 CUAACUCUUUUUGUUUUAUUUACUAAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCCC-----CUGGCUUUAGUUUAGGUC-----ACUUAAUGGAUUUAUUCG-AUUGGAAAGAA (((.((((((((((((((.....)))))))))))))).)))(((((..(((((...))))).-----)))))(((((((((((((-----.(.....)))))))...)-))))))..... ( -31.20) >DroSim_CAF1 60238 109 - 1 CUAACUCUUUUUGUUUUAUUUACUAAAGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCCC-----CUGGCUUAAGUUUCGGUC-----ACUUAAUGGAUUUAUUCG-AUUUGAAAGAA ....((((((((((((((.....)))))))))))))).((((((((..(((((...))))).-----))))))))...(((..((-----(...(((.((.....)).-))))))..))) ( -33.00) >DroEre_CAF1 60295 109 - 1 CUAACUCUUUUUGUUUUAUUAACUCAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCCC-----CUGGCUUUAGUUUCGGUC-----AGUUAAUGGAUUUGUUCA-GCUUGAAAGAA (((.((((((((((((.........)))))))))))).)))(((((..(((((...))))).-----)))))......(((..((-----((....((((....))))-..))))..))) ( -28.50) >DroYak_CAF1 63002 109 - 1 CUAACUCUUUUUGUUUUAUUAACUGAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCGC-----CUGGCUUUAGUUUCGGUC-----AGUUAAUAGAUUUGUUUG-GUUUGAAAGAA (((.((((((((((((((.....)))))))))))))).)))(((((.(((((....))))).-----)))))......(((..((-----((....((((....))))-..))))..))) ( -31.90) >DroAna_CAF1 57462 120 - 1 CUAAUUCUGUUUGUUUUAUUUACGGAUUCAGGAUGAGGUGAACCUGUCGAAGAUCCUUUUCUAGUUUUUGGCAUUACUGUGGAUCUGAGUAACCAGUAGAUUAAAUAGCAUUUACCAGAU .....((((..((((..(((((((((((((((...(((....)))(((((((((.........)))))))))...........)))))))..)).)))))).....)))).....)))). ( -26.10) >consensus CUAACUCUUUUUGUUUUAUUUACUAAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCCC_____CUGGCUUUAGUUUCGGUC_____ACUUAAUGGAUUUAUUCG_AUUUGAAAGAA (((.((((((((((((((.....))))))))))))))..(((((((..(((((...)))))......)))))))......................)))..................... (-19.02 = -19.55 + 0.53)

| Location | 13,297,517 – 13,297,627 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 88.07 |
| Mean single sequence MFE | -37.80 |
| Consensus MFE | -28.50 |
| Energy contribution | -28.82 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.556829 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
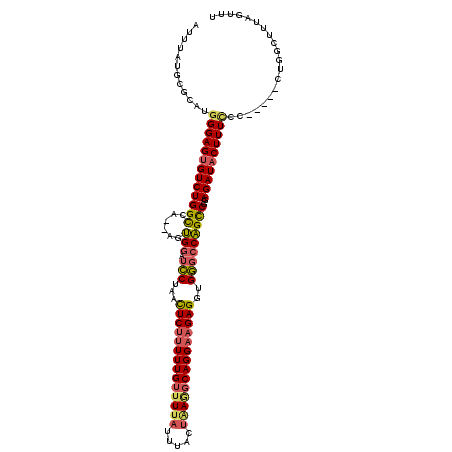
>3L_DroMel_CAF1 13297517 110 - 23771897 AUUUAUGCGCAUGGGAGUGUCUGGCCA--AGUGGAUCCUAACUCUUUUUGUUUUAUUUACUAAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUUCC-----CUGGCUUUAGUUU ......((.((.((((((.((((((..--..(((.(((...((((((((((((((.....))))))))))))))..)))))))))))).)).....)))-----)))))........ ( -39.70) >DroSec_CAF1 59636 110 - 1 AUUUAUGCGCAUGGGAGUGUCUGGCCA--AGUGGAUCCUAACUCUUUUUGUUUUAUUUACUAAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCCC-----CUGGCUUUAGUUU ......((.((.((((((.((((((..--..(((.(((...((((((((((((((.....))))))))))))))..)))))))))))).))....))))-----.))))........ ( -40.70) >DroSim_CAF1 60272 110 - 1 AUUUAUGCGCAUGGGAGUGUCUGGCCA--AGUGGAUCCUAACUCUUUUUGUUUUAUUUACUAAAGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCCC-----CUGGCUUAAGUUU (((((.((.((.((((((.((((((..--..(((.(((...((((((((((((((.....))))))))))))))..)))))))))))).))....))))-----.)))).))))).. ( -42.60) >DroEre_CAF1 60329 110 - 1 AUUUAUGCGCAUGGGAGUGUCUGGCCA--AGUGGAUCCUAACUCUUUUUGUUUUAUUAACUCAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCCC-----CUGGCUUUAGUUU ......((.((.((((((.((((((..--..(((.(((...((((((((((((.........))))))))))))..)))))))))))).))....))))-----.))))........ ( -37.50) >DroYak_CAF1 63036 110 - 1 AUUUAUGCGCAUGGGAGUGUCUGGCCA--AGUGGAUCCUAACUCUUUUUGUUUUAUUAACUGAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCGC-----CUGGCUUUAGUUU ......((.((((..((((((((((..--..(((.(((...((((((((((((((.....))))))))))))))..)))))))))..)))))))..)).-----.))))........ ( -38.80) >DroAna_CAF1 57502 117 - 1 AUUUAUGCACAUCGGAGUGUCUGGUCCACAGAGAACUCUAAUUCUGUUUGUUUUAUUUACGGAUUCAGGAUGAGGUGAACCUGUCGAAGAUCCUUUUCUAGUUUUUGGCAUUACUGU .........(((((((((.((((.....))))..)))))...(((((...........))))).....))))(((....)))(((((((((.........)))))))))........ ( -27.50) >consensus AUUUAUGCGCAUGGGAGUGUCUGGCCA__AGUGGAUCCUAACUCUUUUUGUUUUAUUUACUAAGGCAGGAAGAGGUGGGCCAGCCGGAGAUACUUUCCC_____CUGGCUUUAGUUU ............(((((((((((((......(((.(((...((((((((((((((.....))))))))))))))..)))))))))..)))))))))).................... (-28.50 = -28.82 + 0.31)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:31:33 2006