
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,251,706 – 13,251,861 |
| Length | 155 |
| Max. P | 0.908771 |

| Location | 13,251,706 – 13,251,826 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.83 |
| Mean single sequence MFE | -49.57 |
| Consensus MFE | -35.38 |
| Energy contribution | -34.42 |
| Covariance contribution | -0.97 |
| Combinations/Pair | 1.43 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.80 |
| SVM RNA-class probability | 0.853812 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
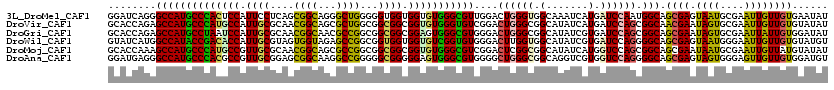
>3L_DroMel_CAF1 13251706 120 - 23771897 GGAUCAGGGCCAUGCCCACUCCAUUCCUCAGCGGCAGGGCUGGGGGUGGUGGUGUGGGCGUUGGACUGGGUGGCAAAUCAUGAUCCAAUGGCAGCGAGUAAUGCGAAUUGUUGUGAAUAU ((((((.((((((((((((.((((((((((((......))))))))..)))).))))))).))).)).(((.....))).))))))..(.(((((((..........))))))).).... ( -51.90) >DroVir_CAF1 2081 120 - 1 GCACCAGAGCCAUGCCCAUGCCAUUGCGCAACGGCAGCGCUGGCGGCGGCGGUGUGGGUGUCGGACUGGGCGGCAUAUCAUGAUCCAGCGGCAACGAAUAGUGCGAAUUGUUGUGUAUAU ((.((((..(((((((((((((...((((....)).))((((....))))))))))))))).)).)))))).(((((.((...((((.((....)).....)).))..)).))))).... ( -44.10) >DroGri_CAF1 2043 120 - 1 GCACCAGAGCCAUGCCUAAUCCAUUGCGCAACGGCAACGCCGGCGGCGGCGGAGUGGGCGUGGGACUGGGCGGCAUAUCGUGAUCCAGCGGCAGCGAAUAGUGCGAAUUAUUGUGGAUAU ((.((((..(((((((((.(((.((((......)))).((((....))))))).)))))))))..))))))...........(((((((....)).((((((....)))))).))))).. ( -49.20) >DroWil_CAF1 18190 120 - 1 GUAUCAUGGCCAUACCGACACCAUUGCGUAGUGGUAGAGCCGGCGGUGGUGGUGUCGGUGUGGGACUUGGUGGCAUAUCGUGAUCCAGGGGCAGCGAGUAAUGGGAAUUGUUGUGUAUGU ....((((.(((((((((((((((..(((...((.....)).)).)..)))))))))))))))..((((((.((.....)).).))))).(((((((..........))))))).)))). ( -48.60) >DroMoj_CAF1 2126 120 - 1 GCACCAAAGCCAUGCCCAUGCCGUUGCGCAACGGCAGCGCCGGCGGCGGCGGUGUGGGCGUCGGACUCGGCGGCAUAUCAUGGUCCAGCGGCAGCGAAUAAUGCGAAUUGUUAUGUAUAU ((((((..(((..((((((((((((((.(..((((...))))..)))))))))))))))((((....)))))))......))))...)).(((...((((((....)))))).))).... ( -50.20) >DroAna_CAF1 6500 120 - 1 GGAUGAGGGCCAUGCCCACGCCGUUGCGGAGCGGCAAGGCCGGGGGCGGGGGAGUGGGCGUGGGGCUGGGCGGCAGGUCGUGGUCCAGGGGCAGCGAGUAGUGGGAGUUGUUGUGGAUGU .........((((((((((.((.((((....((((...))))...)))).)).))))))))))..((((((.((.....)).))))))..(((((((.(......).)))))))...... ( -53.40) >consensus GCACCAGAGCCAUGCCCACGCCAUUGCGCAACGGCAGCGCCGGCGGCGGCGGUGUGGGCGUGGGACUGGGCGGCAUAUCAUGAUCCAGCGGCAGCGAAUAAUGCGAAUUGUUGUGUAUAU ........((((((((((((((.((((....((((...))))...)))).)))))))))))....((((((.(.......).)))))).))).((((.((((....))))))))...... (-35.38 = -34.42 + -0.97)
| Location | 13,251,746 – 13,251,861 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 80.48 |
| Mean single sequence MFE | -43.92 |
| Consensus MFE | -24.40 |
| Energy contribution | -24.57 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.642184 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13251746 115 + 23771897 UGAUUUGCCACCCAGUCCAACGCCCACACCACCACCCCCAGCCCUGCCGCUGAGGAAUGGAGUGGGCAUGGCCCUGAUCCAUGGCAACACCACUGGGCGCAGAUCCUUCAACAAC--- .(((((((..((((((.....((((((.(((....((.((((......)))).))..))).))))))...(((.((...)).)))......)))))).)))))))..........--- ( -45.20) >DroVir_CAF1 2121 114 + 1 UGAUAUGCCGCCCAGUCCGACACCCACACCGCCGCCGCCAGCGCUGCCGUUGCGCAAUGGCAUGGGCAUGGCUCUGGUGCAUGGCAAUACGACUGGCAGGCGUUCCUUUAAUAG---- .((.(((((..((((((......(((...(((((..((((((.((((((((....))))))).).)).))))..)))))..)))......))))))..))))))).........---- ( -45.90) >DroPse_CAF1 16489 118 + 1 CGAUCUGCCGCCCAGUCCCACGCCCACACCACCACCGCCGGCCCUGCCCCUGCGCAAUGGGGUGGGCAUGGCCCUCAUCCAUGGCAACACAACCGGACGGCGAUCCUUCAACAGCAAC .((((.((((.((((..(((.((((((.(((....(((.(((...)))...)))...))).)))))).)))..))......((....)).....)).))))))))............. ( -43.00) >DroMoj_CAF1 2166 114 + 1 UGAUAUGCCGCCGAGUCCGACGCCCACACCGCCGCCGCCGGCGCUGCCGUUGCGCAACGGCAUGGGCAUGGCUUUGGUGCAUGGCAAUACGACCGGCAGGCGUUCCUUCAAUGC---- (((.(((((((((.(((....(((..(((((..((((...((.((((((((....))))))).).)).))))..)))))...))).....))))))).)))))....)))....---- ( -50.60) >DroAna_CAF1 6540 117 + 1 CGACCUGCCGCCCAGCCCCACGCCCACUCCCCCGCCCCCGGCCUUGCCGCUCCGCAACGGCGUGGGCAUGGCCCUCAUCCACGGCAAUACCACCGGACGCCGAUCCUUCAACAGCAG- ....((((.(((..((((.(((((.........(((...))).((((......)))).)))))))))..))).........((((.............))))...........))))- ( -35.82) >DroPer_CAF1 15428 118 + 1 CGAUCUGCCGCCCAGUCCCACGCCCACACCACCACCGCCGGCCCUGCCCCUGCGCAAUGGGGUGGGCAUGGCCCUCAUCCAUGGCAACACAACCGGACGGCGAUCCUUCAACAGCAAC .((((.((((.((((..(((.((((((.(((....(((.(((...)))...)))...))).)))))).)))..))......((....)).....)).))))))))............. ( -43.00) >consensus CGAUCUGCCGCCCAGUCCCACGCCCACACCACCACCGCCGGCCCUGCCGCUGCGCAAUGGCGUGGGCAUGGCCCUCAUCCAUGGCAACACAACCGGACGGCGAUCCUUCAACAGC___ .((.....((((....((...((((((....(((..((((((......)))).))..))).))))))...(((.........))).........))..)))).....))......... (-24.40 = -24.57 + 0.17)


| Location | 13,251,746 – 13,251,861 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 80.48 |
| Mean single sequence MFE | -56.02 |
| Consensus MFE | -40.87 |
| Energy contribution | -42.27 |
| Covariance contribution | 1.39 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.73 |
| SVM decision value | 1.06 |
| SVM RNA-class probability | 0.908771 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
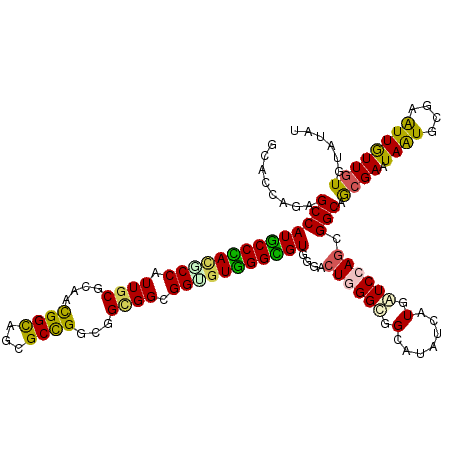
>3L_DroMel_CAF1 13251746 115 - 23771897 ---GUUGUUGAAGGAUCUGCGCCCAGUGGUGUUGCCAUGGAUCAGGGCCAUGCCCACUCCAUUCCUCAGCGGCAGGGCUGGGGGUGGUGGUGUGGGCGUUGGACUGGGUGGCAAAUCA ---..........(((.(((((((((((((.(((........))).)))((((((((.((((((((((((......))))))))..)))).))))))))...))))))).))).))). ( -56.10) >DroVir_CAF1 2121 114 - 1 ----CUAUUAAAGGAACGCCUGCCAGUCGUAUUGCCAUGCACCAGAGCCAUGCCCAUGCCAUUGCGCAACGGCAGCGCUGGCGGCGGCGGUGUGGGUGUCGGACUGGGCGGCAUAUCA ----.............(((..((((((....((((.((((((...(((..(((...((((..((((.......))))))))))))))))))))))))...))))))..)))...... ( -44.50) >DroPse_CAF1 16489 118 - 1 GUUGCUGUUGAAGGAUCGCCGUCCGGUUGUGUUGCCAUGGAUGAGGGCCAUGCCCACCCCAUUGCGCAGGGGCAGGGCCGGCGGUGGUGGUGUGGGCGUGGGACUGGGCGGCAGAUCG .............(((((((((((((((......((........)).((((((((((.((((..(((...(((...))).)).)..)))).))))))))))))))))))))).)))). ( -61.90) >DroMoj_CAF1 2166 114 - 1 ----GCAUUGAAGGAACGCCUGCCGGUCGUAUUGCCAUGCACCAAAGCCAUGCCCAUGCCGUUGCGCAACGGCAGCGCCGGCGGCGGCGGUGUGGGCGUCGGACUCGGCGGCAUAUCA ----.............(((.((((((.((((....)))))))..((((((((((((((((((((.(..((((...))))..))))))))))))))))).)).)).))))))...... ( -54.30) >DroAna_CAF1 6540 117 - 1 -CUGCUGUUGAAGGAUCGGCGUCCGGUGGUAUUGCCGUGGAUGAGGGCCAUGCCCACGCCGUUGCGGAGCGGCAAGGCCGGGGGCGGGGGAGUGGGCGUGGGGCUGGGCGGCAGGUCG -((((((((((....)))))(((((((((.....))...........((((((((((.((.((((....((((...))))...)))).)).)))))))))).)))))))))))).... ( -57.40) >DroPer_CAF1 15428 118 - 1 GUUGCUGUUGAAGGAUCGCCGUCCGGUUGUGUUGCCAUGGAUGAGGGCCAUGCCCACCCCAUUGCGCAGGGGCAGGGCCGGCGGUGGUGGUGUGGGCGUGGGACUGGGCGGCAGAUCG .............(((((((((((((((......((........)).((((((((((.((((..(((...(((...))).)).)..)))).))))))))))))))))))))).)))). ( -61.90) >consensus ___GCUGUUGAAGGAUCGCCGUCCGGUCGUAUUGCCAUGGAUCAGGGCCAUGCCCACGCCAUUGCGCAGCGGCAGGGCCGGCGGCGGUGGUGUGGGCGUGGGACUGGGCGGCAGAUCA .................(((((((((((.....(((.........))).((((((((.(((((((((..((((...)))))).))))))).))))))))..)))))))))))...... (-40.87 = -42.27 + 1.39)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:31:15 2006