
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,124,869 – 13,125,029 |
| Length | 160 |
| Max. P | 0.966671 |

| Location | 13,124,869 – 13,124,989 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.57 |
| Mean single sequence MFE | -55.00 |
| Consensus MFE | -45.54 |
| Energy contribution | -45.98 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.44 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.34 |
| SVM RNA-class probability | 0.695293 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
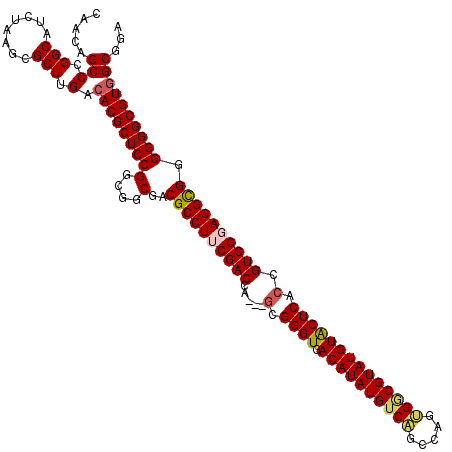
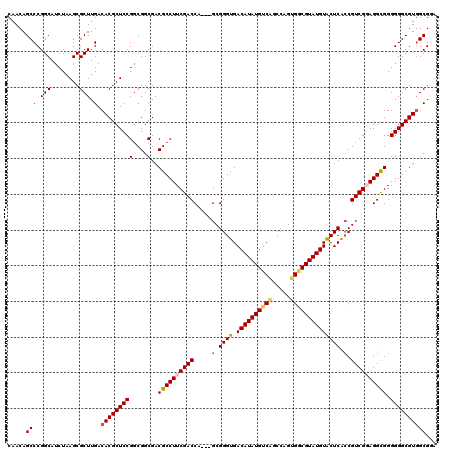
>3L_DroMel_CAF1 13124869 120 + 23771897 CAACAGCCCGGCAUCUAAGCGCUUGACACGCUCCGGCGGCGACGCCUUCGACCAGCAGCGGGUGACAUAUGUCAGCCAGUGGCGUAUGUACUCACCGUCGGAGGCGGGGGGCGUGGCGGA .......(((((......))......((((((((...(....)((((((((((......)((((((((((((((.....)))))))))...))))))))))))))..)))))))).))). ( -52.20) >DroSec_CAF1 112755 117 + 1 CAACAGCCCGGCAUCUAAGCGCUUGACACGCUCCGGCGGCGACGCCUUCGACCA---GCGGGUGACAUAUGUCAGCCAGUGGCGUAUGUACUCACCGUCGUAGGUGGGGGGCGUCGCGGA .......((((......((((.......))))))))(.(((((((((((.(((.---(((((((((((((((((.....)))))))))...)))))..))).))).))))))))))).). ( -56.00) >DroSim_CAF1 110988 117 + 1 CAACAGCCCGGCAUCUAAGCGCUUGACACGCUCCGGCGGCGACGCCUUCGACCA---GCGGGUGACAUAUGUCAGCCAGUGGCGUAUGUACUCACCGUCGUAGGUGGGGGGCGUGGCGUA ..((.(((.((......((((.......))))))))).((.((((((((.(((.---(((((((((((((((((.....)))))))))...)))))..))).))).)))))))).)))). ( -52.10) >DroEre_CAF1 109411 117 + 1 CAACAGCCCGGCAUCUAAGCGCUUGACACGCUCCGGCGGCGACGCCUUCGACCA---GCGGGUGACAUAUGUCAGCCGGCGUCGUAUGUGCUCGGCGUCGGAGGCGGGGGGCGUGGCGAA .....((((.((......))((((..(.(.((((((((.((((((...((((..---.(((.((((....)))).)))..))))...))).))).))))))))).)..))))).)))... ( -53.90) >DroYak_CAF1 113028 117 + 1 CAACAGCCCGGCAUCUAAGCGCUUGACACGCUCCGCCGGCGACGCCUUCGACCA---GCGGGUGACAUAUGUCGCCCAGCGACGUAUGUACUCACCGUCGGAGGCGGGGGGCGUGGCGGA .......(((((......))......((((((((.(((....)(((((((((..---(.(((((.((((((((((...))))))))))))))).).))))))))))))))))))).))). ( -60.80) >consensus CAACAGCCCGGCAUCUAAGCGCUUGACACGCUCCGGCGGCGACGCCUUCGACCA___GCGGGUGACAUAUGUCAGCCAGUGGCGUAUGUACUCACCGUCGGAGGCGGGGGGCGUGGCGGA .....(((.(((........))).).(((((((((....)..((((((((((.....(.((((.((((((((((.....)))))))))))))).).)))))))))).))))))))))... (-45.54 = -45.98 + 0.44)

| Location | 13,124,909 – 13,125,029 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.72 |
| Mean single sequence MFE | -47.02 |
| Consensus MFE | -40.42 |
| Energy contribution | -40.94 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.966671 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
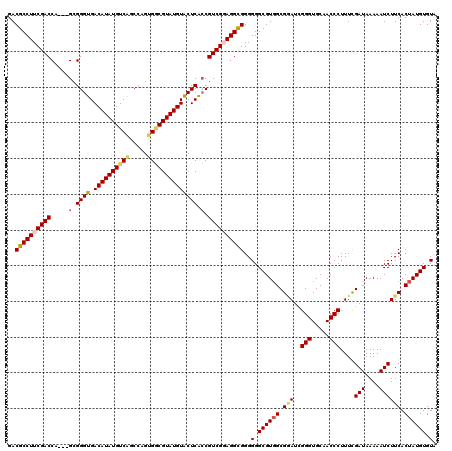
>3L_DroMel_CAF1 13124909 120 + 23771897 GACGCCUUCGACCAGCAGCGGGUGACAUAUGUCAGCCAGUGGCGUAUGUACUCACCGUCGGAGGCGGGGGGCGUGGCGGAUCGGGUGCAACCCUUUCGAUAAAAAUCUUCACUAUGUGUA ..(((((((((((......)((((((((((((((.....)))))))))...)))))))))))))))..(.((((((.(((..(((.....)))....(((....)))))).)))))).). ( -45.50) >DroSec_CAF1 112795 117 + 1 GACGCCUUCGACCA---GCGGGUGACAUAUGUCAGCCAGUGGCGUAUGUACUCACCGUCGUAGGUGGGGGGCGUCGCGGAUCGGGUGCAACCCUUUUGAUAAAAAUCUUCACUAUGUGUA (((((((((.(((.---(((((((((((((((((.....)))))))))...)))))..))).))).)))))))))(((.((.(((.....)))....(((....)))......)).))). ( -46.40) >DroSim_CAF1 111028 117 + 1 GACGCCUUCGACCA---GCGGGUGACAUAUGUCAGCCAGUGGCGUAUGUACUCACCGUCGUAGGUGGGGGGCGUGGCGUAUCGGGUGCAACCCUUUUGAUAAAAAUCUUCACUAUGUGUA .((((((((.(((.---(((((((((((((((((.....)))))))))...)))))..))).))).)))))))).(((((..(((.....)))....(((....))).....)))))... ( -44.60) >DroEre_CAF1 109451 117 + 1 GACGCCUUCGACCA---GCGGGUGACAUAUGUCAGCCGGCGUCGUAUGUGCUCGGCGUCGGAGGCGGGGGGCGUGGCGAAUUGGGUGCAACCCUUUCGAUAAAAAUCUUCACUAUGUGUA ..((((((((((..---((.((((((....)))).)).))((((........))))))))))))))..(.((((((.(((..(((.....)))....(((....)))))).)))))).). ( -47.60) >DroYak_CAF1 113068 117 + 1 GACGCCUUCGACCA---GCGGGUGACAUAUGUCGCCCAGCGACGUAUGUACUCACCGUCGGAGGCGGGGGGCGUGGCGGAUCGGGUGCAACCCUUUCGAUAAAAAUCUUCACUAUGUGUG ..((((((((((..---(.(((((.((((((((((...))))))))))))))).).))))))))))..(.((((((.(((..(((.....)))....(((....)))))).)))))).). ( -51.00) >consensus GACGCCUUCGACCA___GCGGGUGACAUAUGUCAGCCAGUGGCGUAUGUACUCACCGUCGGAGGCGGGGGGCGUGGCGGAUCGGGUGCAACCCUUUCGAUAAAAAUCUUCACUAUGUGUA ..((((((((((.....(.((((.((((((((((.....)))))))))))))).).))))))))))..(.((((((.(((..(((.....)))....(((....)))))).)))))).). (-40.42 = -40.94 + 0.52)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:27:22 2006