
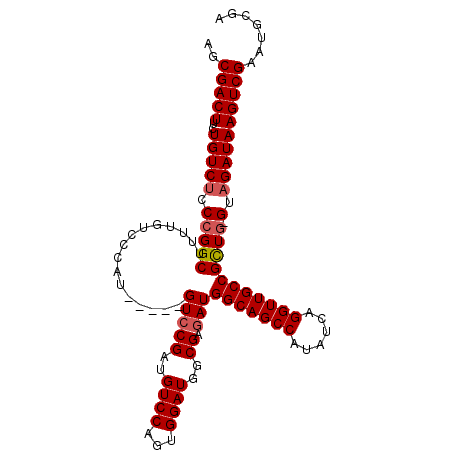
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,083,723 – 13,083,863 |
| Length | 140 |
| Max. P | 0.980688 |

| Location | 13,083,723 – 13,083,823 |
|---|---|
| Length | 100 |
| Sequences | 5 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 93.57 |
| Mean single sequence MFE | -41.90 |
| Consensus MFE | -32.48 |
| Energy contribution | -32.92 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.72 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.63 |
| SVM RNA-class probability | 0.968665 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13083723 100 + 23771897 AGCGACUUCUGUCUCCCGGCUUUUGUCCCAU-----GUCCGAUGUCCAGUGGAUGGCGAGAUGGCAGCCAUAUCAGGUUGCCGCUG-GUAGAUAAGUCGAAUGCGA ..(((((..(((((.((((((((((((....-----(((((........)))))))))))).(((((((......)))))))))))-).))))))))))....... ( -40.40) >DroSec_CAF1 71372 101 + 1 AGCGACUUCUGUCUCCCGGCUUUUGUCCCAU-----GUCCGAUGUCCAGUGGAUGGCGAGAUGGCAGCCAUAUCAGGUUGCCGCUGGGUAGAUAAGUCGAAUGCGA ..(((((..((((((((((((((((((....-----(((((........)))))))))))).(((((((......))))))))))))).))))))))))....... ( -43.40) >DroSim_CAF1 66802 101 + 1 AGCGACUUCUGUCUCCCGGCUUUUGUCCCAU-----GUCCGAUGUCCAGUGGAUGGCGAGAUGGCAGCCAUAUCAGGUUGCCGUUGGGUAGAUAAGUCGAAUGCGA ..(((((..((((((((((((((((((....-----(((((........)))))))))))).(((((((......))))))))))))).))))))))))....... ( -40.90) >DroEre_CAF1 68066 105 + 1 AACGACUUCUGUCCCCAGGCUUUUGUGCCAUGCAGUGUGCGAUGUCCAGUGGAUGGCGAGAUGGCAGCCAUAUCAGGUUGCCGCUG-GUAGAUAAGUCGAAUGCGA ..(((((..((((.((((.......((((((.((.((..(...)..)).)).))))))....(((((((......))))))).)))-)..)))))))))....... ( -40.20) >DroYak_CAF1 70480 105 + 1 AACGACUUCUGUCUCCCGGCUUUUGUCCCAUUCAGUGUCCGUUGUCCAGUGGAUGGCGAGAUGGCAGCCAUAUCAGGUUGCCGCUG-GUAGAUAAGUCGAAUGCGA ..(((((..(((((.(((((...(.(((((((((.((..(...)..)).))))))).)).).(((((((......)))))))))))-).))))))))))....... ( -44.60) >consensus AGCGACUUCUGUCUCCCGGCUUUUGUCCCAU_____GUCCGAUGUCCAGUGGAUGGCGAGAUGGCAGCCAUAUCAGGUUGCCGCUG_GUAGAUAAGUCGAAUGCGA ..(((((..(((((.(((((................(((((..((((...))))..)).)))(((((((......))))))))))).).))))))))))....... (-32.48 = -32.92 + 0.44)


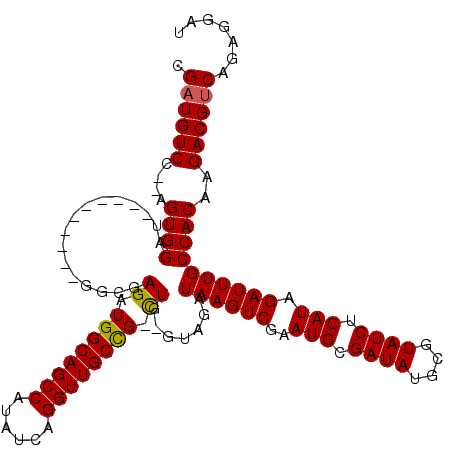
| Location | 13,083,757 – 13,083,863 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.01 |
| Mean single sequence MFE | -39.60 |
| Consensus MFE | -33.68 |
| Energy contribution | -33.57 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.76 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.980688 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13083757 106 + 23771897 CGAUGUCC--AGUGGAU----------GGCGAGAUGGCAGCCAUAUCAGGUUGCCG-CUG-GUAGAUAAGUCGAAUGCGAUAUGCGUAUCUCAUAGAUUUGCCACAAGACGUCAGAGGAU .((((((.--.((((..----------.((.((.((((((((......))))))))-)).-))...((((((..(((.((((....)))).))).))))))))))..))))))....... ( -40.30) >DroSec_CAF1 71406 107 + 1 CGAUGUCC--AGUGGAU----------GGCGAGAUGGCAGCCAUAUCAGGUUGCCG-CUGGGUAGAUAAGUCGAAUGCGAUAUGCGUAUCUCAUAGAUUUGCCACAAGACGUCAGAGGAU .((((((.--.((((.(----------(.(.((.((((((((......))))))))-)).).))..((((((..(((.((((....)))).))).))))))))))..))))))....... ( -39.80) >DroSim_CAF1 66836 107 + 1 CGAUGUCC--AGUGGAU----------GGCGAGAUGGCAGCCAUAUCAGGUUGCCG-UUGGGUAGAUAAGUCGAAUGCGAUAUGCGUAUCUCAUAGAUUUGCCACAAGACGUCAGAGGAU .((((((.--.((((..----------.((..((((((((((......))))))))-))..))...((((((..(((.((((....)))).))).))))))))))..))))))....... ( -40.50) >DroEre_CAF1 68105 106 + 1 CGAUGUCC--AGUGGAU----------GGCGAGAUGGCAGCCAUAUCAGGUUGCCG-CUG-GUAGAUAAGUCGAAUGCGAUAUGCGUAUCUCAUAGAUUUGCCACAAGACGUCAGAGGAU .((((((.--.((((..----------.((.((.((((((((......))))))))-)).-))...((((((..(((.((((....)))).))).))))))))))..))))))....... ( -40.30) >DroYak_CAF1 70519 106 + 1 CGUUGUCC--AGUGGAU----------GGCGAGAUGGCAGCCAUAUCAGGUUGCCG-CUG-GUAGAUAAGUCGAAUGCGAUAUGCGUAUCUCAUAGAUUUGCCACAAGACGUCAGAGGAU (((..(((--...))).----------.))).((((((((((......)))))))(-.((-((((((..((.(((((((.....))))).))))..))))))))).....)))....... ( -35.60) >DroAna_CAF1 65771 115 + 1 -GAUGUCCACAGUGGCUUUCCAGCCCCGGAGAGAUGGCAGCCAUAUCAGGUUGCUGGCUG-CUAGAUAAGUCGAAUGCGAUAUGCGUAUCUCAUAGAUUUGCCACAAGACGUCGCAG--- -((((((....((((((((((......)))))..(((((((((((......)).))))))-)))...(((((..(((.((((....)))).))).))))))))))..))))))....--- ( -41.10) >consensus CGAUGUCC__AGUGGAU__________GGCGAGAUGGCAGCCAUAUCAGGUUGCCG_CUG_GUAGAUAAGUCGAAUGCGAUAUGCGUAUCUCAUAGAUUUGCCACAAGACGUCAGAGGAU .((((((....((((................((.((((((((......)))))))).)).......((((((..(((.((((....)))).))).))))))))))..))))))....... (-33.68 = -33.57 + -0.11)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:26:13 2006