
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 13,003,450 – 13,003,575 |
| Length | 125 |
| Max. P | 0.926351 |

| Location | 13,003,450 – 13,003,541 |
|---|---|
| Length | 91 |
| Sequences | 5 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 81.36 |
| Mean single sequence MFE | -27.60 |
| Consensus MFE | -19.08 |
| Energy contribution | -20.76 |
| Covariance contribution | 1.68 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.16 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.915040 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
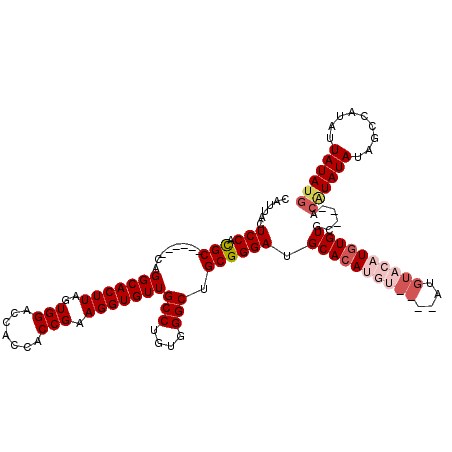
>3L_DroMel_CAF1 13003450 91 - 23771897 CCGAAGGUGUUGCCUGUGGGCUGCGGGAUGCACAUGU----AUGUACAUCUGUGUACUACAUAUAUAGCCAUAUUAUAUGCGUUUUUUAUGCCAU--------- .((.(((.....))).))(((((..((((((..((((----(.(((((....))))))))))((((((.....))))))))))))..)).)))..--------- ( -24.00) >DroSec_CAF1 10473 92 - 1 CCGAAGGUGUUGCCUGUGGGCUGCGGGAUGCACAUGUACAUAUGUACAUGUGUGC---ACAUAUAUAGCCAUAUUAUAUGCGUUCUUUAUGCCAU--------- .((.(((.....))).))(((...((((.((((((((((....))))))))))((---.((((((.........)))))).))))))...)))..--------- ( -29.10) >DroSim_CAF1 13001 92 - 1 CCGAAGGUGUUGCCUGUGGGCUGCGGGAUGCACAUGUACAUAUGUACAUGUGUGC---ACAUAUAUAGCCAUAUUAUAUGCGUUCUUUAUGCCAU--------- .((.(((.....))).))(((...((((.((((((((((....))))))))))((---.((((((.........)))))).))))))...)))..--------- ( -29.10) >DroEre_CAF1 10398 81 - 1 CCGAAGGUGUUGCCU---GGCUGCGGGAUGCACAC----------ACAUGUGUGCACU-CGUAUAUGGCCAUAUUAUAUGCGUUCUGUAUGCCAU--------- ....(((.....)))---(((((((((((((((((----------....)))))))..-(((((((((.....)))))))))))))))).)))..--------- ( -31.40) >DroYak_CAF1 10880 84 - 1 CCGAAGGUGUUGCCU---GGCUGCGGGAUGCACAC----------ACAUGUGUG-------UAUAUAGCCAUAUUAUAUGCGUUCUUUAUGCCAUACUAUACUG .....(((((.(..(---(((((.(((((((((((----------(....))))-------)((((((.....))))))))))))).)).))))..).))))). ( -24.40) >consensus CCGAAGGUGUUGCCUGUGGGCUGCGGGAUGCACAUGU____AUGUACAUGUGUGC___ACAUAUAUAGCCAUAUUAUAUGCGUUCUUUAUGCCAU_________ .....(((((.(((....))).))(((((((((((((((....))))))))...........((((((.....)))))))))))))....)))........... (-19.08 = -20.76 + 1.68)

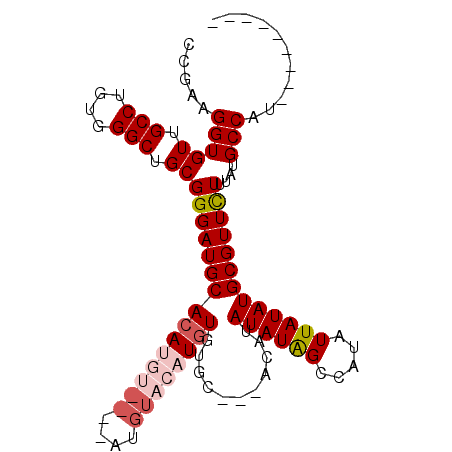
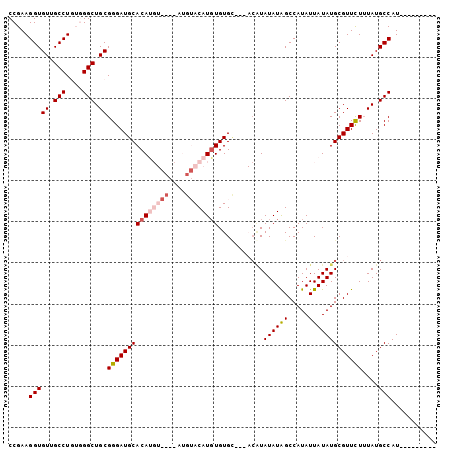
| Location | 13,003,465 – 13,003,575 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.01 |
| Mean single sequence MFE | -36.34 |
| Consensus MFE | -23.36 |
| Energy contribution | -25.88 |
| Covariance contribution | 2.52 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.909737 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13003465 110 + 23771897 CAUAUAAUAUGGCUAUAUAUGUAGUACACAGAUGUACAU----ACAUGUGCAUCCCGCAGCCCACAGGCAACACCUUCGGUGGUGGUCCACUAAGUGCCUG------GCGUGGAGUAAUG ...........(((.(((((((((((((....))))).)----)))))))....((((.(((...(((((....(((.(((((....))))))))))))))------))))))))).... ( -35.00) >DroSec_CAF1 10488 111 + 1 CAUAUAAUAUGGCUAUAUAUGU---GCACACAUGUACAUAUGUACAUGUGCAUCCCGCAGCCCACAGGCAACACCUUCGGUGGUGGUCCACUAAGUGCCUG------GCGUGGAGUAAUG ((((((.........))))))(---((.(((((((((....)))))))))....((((.(((...(((((....(((.(((((....))))))))))))))------)))))).)))... ( -37.90) >DroSim_CAF1 13016 111 + 1 CAUAUAAUAUGGCUAUAUAUGU---GCACACAUGUACAUAUGUACAUGUGCAUCCCGCAGCCCACAGGCAACACCUUCGGUGGUGGUCCACUAAGUGCCUG------GCAUGGAGCAAUG ...........(((((((((((---(((....))))))))))).(((((((.....))......((((((....(((.(((((....))))))))))))))------))))).))).... ( -37.00) >DroEre_CAF1 10413 97 + 1 CAUAUAAUAUGGCCAUAUACG-AGUGCACACAUGU----------GUGUGCAUCCCGCAGCC---AGGCAACACCUUCGGU---GGUCCACUAAGUGCCUG------GCGCGGAGCAAUG ...........((........-.((((((((....----------)))))))).((((.(((---(((((.((((...)))---)..........))))))------)))))).)).... ( -39.70) >DroYak_CAF1 10904 97 + 1 CAUAUAAUAUGGCUAUAUA-------CACACAUGU----------GUGUGCAUCCCGCAGCC---AGGCAACACCUUCGGU---GGUCCACUAAGUGCCUGCCUGGAGCGCGGAGUAAUG ...........(((.((((-------(((....))----------)))))....((((..((---(((((.(((.((.(((---(...)))))))))..)))))))...))))))).... ( -32.10) >consensus CAUAUAAUAUGGCUAUAUAUGU___GCACACAUGUACAU____ACAUGUGCAUCCCGCAGCCCACAGGCAACACCUUCGGUGGUGGUCCACUAAGUGCCUG______GCGUGGAGUAAUG ...........((...............(((((((((....)))))))))..((((((......((((((....(((.(((((....))))))))))))))......))).))))).... (-23.36 = -25.88 + 2.52)

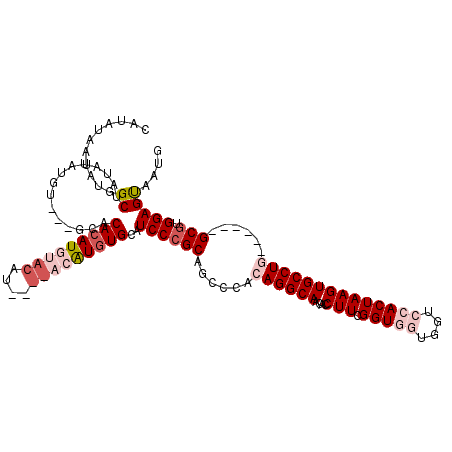
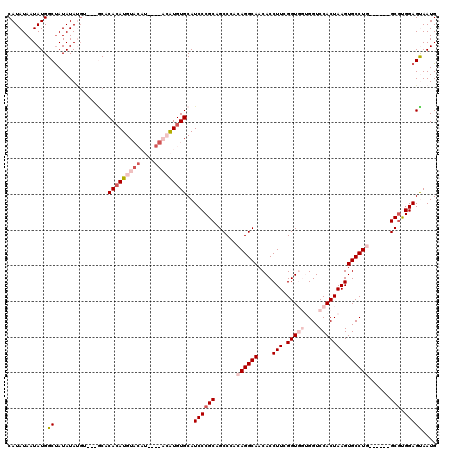
| Location | 13,003,465 – 13,003,575 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.01 |
| Mean single sequence MFE | -37.18 |
| Consensus MFE | -24.60 |
| Energy contribution | -26.72 |
| Covariance contribution | 2.12 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.18 |
| SVM RNA-class probability | 0.926351 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 13003465 110 - 23771897 CAUUACUCCACGC------CAGGCACUUAGUGGACCACCACCGAAGGUGUUGCCUGUGGGCUGCGGGAUGCACAUGU----AUGUACAUCUGUGUACUACAUAUAUAGCCAUAUUAUAUG .............------..(((.(((.((((....))))..)))((((..(((((.....)))))..))))((((----(.(((((....)))))))))).....))).......... ( -30.50) >DroSec_CAF1 10488 111 - 1 CAUUACUCCACGC------CAGGCACUUAGUGGACCACCACCGAAGGUGUUGCCUGUGGGCUGCGGGAUGCACAUGUACAUAUGUACAUGUGUGC---ACAUAUAUAGCCAUAUUAUAUG ......(((.(((------..(((((((.((((....))))...)))))))(((....))).)))))).((((((((((....))))))))))..---.((((((.........)))))) ( -38.20) >DroSim_CAF1 13016 111 - 1 CAUUGCUCCAUGC------CAGGCACUUAGUGGACCACCACCGAAGGUGUUGCCUGUGGGCUGCGGGAUGCACAUGUACAUAUGUACAUGUGUGC---ACAUAUAUAGCCAUAUUAUAUG ...((((((.(((------..(((((((.((((....))))...)))))))(((....))).)))))).((((((((((....))))))))))))---)((((((.........)))))) ( -38.30) >DroEre_CAF1 10413 97 - 1 CAUUGCUCCGCGC------CAGGCACUUAGUGGACC---ACCGAAGGUGUUGCCU---GGCUGCGGGAUGCACAC----------ACAUGUGUGCACU-CGUAUAUGGCCAUAUUAUAUG ((((((.((((((------((((((((....))..(---(((...)))).)))))---))).))))(((((((((----------....))))))).)-)))).)))............. ( -39.80) >DroYak_CAF1 10904 97 - 1 CAUUACUCCGCGCUCCAGGCAGGCACUUAGUGGACC---ACCGAAGGUGUUGCCU---GGCUGCGGGAUGCACAC----------ACAUGUGUG-------UAUAUAGCCAUAUUAUAUG ......((((((..((((((((.(((((..(((...---.))).)))))))))))---)).))))))((((((((----------....)))))-------)))................ ( -39.10) >consensus CAUUACUCCACGC______CAGGCACUUAGUGGACCACCACCGAAGGUGUUGCCUGUGGGCUGCGGGAUGCACAUGU____AUGUACAUGUGUGC___ACAUAUAUAGCCAUAUUAUAUG ......(((.(((........(((((((..(((.......))).)))))))(((....))).)))))).((((((((((....))))))))))......((((((.........)))))) (-24.60 = -26.72 + 2.12)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:25:19 2006