
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,994,435 – 12,994,669 |
| Length | 234 |
| Max. P | 0.972319 |

| Location | 12,994,435 – 12,994,552 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.36 |
| Mean single sequence MFE | -46.25 |
| Consensus MFE | -25.86 |
| Energy contribution | -26.76 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.56 |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.901250 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
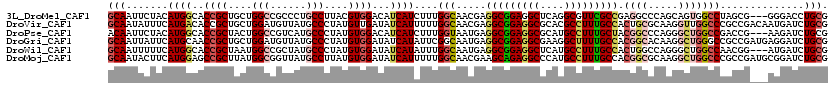
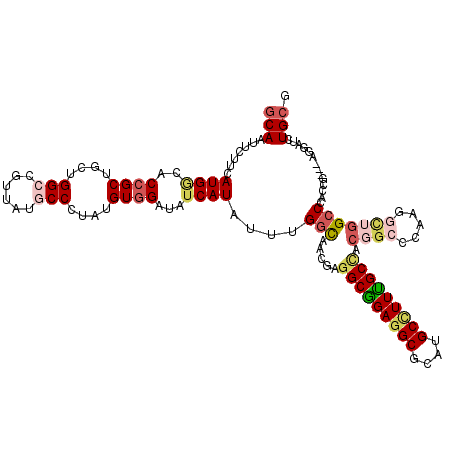
>3L_DroMel_CAF1 12994435 117 - 23771897 CGAGAUUGGCAAGCGACUGGUGGCGAUGGAGAAGAAAAACCCGACGCGGCCGCGCAUUGCCAUUCUCACGCAGGGCUGCGAUCCUGUACUGCUUAUACAGCAGGAU---UCGGUGCAGGA .((((.(((((((((...(((.(((.(((.(........)))).))).))).))).)))))).)))).((((....)))).(((((((((((((.......)))..---.)))))))))) ( -51.10) >DroVir_CAF1 1677 117 - 1 UGAGAUUGGCAAGCGCCUGGUCGCGCUGAAUAAACUCAAUACGGGGCGACCACGCAUUGCCAUACUCACGCAGGGCUGUGACCCCGUGCUGCUUAUACAGCACGAU---UCGGUCCAGGA ((((..((((((..((.(((((((.(((.((.......)).))).))))))).)).))))))..)))).......(((.((((.(((((((......)))))))..---..))))))).. ( -52.70) >DroGri_CAF1 1527 120 - 1 UGAGAUUGGCAGGCGUCUGGUUGCACUGGACAAAUUGAAUUCAGCGCGACCACGCAUUGCGAUACUCACGCAGGGCUGUGAUCCCGUGCUGCUCAUCCAGCACGACGCUCCGGUCCAGGA ((((..(.((((((((..((((((.(((((.........))))).)))))))))).)))).)..))))....((((((.((.(.(((((((......)))))))..).)))))))).... ( -48.30) >DroWil_CAF1 2357 117 - 1 CGAGAUUGGCAAACGUAUAGUCGGUCUGGAGAAACUAAAUCCAAGUCGGCCACGCAUUGCCAUACUAACGCAGGGCUGUGAUCCGGUGUUGCUCAUUCAAAAAGAU---UCGGUUGAAGA ((((..((((((.(((...(((((..((((.........))))..))))).)))..)))))).......((((.((((.....)))).)))).............)---)))........ ( -32.70) >DroMoj_CAF1 1595 117 - 1 UGAGAUUGGCAAACGUUUGGUGGCACUGCCCAAAAUCAAUUCAGAGCGACCACGCAUUGCCAUACUCACGCAGGGCUGUGACCCCGUGCUGCUCAUCCAGCACGAC---AAGGUCCAGGA ((((..((((((..((.((((.((.(((.............))).)).)))).)).))))))..)))).......(((.((((.(((((((......)))))))..---..))))))).. ( -43.12) >DroAna_CAF1 1509 117 - 1 AGAGAUUGGCAAGCGGCUGGUGGCGUUCGACAAGCUGAACUCGUCGAGGCCGCGCAUUGCCAUUCUCACGCAGGGCUGUGAUCCCGUGCUGCUCUUCCAGCAGGAC---UCGGUGCAGGA .((((.((((((...((..(((((.((((((.((.....)).))))))))))))).)))))).))))..((((((.......)))((.(((((.....))))).))---....))).... ( -49.60) >consensus UGAGAUUGGCAAGCGUCUGGUGGCGCUGGACAAACUAAAUUCAGCGCGACCACGCAUUGCCAUACUCACGCAGGGCUGUGAUCCCGUGCUGCUCAUCCAGCACGAC___UCGGUCCAGGA .(((..(((((((((..((((.((..(((...........)))..)).))))))).))))))..))).....((((((........(((((......)))))........)))))).... (-25.86 = -26.76 + 0.89)

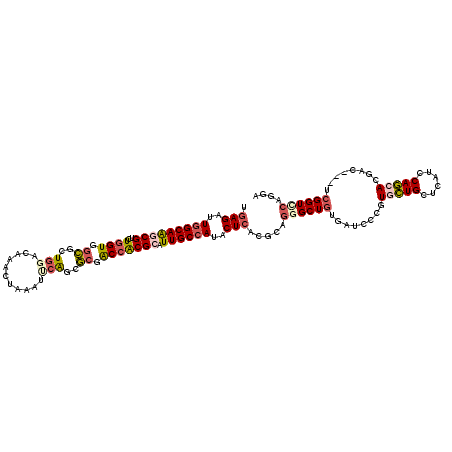

| Location | 12,994,472 – 12,994,589 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.72 |
| Mean single sequence MFE | -52.65 |
| Consensus MFE | -33.10 |
| Energy contribution | -35.43 |
| Covariance contribution | 2.34 |
| Combinations/Pair | 1.23 |
| Mean z-score | -3.00 |
| Structure conservation index | 0.63 |
| SVM decision value | 1.66 |
| SVM RNA-class probability | 0.970445 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12994472 117 + 23771897 CGCAGCCCUGCGUGAGAAUGGCAAUGCGCGGCCGCGUCGGGUUUUUCUUCUCCAUCGCCACCAGUCGCUUGCCAAUCUCGCGCAGGUCCC---CGCUAGGCCACUGCUGGGCCUCGGCGA ......(((((((((((.((((((.((((((..(((..(((.........)))..)))..)).).))))))))).)))))))))))....---(((((((((.(....)))))).)))). ( -58.40) >DroVir_CAF1 1714 120 + 1 CACAGCCCUGCGUGAGUAUGGCAAUGCGUGGUCGCCCCGUAUUGAGUUUAUUCAGCGCGACCAGGCGCUUGCCAAUCUCACGCAGAUCAUUGUCGGCGGGCCAACCUUGCGCAGUGGCAA ....((((((((((((..(((((((((.(((((((...((..((((....))))))))))))).))).))))))..)))))))))...(((((..(((((.....))))))))))))).. ( -55.50) >DroPse_CAF1 1301 117 + 1 CACAGCCCUGCGUUAGUAUGGCAAUGCGAGGUCGCGCCGGAUUCAGUUUGUCCAGCGCCACCAGACGCUUGCCAAUCUCGCGCAGAUCUU---CGGUCGGCCAGCCCUGGGCCGUAGCAA ....((.((((((.((..((((((.(((.(((.((((.((((.......)))).)))).)))...)))))))))..)).)))))).....---....(((((.......)))))..)).. ( -50.90) >DroWil_CAF1 2394 117 + 1 CACAGCCCUGCGUUAGUAUGGCAAUGCGUGGCCGACUUGGAUUUAGUUUCUCCAGACCGACUAUACGUUUGCCAAUCUCGCGCAGAUCAU---CCGUUGGCCAGCCCUGGCCAGUGGCAA ....(((((((((.((..((((((.(((((..((..(((((.........)))))..))....)))))))))))..)).)))))).....---...(((((((....))))))).))).. ( -44.80) >DroAna_CAF1 1546 117 + 1 CACAGCCCUGCGUGAGAAUGGCAAUGCGCGGCCUCGACGAGUUCAGCUUGUCGAACGCCACCAGCCGCUUGCCAAUCUCUCGCAGAUCCC---CACUCGGCCAGCUCUGGGCCUCGGCGA ....((((((((.((((.((((((.(((((((.((((((((.....))))))))..)))....).))))))))).)))).))))).....---.....((((.......))))..))).. ( -55.40) >DroPer_CAF1 1301 117 + 1 CACAGCCCUGCGUUAGUAUGGCAAUGCGAGGUCGCGCCGGAUUCAGUUUGUCCAGCGCCACCAGACGCUUGCCAAUCUCGCGCAGAUCUU---CGGUCGGCCAGCCCUGGGCCGUAGCAA ....((.((((((.((..((((((.(((.(((.((((.((((.......)))).)))).)))...)))))))))..)).)))))).....---....(((((.......)))))..)).. ( -50.90) >consensus CACAGCCCUGCGUGAGUAUGGCAAUGCGAGGCCGCGCCGGAUUCAGUUUGUCCAGCGCCACCAGACGCUUGCCAAUCUCGCGCAGAUCAU___CGGUCGGCCAGCCCUGGGCCGUGGCAA ....(((((((((.((..((((((.(((.((..((((.((((.......)))).))))..))...)))))))))..)).))))))............(((((.......))))).))).. (-33.10 = -35.43 + 2.34)


| Location | 12,994,472 – 12,994,589 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.72 |
| Mean single sequence MFE | -55.87 |
| Consensus MFE | -35.85 |
| Energy contribution | -36.30 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.06 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.972319 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12994472 117 - 23771897 UCGCCGAGGCCCAGCAGUGGCCUAGCG---GGGACCUGCGCGAGAUUGGCAAGCGACUGGUGGCGAUGGAGAAGAAAAACCCGACGCGGCCGCGCAUUGCCAUUCUCACGCAGGGCUGCG ((.(((((((((....).)))))..))---).))((((((.((((.(((((((((...(((.(((.(((.(........)))).))).))).))).)))))).)))).))))))...... ( -60.70) >DroVir_CAF1 1714 120 - 1 UUGCCACUGCGCAAGGUUGGCCCGCCGACAAUGAUCUGCGUGAGAUUGGCAAGCGCCUGGUCGCGCUGAAUAAACUCAAUACGGGGCGACCACGCAUUGCCAUACUCACGCAGGGCUGUG ....(((.((.((..(((((....)))))..)).((((((((((..((((((..((.(((((((.(((.((.......)).))).))))))).)).))))))..)))))))))))).))) ( -59.30) >DroPse_CAF1 1301 117 - 1 UUGCUACGGCCCAGGGCUGGCCGACCG---AAGAUCUGCGCGAGAUUGGCAAGCGUCUGGUGGCGCUGGACAAACUGAAUCCGGCGCGACCUCGCAUUGCCAUACUAACGCAGGGCUGUG ....((((((((..((....)).....---......((((..((..(((((((((...(((.((((((((.........)))))))).))).))).))))))..))..)))))))))))) ( -56.20) >DroWil_CAF1 2394 117 - 1 UUGCCACUGGCCAGGGCUGGCCAACGG---AUGAUCUGCGCGAGAUUGGCAAACGUAUAGUCGGUCUGGAGAAACUAAAUCCAAGUCGGCCACGCAUUGCCAUACUAACGCAGGGCUGUG ..(((..((((((....))))))....---.....(((((..((..((((((.(((...(((((..((((.........))))..))))).)))..))))))..))..)))))))).... ( -46.20) >DroAna_CAF1 1546 117 - 1 UCGCCGAGGCCCAGAGCUGGCCGAGUG---GGGAUCUGCGAGAGAUUGGCAAGCGGCUGGUGGCGUUCGACAAGCUGAACUCGUCGAGGCCGCGCAUUGCCAUUCUCACGCAGGGCUGUG ((.(((.((((.......))))...))---).))((((((.((((.((((((...((..(((((.((((((.((.....)).))))))))))))).)))))).)))).))))))...... ( -56.60) >DroPer_CAF1 1301 117 - 1 UUGCUACGGCCCAGGGCUGGCCGACCG---AAGAUCUGCGCGAGAUUGGCAAGCGUCUGGUGGCGCUGGACAAACUGAAUCCGGCGCGACCUCGCAUUGCCAUACUAACGCAGGGCUGUG ....((((((((..((....)).....---......((((..((..(((((((((...(((.((((((((.........)))))))).))).))).))))))..))..)))))))))))) ( -56.20) >consensus UUGCCACGGCCCAGGGCUGGCCGACCG___AAGAUCUGCGCGAGAUUGGCAAGCGUCUGGUGGCGCUGGACAAACUGAAUCCGGCGCGACCACGCAUUGCCAUACUAACGCAGGGCUGUG ..(((..((((.......)))).............(((((..((..(((((((((...(((.(((.(((...........))).))).))).))).))))))..))..)))))))).... (-35.85 = -36.30 + 0.45)

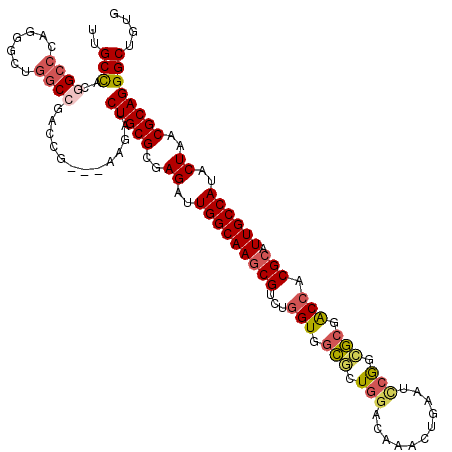
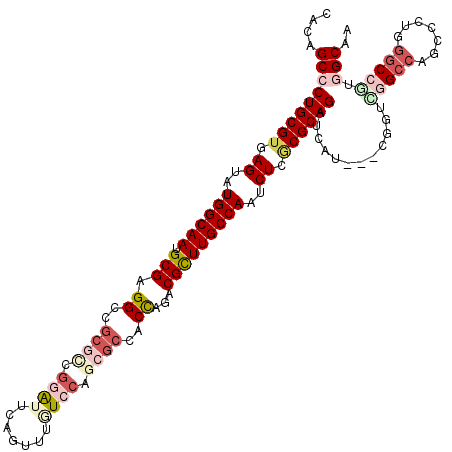
| Location | 12,994,552 – 12,994,669 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.78 |
| Mean single sequence MFE | -48.50 |
| Consensus MFE | -27.32 |
| Energy contribution | -28.80 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.826698 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12994552 117 - 23771897 GCAAUUCUACAUGGCACCGCUGCUGGCCGCCCUGCCUUACGUGGACAUCAUCUUUGGCAACGAGGCGGAGGCUCAGGCGUUCGCCGAGGCCCAGCAGUGGCCUAGCG---GGGACCUGCG (((.((((...(((..(((((((((((((((((((((..(((...((.......))...))))))))).)))...(((....)))..)).)))))))))).)))...---))))..))). ( -54.20) >DroVir_CAF1 1794 120 - 1 GCAAUAUUUCAUGACACCGCUGCUGGAUGUUAUGCCCUAUGUUGAUAUCAUUUUUGGCAACGAGGCGGAGGCGCACGCCUUUGCCACUGCGCAAGGUUGGCCCGCCGACAAUGAUCUGCG .((((((..((((((((((....))).)))))))....))))))..((((((.(((((.....(((((((((....)))))))))....(....)........))))).))))))..... ( -42.90) >DroPse_CAF1 1381 117 - 1 ACAAUUCUACAUGGCACCGCUACUGGCCGUCAUGCCCUAUGUGGACAUCAUCUUUGGUAAUGAGGCGGAGGCGCAUGCCUUUGCUACGGCCCAGGGCUGGCCGACCG---AAGAUCUGCG .....(((((((((((..((........))..))))..)))))))....(((((((((.....(((((((((....))))))))).(((((.......)))))))))---)))))..... ( -45.40) >DroGri_CAF1 1647 120 - 1 GCAAUUAUUCAUGCAACCGCUGCUGGAUGUUAUGCCCUAUGUGGAUAUCAUAUUCGGCAAUGAGGCGGAGGCGAAGGCUUUUGCCACGGCACAAGGCUGGGCCGCCGAUGAGGAUCUGCG (((..((((((.(((.....)))))))))...))).....(..(((.((((...((((.....(((((((((....))))))))).((((.....)))).))))...))))..)))..). ( -47.80) >DroWil_CAF1 2474 117 - 1 GCAAUUUUUCAUGGCACCGCUAAUGGCCGCUAUGCCCUAUGUGGAUAUCAUAUUUGGCAAUGAGGCGGAGGCUCAUGCCUUUGCCACUGGCCAGGGCUGGCCAACGG---AUGAUCUGCG (((..((.((.((((.(.(((..(((((....((((..(((((.....)))))..))))....(((((((((....)))))))))...))))).))).)))))...)---).))..))). ( -51.20) >DroMoj_CAF1 1712 120 - 1 GCAAUACUUCAUGGAGCCGCUUAUGGCGGUUAUGCCUUAUGUGGAUAUCAUUUUUGGCAACGAAGCAGAGGCCCAUGCCUUUGCCACGGCGCAAGGCUGGCCCGCCGAUGCGGAUCUGCG (((...(((....)))((((...(((((((((.(((((.(((.(........((((....))))((((((((....))))))))..).))).)))))))).))))))..))))...))). ( -49.50) >consensus GCAAUUCUUCAUGGCACCGCUGCUGGCCGUUAUGCCCUAUGUGGAUAUCAUAUUUGGCAACGAGGCGGAGGCGCAUGCCUUUGCCACGGCCCAAGGCUGGCCCACCG___AGGAUCUGCG (((.......((((..((((....(((......)))....))))...))))....(((.....(((((((((....))))))))).((((.....)))))))..............))). (-27.32 = -28.80 + 1.48)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:25:06 2006