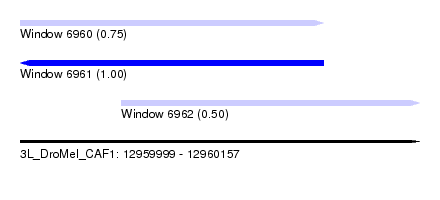
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,959,999 – 12,960,157 |
| Length | 158 |
| Max. P | 0.999943 |

| Location | 12,959,999 – 12,960,119 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.41 |
| Mean single sequence MFE | -27.83 |
| Consensus MFE | -25.44 |
| Energy contribution | -25.16 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.750879 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12959999 120 + 23771897 CUGAAACAACCCACUCCGUGUGAUAUAAAAAUCAUAUACACACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGACGCACAACAAAAUGUUUUGUUUCUC ..(((((((.(((...((((((((((.......)))).))))))....)))....((((((((.(((((((.........))))...(((.....))).))).))))))))))))))).. ( -29.20) >DroSec_CAF1 77231 120 + 1 CUGAAACAACCCACUCCGUGUGGUAUAAAAAUCAUAUACACACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGAGGCACAACAAAAUGUUUUGUUUUUC ..(((((((.((((.((((((((((((.......))))).))))....)))..))))((((((.(((...((.(((((((..((...))))))))))).))).))))))..))))))).. ( -27.50) >DroSim_CAF1 81982 120 + 1 CUGAAACAACCCACUCCGUGUGGUAUAAAAAUCAUAUACACACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGACGCACAACAAAAUGUUUUGUUUUUC ..(((((((.((((.((((((((((((.......))))).))))....)))..))))((((((.(((((((.........))))...(((.....))).))).))))))..))))))).. ( -26.90) >DroEre_CAF1 83220 119 + 1 CAGAAACAACCCACUCCGCGUGGUAUAAAAAUCAUAUACACACGUUUCUGGCUGCGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGAUGCACAACAAAAUGUUUUGUUUU-C ..(((((((.(((....((((((((((.......))))).)))))...)))....((((((((.((((((.(((.................))).))).))).)))))))))))))))-. ( -28.23) >DroYak_CAF1 81080 119 + 1 CAGGAACAACCCACUCCGUGUGGUAUAAAAAUCAUAUACACACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUCGCAAAGCGUGAGACGCACAACAAAAUGUUUUGUUUU-C (((((((....(((.....)))(((((.......)))))....)))))))((...((((((((.(((((..(((.......(((...))).)))..)).))).))))))))..))...-. ( -27.30) >consensus CUGAAACAACCCACUCCGUGUGGUAUAAAAAUCAUAUACACACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGACGCACAACAAAAUGUUUUGUUUUUC ..(((((((.(((...((((((.((((.......))))))))))....)))....((((((((.(((((..(((.......(((...))).)))..)).))).))))))))))))))).. (-25.44 = -25.16 + -0.28)

| Location | 12,959,999 – 12,960,119 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.41 |
| Mean single sequence MFE | -33.81 |
| Consensus MFE | -33.01 |
| Energy contribution | -32.77 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.98 |
| SVM decision value | 4.72 |
| SVM RNA-class probability | 0.999943 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12959999 120 - 23771897 GAGAAACAAAACAUUUUGUUGUGCGUCUCACGCUUUGCAAAUAAAAGAGCUGCCAAUAAAAUGCCACAGCCAGAAACGUGUGUAUAUGAUUUUUAUAUCACACGGAGUGGGUUGUUUCAG ..(((((....((((((((((.(((.....)(((((.........))))).))))))))))))....((((.....((((((.((((((...)))))))))))).....))))))))).. ( -35.60) >DroSec_CAF1 77231 120 - 1 GAAAAACAAAACAUUUUGUUGUGCCUCUCACGCUUUGCAAAUAAAAGAGCUGCCAAUAAAAUGCCACAGCCAGAAACGUGUGUAUAUGAUUUUUAUACCACACGGAGUGGGUUGUUUCAG (((........((((((((((.((..(((.................)))..))))))))))))..((((((.....((((((((((.......)))).)))))).....))))))))).. ( -32.33) >DroSim_CAF1 81982 120 - 1 GAAAAACAAAACAUUUUGUUGUGCGUCUCACGCUUUGCAAAUAAAAGAGCUGCCAAUAAAAUGCCACAGCCAGAAACGUGUGUAUAUGAUUUUUAUACCACACGGAGUGGGUUGUUUCAG (((........((((((((((.(((.....)(((((.........))))).))))))))))))..((((((.....((((((((((.......)))).)))))).....))))))))).. ( -33.90) >DroEre_CAF1 83220 119 - 1 G-AAAACAAAACAUUUUGUUGUGCAUCUCACGCUUUGCAAAUAAAAGAGCUGCCAAUAAAAUGCCGCAGCCAGAAACGUGUGUAUAUGAUUUUUAUACCACGCGGAGUGGGUUGUUUCUG (-((.......((((((((((.(((.(((.................))).)))))))))))))..((((((.....((((((((((.......)))).)))))).....))))))))).. ( -33.93) >DroYak_CAF1 81080 119 - 1 G-AAAACAAAACAUUUUGUUGUGCGUCUCACGCUUUGCGAAUAAAAGAGCUGCCAAUAAAAUGCCACAGCCAGAAACGUGUGUAUAUGAUUUUUAUACCACACGGAGUGGGUUGUUCCUG .-.........((((((((((.(((.(((.(((...))).......))).)))))))))))))..((((((.....((((((((((.......)))).)))))).....))))))..... ( -33.30) >consensus GAAAAACAAAACAUUUUGUUGUGCGUCUCACGCUUUGCAAAUAAAAGAGCUGCCAAUAAAAUGCCACAGCCAGAAACGUGUGUAUAUGAUUUUUAUACCACACGGAGUGGGUUGUUUCAG ...........((((((((((.(((.(((.................))).)))))))))))))..((((((.....((((((((((.......)))).)))))).....))))))..... (-33.01 = -32.77 + -0.24)

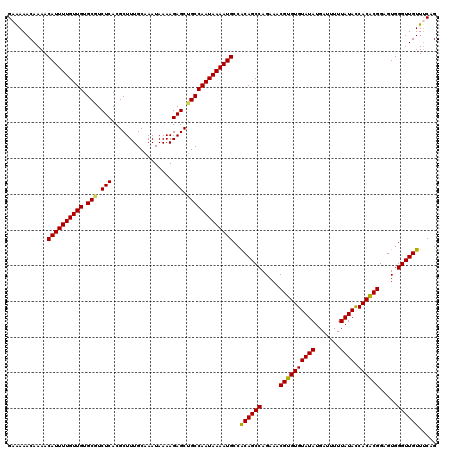
| Location | 12,960,039 – 12,960,157 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.62 |
| Mean single sequence MFE | -28.82 |
| Consensus MFE | -19.28 |
| Energy contribution | -19.40 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.67 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12960039 118 + 23771897 CACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGACGCACAACAAAAUGUUUUGUUUCUCAUGUUUUGCCUUAUGUUUAU--AUUUAGUGAAGGAAAAAA ..........(((((.(........).)))))(((((((((.(((((((((((((.(((.(((.....))).))).)))))))))))))...........--....)))))))))..... ( -33.69) >DroSec_CAF1 77271 105 + 1 CACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGAGGCACAACAAAAUGUUUUGUUUUUCAUG------------UUUAU--AUUUAGUGAAGGAA-AAA ..........(((((.(........).)))))(((((((((....((((((((((((((.(((.....))).)))))))))))------------)))..--....))))))))).-... ( -29.30) >DroSim_CAF1 82022 105 + 1 CACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGACGCACAACAAAAUGUUUUGUUUUUCAUG------------UUUAU--AUUUAGUGAAGGAA-AAA ..........(((((.(........).)))))(((((((((....((((((((((.(((.(((.....))).))).)))))))------------)))..--....))))))))).-... ( -26.50) >DroEre_CAF1 83260 116 + 1 CACGUUUCUGGCUGCGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGAUGCACAACAAAAUGUUUUGUUUU-CAUGUUUCGCUUCAUGUUUAU--AUUUAGUAAAGGAA-AAA ..........(((((.(........).)))))(((((((((.((.((((((((((.(((.(((.....))).))))))-))))))).))...........--....))))))))).-... ( -27.59) >DroYak_CAF1 81120 118 + 1 CACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUCGCAAAGCGUGAGACGCACAACAAAAUGUUUUGUUUU-CAUGUCUUGCUUCAUGUUUAUAAAUUUAGUGAAGGCA-UAG (((......((((((.(........).)))))).........((((..(((((((.(((.(((.....))).))))))-))))..))))..................)))......-... ( -27.00) >consensus CACGUUUCUGGCUGUGGCAUUUUAUUGGCAGCUCUUUUAUUUGCAAAGCGUGAGACGCACAACAAAAUGUUUUGUUUUUCAUGU_U_GC_U_AUGUUUAU__AUUUAGUGAAGGAA_AAA ..........(((((.(........).)))))(((((((((.((...))((((((.(((.(((.....))).))).))))))........................)))))))))..... (-19.28 = -19.40 + 0.12)

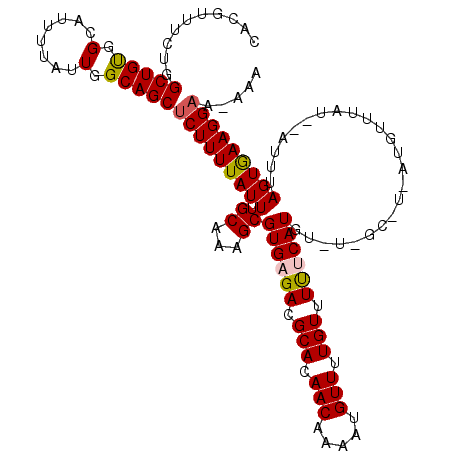
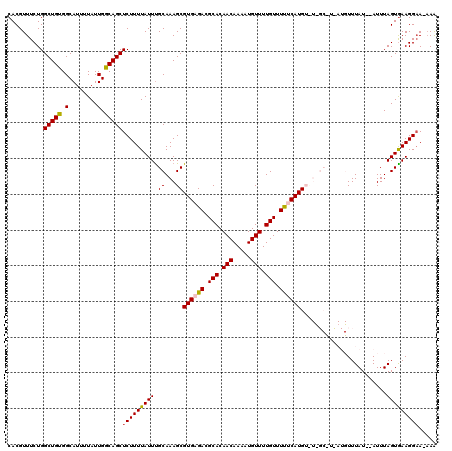
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:23:57 2006