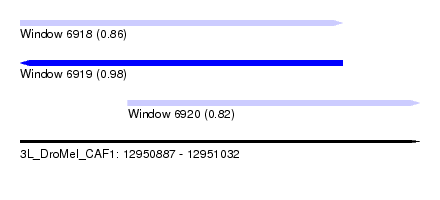
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,950,887 – 12,951,032 |
| Length | 145 |
| Max. P | 0.984788 |

| Location | 12,950,887 – 12,951,004 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.79 |
| Mean single sequence MFE | -35.64 |
| Consensus MFE | -31.04 |
| Energy contribution | -31.64 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.863054 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12950887 117 + 23771897 AGCUGUCGCCUUCAUCUU-UAAACUUUAAUGGCCAGCCAAAAGUUUUGGGCCAUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA--GCCAGAUGCUUCCGCUCUAG .(((((............-.(((((((..(((....))))))))))..(((((((.(...(((..((((......))))..)))..).))))))))))--)).(((.((....))))).. ( -37.10) >DroSec_CAF1 68111 117 + 1 AGCUGUCGCCUUCAUCUU-UAAACUUUAAUGGCCAGCCAAAAGUUUUGGGCCGUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA--ACCAGAUGCUUCCGCUCUAG ...(((.(((.....((.-.(((((((..(((....))))))))))..)).((((((((((((((.(....))))).))))))))))....))).)))--...(((.((....))))).. ( -33.20) >DroSim_CAF1 72956 118 + 1 AGCUGUCGCCUUCAUCUUUUAAACUUUAAUGGCCAGCCAAAAGUUUUGGGCCGUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA--GCCAGAUGCUUCCGCUCUAG .(((((.(((.....((...(((((((..(((....)))))))))).))(.((((((((((((((.(....))))).)))))))))).)..))).)))--)).(((.((....))))).. ( -38.80) >DroEre_CAF1 74211 119 + 1 AGCUGUCGCCUUCAUCUU-UAAACUUUAAUGGCCAGCCAAAAGUUUUGGGCCGUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACAGAGCCAGAUGCUUCCGUUCUAG (((.(((((((.......-.(((((((..(((....)))))))))).))))((((((((((((((.(....))))).))))))))))...((((......)))))))))).......... ( -34.90) >DroYak_CAF1 71984 117 + 1 AGCUGUCGCCUUCAUCUU-UAAACUUUAAUGGCCAGCCAAAAGUUUUGGGCCGUAUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA--GCCAGAUGCCUCCGUUCUGG .(((((.(((.....((.-.(((((((..(((....))))))))))..)).(((..(((((((((.(....))))).)))))..)))....))).)))--))((((.........)))). ( -34.20) >consensus AGCUGUCGCCUUCAUCUU_UAAACUUUAAUGGCCAGCCAAAAGUUUUGGGCCGUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA__GCCAGAUGCUUCCGCUCUAG .((.(((((((.........(((((((..(((....)))))))))).))))((((((((((((((.(....))))).))))))))))...((((......)))))))))........... (-31.04 = -31.64 + 0.60)

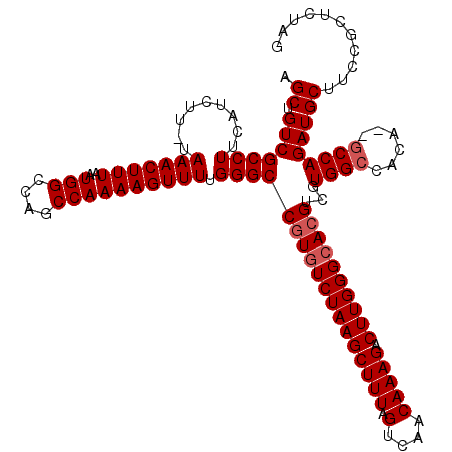
| Location | 12,950,887 – 12,951,004 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.79 |
| Mean single sequence MFE | -38.50 |
| Consensus MFE | -34.32 |
| Energy contribution | -34.40 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.99 |
| SVM RNA-class probability | 0.984788 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12950887 117 - 23771897 CUAGAGCGGAAGCAUCUGGC--UGUGGCCACGACGUGCCCAAGUCUUUGUUGACUAAAGCUUAGACAUGGCCCAAAACUUUUGGCUGGCCAUUAAAGUUUA-AAGAUGAAGGCGACAGCU .((((((....)).))))((--(((.(((.....(.(((...((((..(((......)))..))))..))).).((((((((((....)))..))))))).-........))).))))). ( -41.40) >DroSec_CAF1 68111 117 - 1 CUAGAGCGGAAGCAUCUGGU--UGUGGCCACGACGUGCCCAAGUCUUUGUUGACUAAAGCUUAGACACGGCCCAAAACUUUUGGCUGGCCAUUAAAGUUUA-AAGAUGAAGGCGACAGCU .((((((....)).))))((--(((.(((.....(.(((...((((..(((......)))..))))..))).).((((((((((....)))..))))))).-........))).))))). ( -39.00) >DroSim_CAF1 72956 118 - 1 CUAGAGCGGAAGCAUCUGGC--UGUGGCCACGACGUGCCCAAGUCUUUGUUGACUAAAGCUUAGACACGGCCCAAAACUUUUGGCUGGCCAUUAAAGUUUAAAAGAUGAAGGCGACAGCU .((((((....)).))))((--(((.(((.....(.(((...((((..(((......)))..))))..))).).((((((((((....)))..)))))))..........))).))))). ( -41.40) >DroEre_CAF1 74211 119 - 1 CUAGAACGGAAGCAUCUGGCUCUGUGGCCACGACGUGCCCAAGUCUUUGUUGACUAAAGCUUAGACACGGCCCAAAACUUUUGGCUGGCCAUUAAAGUUUA-AAGAUGAAGGCGACAGCU (((((.(....)..)))))..((((.(((.....(.(((...((((..(((......)))..))))..))).).((((((((((....)))..))))))).-........))).)))).. ( -34.80) >DroYak_CAF1 71984 117 - 1 CCAGAACGGAGGCAUCUGGC--UGUGGCCACGACGUGCCCAAGUCUUUGUUGACUAAAGCUUAGAUACGGCCCAAAACUUUUGGCUGGCCAUUAAAGUUUA-AAGAUGAAGGCGACAGCU ......((((....))))((--(((.(((.....(.(((...((((..(((......)))..))))..))).).((((((((((....)))..))))))).-........))).))))). ( -35.90) >consensus CUAGAGCGGAAGCAUCUGGC__UGUGGCCACGACGUGCCCAAGUCUUUGUUGACUAAAGCUUAGACACGGCCCAAAACUUUUGGCUGGCCAUUAAAGUUUA_AAGAUGAAGGCGACAGCU (((((((....)).)))))...(((.(((.....(.(((...((((..(((......)))..))))..))).).((((((((((....)))..)))))))..........))).)))... (-34.32 = -34.40 + 0.08)



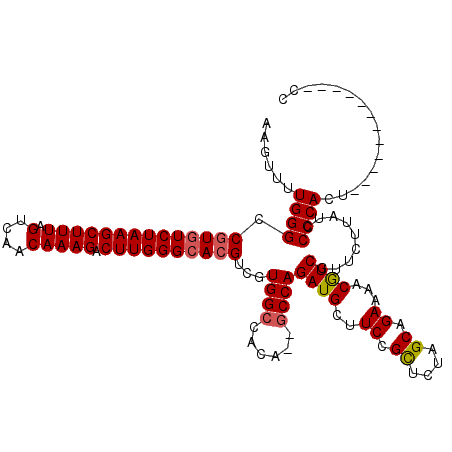
| Location | 12,950,926 – 12,951,032 |
|---|---|
| Length | 106 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.89 |
| Mean single sequence MFE | -33.64 |
| Consensus MFE | -27.16 |
| Energy contribution | -27.36 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.67 |
| SVM RNA-class probability | 0.816855 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12950926 106 + 23771897 AAGUUUUGGGCCAUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA--GCCAGAUGCUUCCGCUCUAGCAGAAAACAUCGUUCUUAUCCCCACU------------CC ..(((((.(((((((.(...(((..((((......))))..)))..).)))))))...--((.(((.((....))))).))..)))))..................------------.. ( -31.40) >DroSec_CAF1 68150 106 + 1 AAGUUUUGGGCCGUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA--ACCAGAUGCUUCCGCUCUAGCGGAAAACGUCGUUCUUAUCCCCACU------------CC ......((((.((((((((((((((.(....))))).))))))))))...(((.....--.)))((((.((((((....))))))..)))).........))))..------------.. ( -34.50) >DroSim_CAF1 72996 106 + 1 AAGUUUUGGGCCGUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA--GCCAGAUGCUUCCGCUCUAGCGGAAAACGUCGUUCUUAUCCCCACU------------CC ......((((.((((((((((((((.(....))))).))))))))))...((((....--))))((((.((((((....))))))..)))).........))))..------------.. ( -37.60) >DroEre_CAF1 74250 120 + 1 AAGUUUUGGGCCGUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACAGAGCCAGAUGCUUCCGUUCUAGCAGAAAACGUCGUCCUUAUCCCCACUCCCGCAUCCACUCC ..(((((((((((((((((((((((.(....))))).))))))))).....)))).))))))..(((((....((....)).((.....))..................)))))...... ( -31.50) >DroYak_CAF1 72023 118 + 1 AAGUUUUGGGCCGUAUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA--GCCAGAUGCCUCCGUUCUGGCAGAAAACGUCGUCCUUAUUCCCACUGCCGCACCCACUCC ......((((.(((..(((((((((.(....))))).)))))..)))..(((((....--((((((.........)))))).((.....))...............))))).)))).... ( -33.20) >consensus AAGUUUUGGGCCGUGUCUAAGCUUUAGUCAACAAAGACUUGGGCACGUCGUGGCCACA__GCCAGAUGCUUCCGCUCUAGCAGAAAACGUCGUUCUUAUCCCCACU____________CC ......((((.((((((((((((((.(....))))).))))))))))...((((......))))((((..((.((....)).))...)))).........))))................ (-27.16 = -27.36 + 0.20)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:23:16 2006