
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,912,108 – 12,912,297 |
| Length | 189 |
| Max. P | 0.995526 |

| Location | 12,912,108 – 12,912,207 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.34 |
| Mean single sequence MFE | -23.68 |
| Consensus MFE | -18.76 |
| Energy contribution | -18.68 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.607207 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12912108 99 - 23771897 UCUCGUGCAUCUAGUG--CAUGUAGGCUUAAUUGCUGCUUUUGCUUUUGAUAUGCAAAAUUAUGCACAGUUUAUCAACGCUGCGAUGCGUGCCU-------------UU------CCCAU ....((((((....((--(((((.(((......)))((....)).....))))))).....)))))).........((((......))))....-------------..------..... ( -25.50) >DroPse_CAF1 41630 98 - 1 UCUCAUGCAUCUCCUGCCCAUGCUGACUUAAUUGCUGCUUUUGCUUUUGAUAUGCAAAAUUAUGCACAGUUUAUCAACGCUGCGAUGCGUG----------------UU------ACCGA ...((((((((.(..((....(.(((...(((((.(((((((((.........))))))....))))))))..))).))).).))))))))----------------..------..... ( -22.10) >DroEre_CAF1 38115 107 - 1 UCUCGUGCAUCUAGUG--CAUGUAGGCUUAAUUGCUGCAUUUGCUUUUGAUAUGCAAAAUUAUGCACAGUUUAUCAACGCUGCGAUGCGUGCCUUUUC-----CACUUU------CCCAC ...(((((((...)))--)))).((((..(((((.(((((((((.........))))....)))))))))).....((((......))))))))....-----......------..... ( -25.90) >DroYak_CAF1 35389 107 - 1 UCUCGUGCAUCUAGUG--CAUGUAGGCUUAAUUGCUGCUUUUGCUUUUGAUAUGCAAAAUUAUGCACAGUUUAUCAACGCUGCGAUGCGUGCUUUUCC-----CACUUU------CCCAC ....((((((....((--(((((.(((......)))((....)).....))))))).....)))))).........((((......))))........-----......------..... ( -25.50) >DroAna_CAF1 59165 118 - 1 UCUCGUGCAUCUCCUA--CAUGUAGGCUUAAUUGCUGCUUUUGCUUUUGAUAUGCAAAAUUAUGCACAGUUUAUCAACGCUGCGAAGCAUGCUUUUUCCUCACCACUUCCAUCCACCCAC ....(((.....((((--....)))).......((((((((.((..((((((((((......)))).....))))))....)))))))).))...........))).............. ( -21.00) >DroPer_CAF1 37248 98 - 1 UCUCAUGCAUCUCCUGCCCAUGCUGACUUAAUUGCUGCUUUUGCUUUUGAUAUGCAAAAUUAUGCACAGUUUAUCAACGCUGCGAUGCGUG----------------UU------ACCGA ...((((((((.(..((....(.(((...(((((.(((((((((.........))))))....))))))))..))).))).).))))))))----------------..------..... ( -22.10) >consensus UCUCGUGCAUCUACUG__CAUGUAGGCUUAAUUGCUGCUUUUGCUUUUGAUAUGCAAAAUUAUGCACAGUUUAUCAACGCUGCGAUGCGUGC_U_____________UU______CCCAC ...((((((((..........((((.(......)))))....((..((((((((((......)))).....))))))....))))))))))............................. (-18.76 = -18.68 + -0.08)

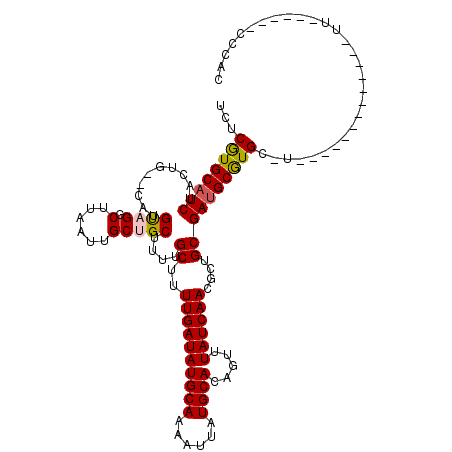
| Location | 12,912,169 – 12,912,267 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 98 |
| Reading direction | forward |
| Mean pairwise identity | 89.25 |
| Mean single sequence MFE | -26.85 |
| Consensus MFE | -22.83 |
| Energy contribution | -23.02 |
| Covariance contribution | 0.19 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.22 |
| SVM RNA-class probability | 0.639948 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12912169 98 + 23771897 AAGCAGCAAUUAAGCCUACAUGCACUAGAUGCACGAGAGCUGCAUCAUACAACAAUUGCUUUUGAUAGAUAAUUGCUGCAGAUGUGCGGGUGUAUGGG ..((((((((((.((......)).((((((((((....).))))))......(((......))).))).))))))))))....((((....))))... ( -28.00) >DroSec_CAF1 32947 94 + 1 AUGCAGCAAUUAAGUCUACAUGCACUAGAUGCACGAGAGCGGC----UACAACAAUUGCUUUUGAUAGAUAAUUGCUGCAGAUGUGUGGGUGUAUGGG .((((((((((..(((((..((((.....))))(((((((((.----........))))))))).))))))))))))))).................. ( -27.50) >DroSim_CAF1 30721 98 + 1 AUGCAGCAAUUAAGCCUACAUGCACUAGAUGCACGAGAGCGGCAUCAUACAACAAUUGCUUUUGAUAGAUAAUUGCUGCAGAUGUGUGGGUGUAUGUG .....((......)).((((((((((.(((((.((....))))))).((((.((.((((..................)))).)))))))))))))))) ( -26.57) >DroEre_CAF1 38184 90 + 1 AUGCAGCAAUUAAGCCUACAUGCACUAGAUGCACGAGAGCGGCUUCGUACAACAAUUGCUUUUGAUAGAUAAUUGCUGCAGAUGUGUGGG-------- .(((((((((((........((((.....))))(((((((((.............))))))))).....)))))))))))..........-------- ( -25.32) >DroYak_CAF1 35458 98 + 1 AAGCAGCAAUUAAGCCUACAUGCACUAGAUGCACGAGAGCGGCAUCAUACAACAAUUGCUUUUGAUAGAUAAUUGCUGCAGAUGUGUGGGUGUGUGCG .....(((.....(((((((((((...(((((.((....)))))))......((((((((......)).)))))).))))....)))))))...))). ( -28.20) >DroAna_CAF1 59245 98 + 1 AAGCAGCAAUUAAGCCUACAUGUAGGAGAUGCACGAGAGCGGCAUCAUACAACAAUUGCUUUUGAUAGAUAAUUGCUACAAAUGCAUGUGCCUGUGCA ..(((((((((...((((....)))).(((((.((....))))))).......)))))))....((((.((..(((.......)))..)).)))))). ( -25.50) >consensus AAGCAGCAAUUAAGCCUACAUGCACUAGAUGCACGAGAGCGGCAUCAUACAACAAUUGCUUUUGAUAGAUAAUUGCUGCAGAUGUGUGGGUGUAUGCG ..((((((((((........((((.....))))(((((((((.............))))))))).....))))))))))................... (-22.83 = -23.02 + 0.19)

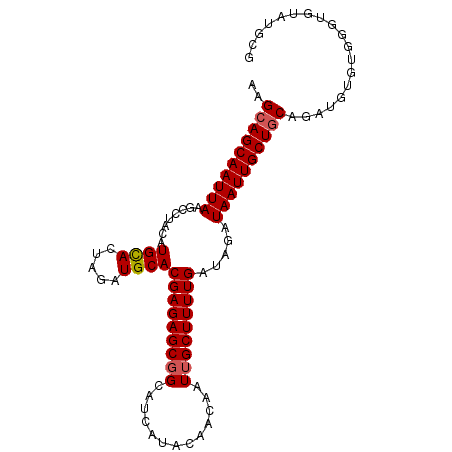

| Location | 12,912,169 – 12,912,267 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 98 |
| Reading direction | reverse |
| Mean pairwise identity | 89.25 |
| Mean single sequence MFE | -28.18 |
| Consensus MFE | -24.25 |
| Energy contribution | -25.45 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.53 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.59 |
| SVM RNA-class probability | 0.995526 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12912169 98 - 23771897 CCCAUACACCCGCACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCAGCUCUCGUGCAUCUAGUGCAUGUAGGCUUAAUUGCUGCUU ...................((((((((((..(((((..(((....)))...))))).((((..(((((((...)))))))..)))))))))))))).. ( -30.00) >DroSec_CAF1 32947 94 - 1 CCCAUACACCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUA----GCCGCUCUCGUGCAUCUAGUGCAUGUAGACUUAAUUGCUGCAU ..................(((((((((((.((((...((....))...)))----)....((((((((((...))))))).)))..))))))))))). ( -25.00) >DroSim_CAF1 30721 98 - 1 CACAUACACCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCCGCUCUCGUGCAUCUAGUGCAUGUAGGCUUAAUUGCUGCAU ..................(((((((((((..(((((..(((....)))...)))))..(((..(((((((...)))))))..))).))))))))))). ( -30.60) >DroEre_CAF1 38184 90 - 1 --------CCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUACGAAGCCGCUCUCGUGCAUCUAGUGCAUGUAGGCUUAAUUGCUGCAU --------..........(((((((((((((((.((...(((...(.(((((((........))))))).)...))).))))))..))))))))))). ( -28.90) >DroYak_CAF1 35458 98 - 1 CGCACACACCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCCGCUCUCGUGCAUCUAGUGCAUGUAGGCUUAAUUGCUGCUU ...................((((((((((..(((((..(((....)))...)))))..(((..(((((((...)))))))..))).)))))))))).. ( -29.50) >DroAna_CAF1 59245 98 - 1 UGCACAGGCACAUGCAUUUGUAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCCGCUCUCGUGCAUCUCCUACAUGUAGGCUUAAUUGCUGCUU ((((........))))...((((((((((.........(((......((((.(((((((....)).)))))...)))).....))))))))))))).. ( -25.10) >consensus CCCAUACACCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCCGCUCUCGUGCAUCUAGUGCAUGUAGGCUUAAUUGCUGCAU ...................((((((((((..(((((..(((....)))...)))))..(((..((((((.....))))))..))).)))))))))).. (-24.25 = -25.45 + 1.20)

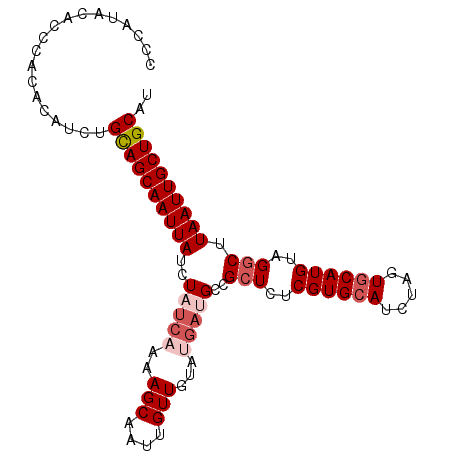

| Location | 12,912,207 – 12,912,297 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 92 |
| Reading direction | reverse |
| Mean pairwise identity | 81.18 |
| Mean single sequence MFE | -23.38 |
| Consensus MFE | -12.86 |
| Energy contribution | -13.92 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.591103 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12912207 90 - 23771897 --UAUGUAUGUGUGAAAGGGACGAGCAUUUCACCCAUACACCCGCACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCAGC --..((((((.((((((.(......).)))))).))))))...((.((((((((((((((..((......)).)))))))))).))))..)) ( -25.40) >DroSec_CAF1 32985 82 - 1 --U----AUGUGUGAAAGGGACGAGCAUUCCACCCAUACACCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUA----GCCGC --.----(((((((...(((..(......)..))).......)))))))(((((((((((..((......)).))))))))))----).... ( -18.70) >DroSim_CAF1 30759 86 - 1 --U----AUGUGUGAAAGGGACGAGCAUUCCACACAUACACCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCCGC --(----(((((((....(......)....))))))))........((((((((((((((..((......)).)))))))))).)))).... ( -20.60) >DroEre_CAF1 38222 84 - 1 AGUAUGUGUGUGUGAAAGGGACGAGCAUUCCA--------CCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUACGAAGCCGC .....(((((((((...((((......)))).--------..))))))((((((((((((..((......)).)))))))))).))...))) ( -19.20) >DroYak_CAF1 35496 91 - 1 -GUGUGUGUGUGUAAAAGGGACAAGCAUUCCACGCACACACCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCCGC -(((.((((((((....((((......))))..)))))))).))).((((((((((((((..((......)).)))))))))).)))).... ( -30.70) >DroAna_CAF1 59283 86 - 1 -GU----GGUUGUGCCAGGAAUGGCCAUCCC-UGCACAGGCACAUGCAUUUGUAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCCGC -((----(.((((((.(((.((....)).))-))))))).)))..(((((..((((((((..((......)).))))))))...)))))... ( -25.70) >consensus __U____AUGUGUGAAAGGGACGAGCAUUCCACCCAUACACCCACACAUCUGCAGCAAUUAUCUAUCAAAAGCAAUUGUUGUAUGAUGCCGC .........(((((...((((......))))...))))).......((((((((((((((..((......)).)))))))))).)))).... (-12.86 = -13.92 + 1.06)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:22:12 2006