
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,830,196 – 12,830,295 |
| Length | 99 |
| Max. P | 0.732133 |

| Location | 12,830,196 – 12,830,295 |
|---|---|
| Length | 99 |
| Sequences | 6 |
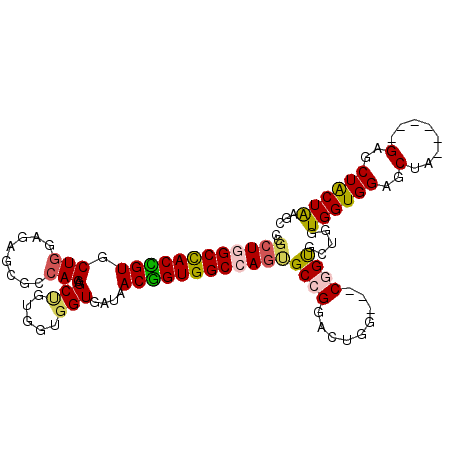
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 79.05 |
| Mean single sequence MFE | -36.82 |
| Consensus MFE | -19.88 |
| Energy contribution | -20.93 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.543268 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12830196 99 + 23771897 GCUUAGUAGCUC------UAGCUCCACCACCAGCACCG---CCAGUCCGGCACUGGCCACCGUUAUCACAACCACAGCUCUGGCGCUCGCCAGUACGGUGGCCAGCGG (((....)))..------..(((........))).(((---((.....))).(((((((((((................(((((....))))).))))))))))).)) ( -38.83) >DroVir_CAF1 92585 108 + 1 CAUUAGCAGCACGGAGAGCAGCUCCACCAGCAGCGCUGUGCCUGGACCUGCACUGGCCACAGUCAUCACCACCACGGCCCUGGCCCUCUACAGCACCGUGGCGAGCAC ........((..((.((((((.(((((((((...)))).)..)))).))))((((....))))..)).)).((((((..(((........)))..))))))...)).. ( -37.50) >DroSec_CAF1 71331 99 + 1 GCUUAGUAGCUC------UAGCUCCACCACCAGCACCG---CCAGUCCGGCACUGGCCACCGUUAUCACCACCACAGCUCUGGCGCUCGCCAGCACGGUGGCCAGCGG (((....)))..------..(((........))).(((---((.....))).(((((((((((................(((((....))))).))))))))))).)) ( -37.43) >DroSim_CAF1 71150 99 + 1 GCUUAGUAGCUC------UAGCUCCACCACCAGCACCG---CCAGUCCGGCGCUGGCCACCGUUAUCACCACCACAGCUCUGGCGCUCGCCAGCACGGUGGCCAGCGG (((....)))..------..(((........))).(((---((.....)))((((((((((((................(((((....))))).)))))))))))))) ( -41.93) >DroYak_CAF1 81131 99 + 1 CCUUAGUAGCUC------CAGUUCCACCACCAGCACCG---CCAGUCCGGCACUGGCCACCGUGAUCACCACCACAGCUCUGGCGCUCUACAGCACGGUGGCCAGCGG ........(((.------..((......)).))).(((---((.....))).((((((((((((.....(.(((......))).)........)))))))))))).)) ( -33.52) >DroMoj_CAF1 82703 105 + 1 CAUCAGCAGCACAGAGACAAGCUCCACCA---GCGCCGUGUCCGUGCCCGCCCUGGCCACAGUGAUCACCACCACGGCUCUGGCCCUGUAUAGCACUGUAGCAACCAC ........((((((.(....((....(((---(.((((((...(((.((.....)).))).(((.....))))))))).))))....))....).)))).))...... ( -31.70) >consensus CCUUAGUAGCUC______UAGCUCCACCACCAGCACCG___CCAGUCCGGCACUGGCCACCGUUAUCACCACCACAGCUCUGGCGCUCGACAGCACGGUGGCCAGCGG .....(.((((........)))).)...........................(((((((((((.............((....))(((....))))))))))))))... (-19.88 = -20.93 + 1.06)
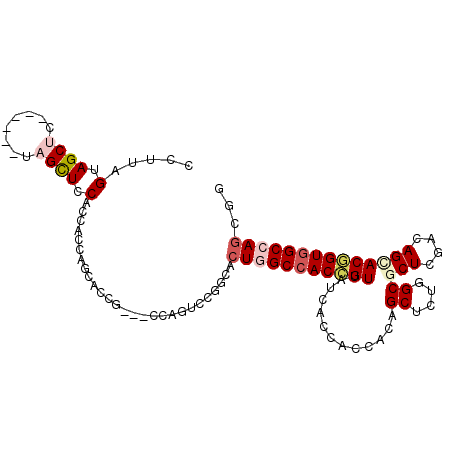
| Location | 12,830,196 – 12,830,295 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 79.05 |
| Mean single sequence MFE | -46.68 |
| Consensus MFE | -31.05 |
| Energy contribution | -30.92 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.732133 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12830196 99 - 23771897 CCGCUGGCCACCGUACUGGCGAGCGCCAGAGCUGUGGUUGUGAUAACGGUGGCCAGUGCCGGACUGG---CGGUGCUGGUGGUGGAGCUA------GAGCUACUAAGC ((((((((((((((.(((((....)))))(((....)))......)))))))))((..(((......---)))..)))))))((((((..------..))).)))... ( -47.30) >DroVir_CAF1 92585 108 - 1 GUGCUCGCCACGGUGCUGUAGAGGGCCAGGGCCGUGGUGGUGAUGACUGUGGCCAGUGCAGGUCCAGGCACAGCGCUGCUGGUGGAGCUGCUCUCCGUGCUGCUAAUG ....(((((((((((((((.....(((.((((((((.((((.((....)).)))).))).))))).)))))))))))).))))))(((.((.......)).))).... ( -49.70) >DroSec_CAF1 71331 99 - 1 CCGCUGGCCACCGUGCUGGCGAGCGCCAGAGCUGUGGUGGUGAUAACGGUGGCCAGUGCCGGACUGG---CGGUGCUGGUGGUGGAGCUA------GAGCUACUAAGC ((((((((((((((.(((((....))))).(((.....)))....)))))))))((..(((......---)))..)))))))((((((..------..))).)))... ( -47.30) >DroSim_CAF1 71150 99 - 1 CCGCUGGCCACCGUGCUGGCGAGCGCCAGAGCUGUGGUGGUGAUAACGGUGGCCAGCGCCGGACUGG---CGGUGCUGGUGGUGGAGCUA------GAGCUACUAAGC ((((((((((((((.(((((....))))).(((.....)))....)))))))))(((((((......---))))))))))))((((((..------..))).)))... ( -49.70) >DroYak_CAF1 81131 99 - 1 CCGCUGGCCACCGUGCUGUAGAGCGCCAGAGCUGUGGUGGUGAUCACGGUGGCCAGUGCCGGACUGG---CGGUGCUGGUGGUGGAACUG------GAGCUACUAAGG .((((((((((((((..((..(.(((((......))))).).))))))))))))))))((..(.(((---(....(..((......))..------).)))).)..)) ( -43.60) >DroMoj_CAF1 82703 105 - 1 GUGGUUGCUACAGUGCUAUACAGGGCCAGAGCCGUGGUGGUGAUCACUGUGGCCAGGGCGGGCACGGACACGGCGC---UGGUGGAGCUUGUCUCUGUGCUGCUGAUG ......(((((((((((((.((.(((....))).))))))....)))))))))...(((((.((((((.((((.((---(.....))))))).))))))))))).... ( -42.50) >consensus CCGCUGGCCACCGUGCUGGAGAGCGCCAGAGCUGUGGUGGUGAUAACGGUGGCCAGUGCCGGACUGG___CGGUGCUGGUGGUGGAGCUA______GAGCUACUAAGC ..((((((((((((.(((........))).(((.....)))....))))))))))))((((.........)))).....((((((..(........)..))))))... (-31.05 = -30.92 + -0.13)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:20:54 2006