
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,824,835 – 12,825,067 |
| Length | 232 |
| Max. P | 0.774021 |

| Location | 12,824,835 – 12,824,947 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 68.47 |
| Mean single sequence MFE | -34.63 |
| Consensus MFE | -21.82 |
| Energy contribution | -20.74 |
| Covariance contribution | -1.08 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.23 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.54 |
| SVM RNA-class probability | 0.774021 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
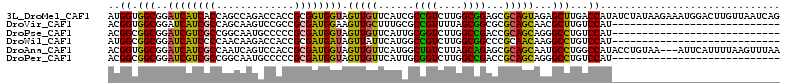
>3L_DroMel_CAF1 12824835 112 - 23771897 AUGGUGGCGGAUCAUCACCAGCCAGACCACCGCGGUGGUAGUUGUUCAUCGCCGUCUUGGCGGAGCGCAGUAGAGCUUGACCAUAUCUAUAAGAAAUGGACUUGUUAAUCAG .((((((.......))))))(((((((....(((((((.......))))))).))).)))).((((........))))..((((.((.....)).))))............. ( -32.70) >DroVir_CAF1 82647 84 - 1 ACGGUGGCGGAUCAUCGCCAGCAAGUCCGCCGCGAUGGAAGUUGCUUUGCGCCGUUUUAGCGGCGCGCAGCAACGCUUGUCCAU---------------------------- ...(((((((((...(....)...))))))))).(((((.((((((.((((((((....)))))))).)))))).....)))))---------------------------- ( -44.80) >DroPse_CAF1 93486 84 - 1 ACGGCGGCGGAUCGUCGCCGGCAAUGCCCCCGCGAUGGUAGUUGUUCAUUGCGGUCUUGGCCGACCGCAGCAGGGCCUGUCCAU---------------------------- ..(((((((...)))))))((((..((((..(((((((.......)))))((((((......)))))).)).)))).))))...---------------------------- ( -38.10) >DroWil_CAF1 95536 84 - 1 AUGGCGGCGGAUCAUCCCCAACAAGACCACCGCGAUGAUAGUUAUUCAUGGCCGUCUUGGCGGCCCGCAACAAGGCCUGUCCAU---------------------------- ..(((((.((.((...........)))).))(((((((.......))))((((((....)))))))))......))).......---------------------------- ( -27.50) >DroAna_CAF1 60593 109 - 1 ACGGUGGCGGAUCAUCGCCAAUCAGUCCACCGCGAUGGUAGUUGUUCAUGGCUGUCUUAGCAGAGCGCAGCAAUGCCUGGCCAUACCUGUAA---AUUCAUUUUAAGUUUAA .(((((((.(((........))).).))))))..(((((....((((...((((...)))).))))(((....)))...)))))........---................. ( -26.60) >DroPer_CAF1 91731 84 - 1 ACGGCGGCGGAUCGUCGCCGGCAAUGCCCCCGCGAUGGUAGUUGUUCAUUGCGGUCUUGGCCGACCGCAGCAGGGCCUGUCCAU---------------------------- ..(((((((...)))))))((((..((((..(((((((.......)))))((((((......)))))).)).)))).))))...---------------------------- ( -38.10) >consensus ACGGCGGCGGAUCAUCGCCAGCAAGACCACCGCGAUGGUAGUUGUUCAUCGCCGUCUUGGCGGACCGCAGCAAGGCCUGUCCAU____________________________ ..((.(((..((((((((.............)))))))).(((((......((((....))))...)))))...)))...)).............................. (-21.82 = -20.74 + -1.08)

| Location | 12,824,907 – 12,825,027 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.11 |
| Mean single sequence MFE | -51.22 |
| Consensus MFE | -38.07 |
| Energy contribution | -38.13 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.749554 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12824907 120 + 23771897 UACCACCGCGGUGGUCUGGCUGGUGAUGAUCCGCCACCAUAUCAGCCGGCUGGAUCCUGGAAUGAGGCACCACCGCCGGCAUAUCAGCCACCACCUGGCUACUAUGGCUGGCUGCCGCAG .......((((..(((((((.(((....))).))))(((((..((((((.(((...((((.(((.(((......)))..))).))))...))).))))))..)))))..)))..)))).. ( -53.00) >DroVir_CAF1 82691 120 + 1 UUCCAUCGCGGCGGACUUGCUGGCGAUGAUCCGCCACCGUACGUGCCAUCUGGUGCCUGGCAUGAGGCUCCUCCACCAGCUUACCAGCCACCACCCGGCUAUUACGGCUGGCUGCCGCAG .......(((((((.((.(((((((......))))).((((...(((...(((((.((((....(((((........))))).)))).)))))...)))...)))))).))))))))).. ( -52.10) >DroGri_CAF1 79243 120 + 1 UUCCAUCGUGGCGGUCUAGCUGGCGAUGAUCCGCCACCGUAUGCGCCCGCUGGUGCUUGGCAUGAGGCACCACCACCGGCUUAUCAGCCACCGCCCGGCUACUAUGGCUGGUUGCCACAG .......(((((((.((((((((((......))))).((((...(((.((((((((((......)))))))).....((((....))))...))..)))...)))))))))))))))).. ( -50.20) >DroWil_CAF1 95580 120 + 1 UAUCAUCGCGGUGGUCUUGUUGGGGAUGAUCCGCCGCCAUAUCAGCCGGCUGGCUCUUGGAAUGAGGCCCCACCACCGGCCUAUCAGCCGCCGCCGGGCUACUACGGCUGGUUGCCACAA .......(((((((((...........)).))))))).....((((((((.((((....((...(((((........))))).)))))))))((((........)))).)))))...... ( -45.90) >DroMoj_CAF1 75645 120 + 1 UUUCAUCGUGGUGGUCUUGCUGGCGAUGAUCCGCCACCAUAUGCGCCAGCCGGUGCCUGGCAUGAGGCUCCACCACCGGCUUACCAGCCACCACCAGGUUAUUAUGGCUGGUUGCCACAA .......((((((((...(((((((...((........))...))))))).((.((((......)))).))))))))(((..((((((((((....)))......))))))).))))).. ( -52.10) >DroAna_CAF1 60662 120 + 1 UACCAUCGCGGUGGACUGAUUGGCGAUGAUCCGCCACCGUACCAGCCGGCUGGAGCUUGGAAUGAGGCUCCGCCUCCGGCUUAUCAGCCACCACCCGGCUACUAUGGCUGGCUGCCCCAG .......(((((((.((((((((((......))))).......((((((.((((((((......))))))))...)))))).))))))))))..((((((.....))))))..))..... ( -54.00) >consensus UACCAUCGCGGUGGUCUUGCUGGCGAUGAUCCGCCACCAUAUCAGCCGGCUGGUGCCUGGAAUGAGGCUCCACCACCGGCUUAUCAGCCACCACCCGGCUACUAUGGCUGGCUGCCACAG .......(((((((((....(((((......)))))(((((...(((.(.(((((.((((....(((((........))))).)))).))))).).)))...)))))..))))))))).. (-38.07 = -38.13 + 0.06)


| Location | 12,824,907 – 12,825,027 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.11 |
| Mean single sequence MFE | -55.15 |
| Consensus MFE | -36.81 |
| Energy contribution | -37.57 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.34 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.661645 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12824907 120 - 23771897 CUGCGGCAGCCAGCCAUAGUAGCCAGGUGGUGGCUGAUAUGCCGGCGGUGGUGCCUCAUUCCAGGAUCCAGCCGGCUGAUAUGGUGGCGGAUCAUCACCAGCCAGACCACCGCGGUGGUA (((((((.((........)).))).(((((((((......)))(((((((((((((......)))((((.((((.(......).))))))))))))))).)))..))))))))))..... ( -54.70) >DroVir_CAF1 82691 120 - 1 CUGCGGCAGCCAGCCGUAAUAGCCGGGUGGUGGCUGGUAAGCUGGUGGAGGAGCCUCAUGCCAGGCACCAGAUGGCACGUACGGUGGCGGAUCAUCGCCAGCAAGUCCGCCGCGAUGGAA .((((((.((((.(((((...((((..(((((.((((((.(..(((......))).).)))))).)))))..))))...)))))))))((((...(....)...))))))))))...... ( -60.10) >DroGri_CAF1 79243 120 - 1 CUGUGGCAACCAGCCAUAGUAGCCGGGCGGUGGCUGAUAAGCCGGUGGUGGUGCCUCAUGCCAAGCACCAGCGGGCGCAUACGGUGGCGGAUCAUCGCCAGCUAGACCGCCACGAUGGAA (((((((.....)))))))....((((((((((((....))))(.((((((((..((.(((((.((.((....)).))......))))))).)))))))).)...)))))).))...... ( -53.30) >DroWil_CAF1 95580 120 - 1 UUGUGGCAACCAGCCGUAGUAGCCCGGCGGCGGCUGAUAGGCCGGUGGUGGGGCCUCAUUCCAAGAGCCAGCCGGCUGAUAUGGCGGCGGAUCAUCCCCAACAAGACCACCGCGAUGAUA ((((((......((((((.(((((.((((((((((....))))((.(((((....)))))))....))).)))))))).))))))((.((.....))))..........))))))..... ( -49.60) >DroMoj_CAF1 75645 120 - 1 UUGUGGCAACCAGCCAUAAUAACCUGGUGGUGGCUGGUAAGCCGGUGGUGGAGCCUCAUGCCAGGCACCGGCUGGCGCAUAUGGUGGCGGAUCAUCGCCAGCAAGACCACCACGAUGAAA (((((((.(((((((((.((......)).)))))))))..)))((((((((.((((......)))).)).(((((((...(((((.....))))))))))))...))))))))))..... ( -56.00) >DroAna_CAF1 60662 120 - 1 CUGGGGCAGCCAGCCAUAGUAGCCGGGUGGUGGCUGAUAAGCCGGAGGCGGAGCCUCAUUCCAAGCUCCAGCCGGCUGGUACGGUGGCGGAUCAUCGCCAAUCAGUCCACCGCGAUGGUA (((.(((.....))).)))..((((.((((((((((((..(((((.((((((((..........))))).)))..)))))....(((((......)))))))))).)))))))..)))). ( -57.20) >consensus CUGUGGCAACCAGCCAUAGUAGCCGGGUGGUGGCUGAUAAGCCGGUGGUGGAGCCUCAUGCCAAGCACCAGCCGGCUCAUACGGUGGCGGAUCAUCGCCAGCAAGACCACCGCGAUGGAA (((((((.....))))))).......(((((((((.....((((((...((.((..........)).)).))))))......(.(((((......))))).).)).)))))))....... (-36.81 = -37.57 + 0.75)

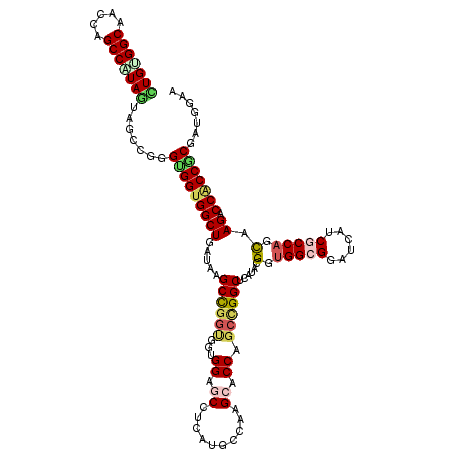
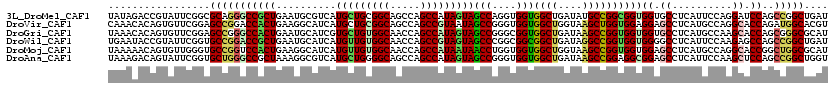
| Location | 12,824,947 – 12,825,067 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.78 |
| Mean single sequence MFE | -50.77 |
| Consensus MFE | -33.26 |
| Energy contribution | -33.55 |
| Covariance contribution | 0.29 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.544135 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12824947 120 - 23771897 UAUAGACCGUAUUCGGCGCAGGGCCGCUGAAUGCGUCAUGCUGCGGCAGCCAGCCAUAGUAGCCAGGUGGUGGCUGAUAUGCCGGCGGUGGUGCCUCAUUCCAGGAUCCAGCCGGCUGAU ....(((.((((((((((......))))))))))))).........((((((.((((.........))))))))))....((((((..(((..(((......)))..))))))))).... ( -49.60) >DroVir_CAF1 82731 120 - 1 CAAACACAGUGUUCGGAGCCGGACCACUGAAGGCAUCAUGCUGCGGCAGCCAGCCGUAAUAGCCGGGUGGUGGCUGGUAAGCUGGUGGAGGAGCCUCAUGCCAGGCACCAGAUGGCACGU ......(((((((((....)))).)))))..(((.....)))(((((.....)))))....((((..(((((.((((((.(..(((......))).).)))))).)))))..)))).... ( -51.70) >DroGri_CAF1 79283 120 - 1 UAAACACAGUGUUCGGAGCCGGGCCACUGAAUGCAUCGUGCUGUGGCAACCAGCCAUAGUAGCCGGGCGGUGGCUGAUAAGCCGGUGGUGGUGCCUCAUGCCAAGCACCAGCGGGCGCAU ......(((((((((....)))).))))).((((....(((((((((.....)))))))))(((.(.((.(((((....))))).)).((((((..........)))))).).))))))) ( -51.80) >DroWil_CAF1 95620 120 - 1 UGAAUACCGUAUUCGGUGCCGGACCGCUGAAUGCAUCAUGUUGUGGCAACCAGCCGUAGUAGCCCGGCGGCGGCUGAUAGGCCGGUGGUGGGGCCUCAUUCCAAGAGCCAGCCGGCUGAU .....((((....))))(((((((((((....(((......)))(((...(((((((.((......)).)))))))....)))))))))..(((.((.......)))))..))))).... ( -47.10) >DroMoj_CAF1 75685 120 - 1 UAAAAACAGUGUUGGGUGCCGGUCCACUGAAGGCAUCAUGUUGUGGCAACCAGCCAUAAUAACCUGGUGGUGGCUGGUAAGCCGGUGGUGGAGCCUCAUGCCAGGCACCGGCUGGCGCAU ........(((((....((((((((((((..(((....(((....)))(((((((((.((......)).)))))))))..)))..)))))))((((......)))).))))).))))).. ( -51.80) >DroAna_CAF1 60702 120 - 1 UAAAGACAGUAUUCGGUGCUGGGCCGCUAAAGGCGUCAUGCUGGGGCAGCCAGCCAUAGUAGCCGGGUGGUGGCUGAUAAGCCGGAGGCGGAGCCUCAUUCCAAGCUCCAGCCGGCUGGU ......((((.((((((..(.((((((((..(((.....(((((.....))))).......)))...)))))))).)...))))))((((((((..........))))).))).)))).. ( -52.60) >consensus UAAACACAGUAUUCGGUGCCGGGCCACUGAAGGCAUCAUGCUGUGGCAACCAGCCAUAGUAGCCGGGUGGUGGCUGAUAAGCCGGUGGUGGAGCCUCAUGCCAAGCACCAGCCGGCUCAU .................(((((((((((..........(((((((((.....)))))))))(((....)))((((....))))))))))((.((..........)).))..))))).... (-33.26 = -33.55 + 0.29)
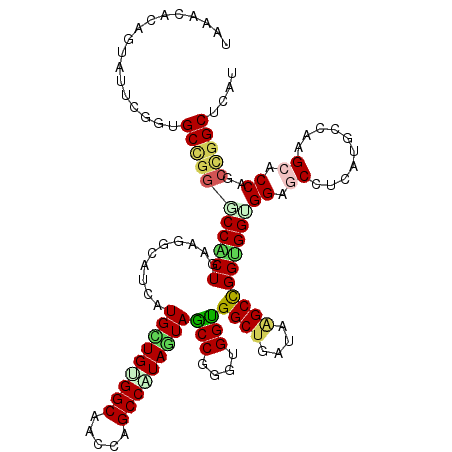

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:20:51 2006