
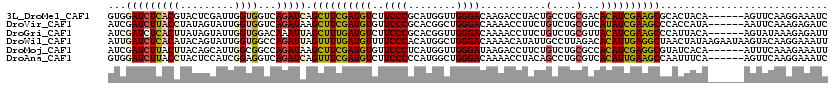
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,509,120 – 12,509,234 |
| Length | 114 |
| Max. P | 0.627016 |

| Location | 12,509,120 – 12,509,234 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.18 |
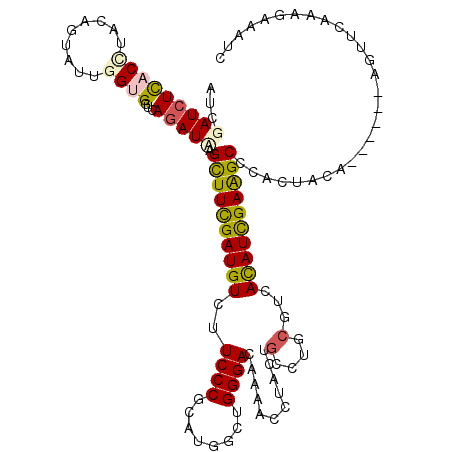
| Mean single sequence MFE | -32.43 |
| Consensus MFE | -17.46 |
| Energy contribution | -17.30 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.75 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.627016 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12509120 114 + 23771897 GUGGAUCUCACGUACUCGAUUGGUGGUCAGAUCAGCUUCGAUGUCUUCCCGCAUGGUUGGGACAAGACCUACUGCCUGCGACACAUCGAAGCGCACUACA------AGUUCAAGGAAAUC ((((((((.((.((((.....)))))).))))).((((((((((.....((((.(((((((......))))..)))))))..)))))))))).)))....------.............. ( -37.40) >DroVir_CAF1 4397 114 + 1 AUCGAUCUUACCUAUAGUAUUGGUGGUCAGAUAAGCUUCGAUGUGUUCCCGCACGGCUGGGACAAAACCUUCUGUCUGCGUCAUAUCGAAGCCCACCAUA------AAUUCAAAGAGAUC ...((((((...........((((((........(((((((((((....((((.(((.(((......)))...))))))).)))))))))))))))))..------........)))))) ( -39.30) >DroGri_CAF1 4289 114 + 1 AUCGAUCUCACUUAUAGUAUUGGUGGACAAAUUAGCUUUGAUGUCUUCCCGCACGGUUGGGACAAAACCUUCUGUCUGCGUUACAUCGAAGCCCAUUACA------AGUAUAAAGAGAUU ...((((((..(((((.(..((((((........((((((((((.....(((((((..((.......))..))))..)))..))))))))))))))))..------).))))).)))))) ( -31.50) >DroWil_CAF1 8175 120 + 1 AUUGAUCUCACAUACAGUAUUGGUGGCCAGAUUAGUUUUGAUGUUUUCCCACAUGGCUGGGACAAAACAUAUUGCCUUAGACACAUUGAGGCUAACUAUAAGAAUAAGUACAAGGAAAUU ((((((((..(((.........)))...))))))))(((((((((((((((......)))))..))))))...(((((((.....)))))))..................))))...... ( -23.40) >DroMoj_CAF1 4485 114 + 1 AUCGAUCUUACUUACAGCAUUGGCGGCCAGAUAAGCUUCGAUGUGUUCCCUCAUGGUUGGGAUAAGACCUUCUGUCUGCGCCACAUCGAGGCGUAUCACA------AUUUCAAAGAAAUU .....((((.......((....)).....((((.(((((((((((.((((........))))..((((.....))))....))))))))))).))))...------......)))).... ( -33.80) >DroAna_CAF1 4484 114 + 1 GUGGAUCUUACCUACUCCAUCGGAGGUCAGAUCAGUUUCGAUGUCUUCCCCCAUGGCUGGGACAAAACCUACAGCCUGCGUCACAUUGAAGCCAAUUUCA------AGUUCAAGGAAAUC ...(((((.((((.((.....)))))).))))).((((((((((.......((.(((((((......))..)))))))....))))))))))...((((.------.......))))... ( -29.20) >consensus AUCGAUCUCACCUACAGUAUUGGUGGUCAGAUAAGCUUCGAUGUCUUCCCGCAUGGCUGGGACAAAACCUACUGCCUGCGUCACAUCGAAGCCCACUACA______AGUUCAAAGAAAUC ...(((((((((.........))))...))))).((((((((((..((((........))))...........(....)...))))))))))............................ (-17.46 = -17.30 + -0.16)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:09 2006