
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,341,527 – 12,341,677 |
| Length | 150 |
| Max. P | 0.806509 |

| Location | 12,341,527 – 12,341,637 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.76 |
| Mean single sequence MFE | -26.07 |
| Consensus MFE | -22.56 |
| Energy contribution | -22.62 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.806509 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12341527 110 - 23771897 CUUCGUUUAUUGUUUGGUUCAUGGCCUG---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACUACUUCCAUUAUUAAUGAUGGCUC----UC-CUAGUGGAU-- ((((((.....((((((((.(((((...---))))).......(((((((....))))))).)))))))))))))).(((((.((((((....))))))...----..-..)))))..-- ( -27.10) >DroSec_CAF1 26747 110 - 1 CUUCGUUUAUUGUUUGGUUCAUGGCCUG---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCAUUUCCAUUAUUAAUGAUGGCUC----UC-CUAGUGGAC-- ((((((.....((((((((.(((((...---))))).......(((((((....))))))).)))))))))))))).(((((.((((((....))))))...----..-..)))))..-- ( -27.10) >DroSim_CAF1 27396 110 - 1 CUUCGUUUAUUGUUUGGUUCAUGGCCUG---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCAUUUCCAUUAUUAAUGAUGGCUC----UC-CUAGUGGAC-- ((((((.....((((((((.(((((...---))))).......(((((((....))))))).)))))))))))))).(((((.((((((....))))))...----..-..)))))..-- ( -27.10) >DroEre_CAF1 26582 110 - 1 AUUCAUUUAUUGUUUGGUUCAGGGCCUU---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCAUUCCCAUUAUUAAUGAUGGCUA----CC-CUAGUGGAC-- ................((((((((....---(((((((....(((((((((((((((((...........))))))).)))))..)))))...)))))))..----))-)...)))))-- ( -25.10) >DroYak_CAF1 26760 111 - 1 CUUCAUUUAUUGUUUGGCUCAUGGCCGU---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCAUUUCCAUUAUUAAUGAUGGCUC----CUUCAAGUGGAC-- ((((((.....((((((((.(((((...---))))).......(((((((....))))))).)))))))))))))).((((((((((((....))))))...----....))))))..-- ( -31.01) >DroAna_CAF1 25389 119 - 1 CUUUAUUUAUUGUUCGCCUCUUGGCCCGGUCGCUACUAAACUAUAAUGAGUCCUUUCUAUAAAGCCGAAUAUGACGACCAUUUCCAUUAUUAAUGAUUGCCCGCCUUC-CUGGUGACUAG .............(((((....(((.((((((.((.(((.....((((.(((..(((.........)))...)))...))))....))).)).))))))...)))...-..))))).... ( -19.00) >consensus CUUCAUUUAUUGUUUGGUUCAUGGCCUG___GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCAUUUCCAUUAUUAAUGAUGGCUC____UC_CUAGUGGAC__ ((((((.....((((((((.(((((......))))).......(((((((....))))))).)))))))))))))).(((((.((((((....))))))............))))).... (-22.56 = -22.62 + 0.06)

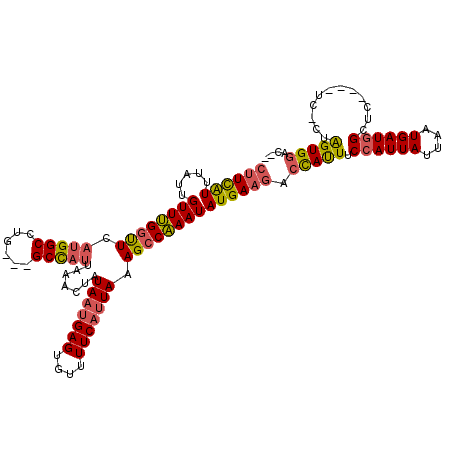

| Location | 12,341,560 – 12,341,677 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.09 |
| Mean single sequence MFE | -25.65 |
| Consensus MFE | -19.24 |
| Energy contribution | -19.47 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.38 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.653609 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12341560 117 - 23771897 UCAAGUUGUUUCAUACGAGGAUACGCGUAAUAACGCAUAGCUUCGUUUAUUGUUUGGUUCAUGGCCUG---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACUA ...((((..(((((((((((.((.((((....)))).)).)))))).....((((((((.(((((...---))))).......(((((((....))))))).))))))))))))))))). ( -32.10) >DroSec_CAF1 26780 116 - 1 CCAAGUUGU-UCAUACGAGGAUACGCGUAAUAACACAUAGCUUCGUUUAUUGUUUGGUUCAUGGCCUG---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCA ....(((((-...((((.(....).)))))))))......((((((.....((((((((.(((((...---))))).......(((((((....))))))).)))))))))))))).... ( -26.10) >DroSim_CAF1 27429 116 - 1 CCAAGUUGU-UCAUACGAGGAUACGCGUAAUAACACAUAGCUUCGUUUAUUGUUUGGUUCAUGGCCUG---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCA ....(((((-...((((.(....).)))))))))......((((((.....((((((((.(((((...---))))).......(((((((....))))))).)))))))))))))).... ( -26.10) >DroEre_CAF1 26615 101 - 1 CCAUGUUGU-UCAUAUGAGGAUACG---------------AUUCAUUUAUUGUUUGGUUCAGGGCCUU---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCA .((((((((-(...((((((((((.---------------..(((((....(((((((....(((...---))))))))))...)))))))))))))))...)))..))))))....... ( -22.20) >DroYak_CAF1 26794 101 - 1 CCAAGUGGU-UCAUAUGAGGGAACG---------------CUUCAUUUAUUGUUUGGCUCAUGGCCGU---GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCA .....((((-(....(((((.....---------------)))))(((((.((((((((.(((((...---))))).......(((((((....))))))).)))))))))))))))))) ( -27.40) >DroAna_CAF1 25428 116 - 1 ACAAGUU-U-UCAUAUGUGC--UCGCGUAAUAACAUGGAACUUUAUUUAUUGUUCGCCUCUUGGCCCGGUCGCUACUAAACUAUAAUGAGUCCUUUCUAUAAAGCCGAAUAUGACGACCA ....(((-.-((((((..((--(...((....))((((((....(((((((((..(((....)))..(((....))).....)))))))))...))))))..)))...)))))).))).. ( -20.00) >consensus CCAAGUUGU_UCAUACGAGGAUACGCGUAAUAACACAUAGCUUCAUUUAUUGUUUGGUUCAUGGCCUG___GCCAUUAAACUAUAAUGAGUGUUUUCAUUAAAGCCAAAUAUGAAGACCA ....(((...((((((((((....................)))))).....((((((((.(((((......))))).......(((((((....))))))).)))))))))))).))).. (-19.24 = -19.47 + 0.22)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:17:01 2006