
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 12,104,307 – 12,104,507 |
| Length | 200 |
| Max. P | 0.999844 |

| Location | 12,104,307 – 12,104,427 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.50 |
| Mean single sequence MFE | -41.38 |
| Consensus MFE | -39.64 |
| Energy contribution | -39.76 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.96 |
| SVM decision value | 4.23 |
| SVM RNA-class probability | 0.999844 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12104307 120 + 23771897 CACGAGCACAACUACGAGCACAUCGAGCAUGUGGAGAUGUACACGCAGCGUGUCAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUC ..(.((((((.(((((.((.......)).)))))..(((.(((((...)))))))).((.(((.((((((.((((.....))))..))))))..)))))..)))))).)........... ( -39.50) >DroSec_CAF1 5430 120 + 1 CACGAGCACAACUACGAGCAUAUCGAGCACGUGGAGAUUUACACGCAGCGCGUGAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUC .....((....(((((.((.......)).)))))...........(((((((((((((......((((((.((((.....))))..))))))..))))).)))))))).))......... ( -41.80) >DroSim_CAF1 4810 120 + 1 CACGAGCACAACUACGAGCACAUCGAGCACGUGGAGAUGUACACGCAGCGCGUGAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUGGAGACCAUUAAUGUGCUGGGCAACAUCAUC ...........(((((.((.......)).))))).(((((...(.(((((((((((((......((((((.((((.....))))..))))))..))))).)))))))).)..)))))... ( -44.10) >DroEre_CAF1 4947 120 + 1 CACGAGCACAACUACGAGCACAUCGAGCACGUGGAGAUGUACACGCAGCGCGUGAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUUGAGAGCAUCAACGUGCUGGGCAACAUCAUC ...........(((((.((.......)).))))).(((((...(.((((((((..((((.....))))...((((.....))))(((((.....))))).)))))))).)..)))))... ( -39.60) >DroYak_CAF1 4948 120 + 1 CACGAGCACAACUACGAGCACAUCGAGCACGUGGAGAUGUACACGCAGCGUGUGAUGGACUGGAUCCACAAGAACCAGAAGUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUC ...........(((((.((.......)).))))).(((((...(.(((((..((((((......((((((.((((.....))))..))))))..))))).)..))))).)..)))))... ( -41.90) >consensus CACGAGCACAACUACGAGCACAUCGAGCACGUGGAGAUGUACACGCAGCGCGUGAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUC ...........(((((.((.......)).))))).(((((...(.(((((((((((((......((((((.((((.....))))..))))))..))))).)))))))).)..)))))... (-39.64 = -39.76 + 0.12)



| Location | 12,104,307 – 12,104,427 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.50 |
| Mean single sequence MFE | -40.52 |
| Consensus MFE | -37.34 |
| Energy contribution | -38.18 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.92 |
| SVM decision value | 2.66 |
| SVM RNA-class probability | 0.996193 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12104307 120 - 23771897 GAUGAUGUUGCCCAGCACAUUGAUGGUCUCCACAUCGAACUUCUCGUUCUUGUGGAUCCAGUCCAUGACACGCUGCGUGUACAUCUCCACAUGCUCGAUGUGCUCGUAGUUGUGCUCGUG ((((.((((....))))))))..(((..((((((..((((.....)))).)))))).)))...((((((((((((((.(((((((...........))))))).))))).)))).))))) ( -43.10) >DroSec_CAF1 5430 120 - 1 GAUGAUGUUGCCCAGCACAUUGAUGGUCUCCACAUCGAACUUCUCGUUCUUGUGGAUCCAGUCCAUCACGCGCUGCGUGUAAAUCUCCACGUGCUCGAUAUGCUCGUAGUUGUGCUCGUG ((.(((.(((((((((.(..((((((.(((((((..((((.....)))).))))))....).)))))).).)))).).)))))))))((((.((.((((((....)).)))).)).)))) ( -38.30) >DroSim_CAF1 4810 120 - 1 GAUGAUGUUGCCCAGCACAUUAAUGGUCUCCACAUCGAACUUCUCGUUCUUGUGGAUCCAGUCCAUCACGCGCUGCGUGUACAUCUCCACGUGCUCGAUGUGCUCGUAGUUGUGCUCGUG ((((((((.((...))))))...(((..((((((..((((.....)))).)))))).)))...)))).(((((((((.(((((((...........))))))).))))).))))...... ( -39.20) >DroEre_CAF1 4947 120 - 1 GAUGAUGUUGCCCAGCACGUUGAUGCUCUCAACAUCGAACUUCUCGUUCUUGUGGAUCCAGUCCAUCACGCGCUGCGUGUACAUCUCCACGUGCUCGAUGUGCUCGUAGUUGUGCUCGUG ...(((((((...((((......))))..)))))))((((.....))))..((((((...))))))((((.((((((.(((((((...........))))))).))))))))))...... ( -38.60) >DroYak_CAF1 4948 120 - 1 GAUGAUGUUGCCCAGCACAUUGAUGGUCUCCACAUCGAACUUCUGGUUCUUGUGGAUCCAGUCCAUCACACGCUGCGUGUACAUCUCCACGUGCUCGAUGUGCUCGUAGUUGUGCUCGUG .............((((((.((((((.(((((((..((((.....)))).))))))....).))))))...((((((.(((((((...........))))))).)))))))))))).... ( -43.40) >consensus GAUGAUGUUGCCCAGCACAUUGAUGGUCUCCACAUCGAACUUCUCGUUCUUGUGGAUCCAGUCCAUCACGCGCUGCGUGUACAUCUCCACGUGCUCGAUGUGCUCGUAGUUGUGCUCGUG .............((((((.((((((.(((((((..((((.....)))).))))))....).))))))...((((((.(((((((...........))))))).)))))))))))).... (-37.34 = -38.18 + 0.84)


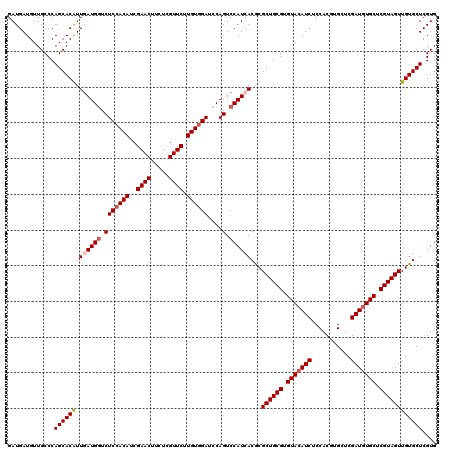
| Location | 12,104,347 – 12,104,467 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.50 |
| Mean single sequence MFE | -41.96 |
| Consensus MFE | -37.70 |
| Energy contribution | -38.14 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.58 |
| SVM RNA-class probability | 0.995496 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12104347 120 + 23771897 ACACGCAGCGUGUCAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCA ....((.((.((((...((.(((.((((((.((((.....))))..))))))..)))))..(((.(((((........))))).))).....))))))(((((........))))).)). ( -41.30) >DroSec_CAF1 5470 120 + 1 ACACGCAGCGCGUGAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCA ....((..((((((((((......((((((.((((.....))))..))))))..)))))..(((.(((((........))))).)))))))).....((((((........)))))))). ( -43.50) >DroSim_CAF1 4850 120 + 1 ACACGCAGCGCGUGAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUGGAGACCAUUAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCA ....((..((((((((((......((((((.((((.....))))..))))))..)))))..(((.(((((........))))).)))))))).....((((((........)))))))). ( -41.10) >DroEre_CAF1 4987 120 + 1 ACACGCAGCGCGUGAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUUGAGAGCAUCAACGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCA ....((..(((((..((..((((((......((((.....))))(((((((..(((((....)))))...)))))))))))))..))))))).....((((((........)))))))). ( -42.70) >DroYak_CAF1 4988 120 + 1 ACACGCAGCGUGUGAUGGACUGGAUCCACAAGAACCAGAAGUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCA ....((.((((.((((((......((((((.((((.....))))..))))))..)))))).(((.(((((........))))).)))))))......((((((........)))))))). ( -41.20) >consensus ACACGCAGCGCGUGAUGGACUGGAUCCACAAGAACGAGAAGUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCA ....((..((((((((((......((((((.((((.....))))..))))))..)))))..(((.(((((........))))).)))))))).....((((((........)))))))). (-37.70 = -38.14 + 0.44)

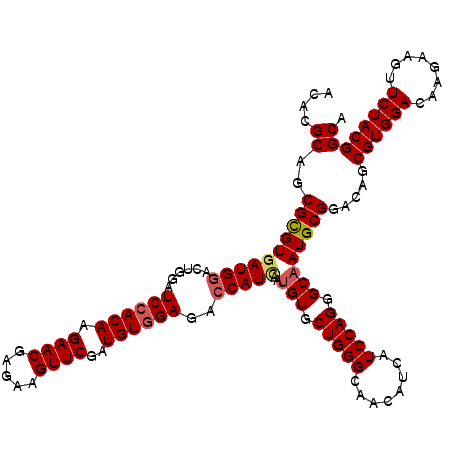

| Location | 12,104,387 – 12,104,507 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.17 |
| Mean single sequence MFE | -42.14 |
| Consensus MFE | -38.54 |
| Energy contribution | -38.76 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.893645 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 12104387 120 + 23771897 GUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCAGUCUUGACAAGCUGUACCGCUUCAUUGUGAACGAGGGCCA ((((((((.(....)))))..(((.(((((........))))).)))..(((((((((.((..(((((....(....)...))))).)).))))).))))........))))........ ( -43.10) >DroSec_CAF1 5510 120 + 1 GUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCAGUCUGGACAAGCUGUACCGCUUCAUUGUGAACGAGGGCCA ((((((((.(....)))))..(((.(((((........))))).)))..(((((((((.((..((.((....(....)...))))..)).))))).))))........))))........ ( -42.00) >DroSim_CAF1 4890 120 + 1 GUUCGAUGUGGAGACCAUUAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCAGUCUGGACAAGCUGUACCGCUUCAUUGUGAACGAGGGCCA (((((..(((((.........(((.(((((........))))).)))..(((((((((.((..((.((....(....)...))))..)).))))).)))))))))..)))))........ ( -39.40) >DroEre_CAF1 5027 120 + 1 GUUCGAUGUUGAGAGCAUCAACGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCAGUCUGGACAAGCUGUACCGCUUCAUCGUGAACGAGGGUAA ....(((((((..(((((....)))))...)))))))((((........(((((((((.((..((.((....(....)...))))..)).))))).))))....(((....))))))).. ( -43.10) >DroYak_CAF1 5028 120 + 1 GUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCAGUCUGGACAAGCUGUACCGCUUCAUCGUGAACGAGGGCCA ((((((((.(....)))))..(((.(((((........))))).)))..(((((((((.((..((.((....(....)...))))..)).))))).))))....(((....))))))).. ( -43.10) >consensus GUUCGAUGUGGAGACCAUCAAUGUGCUGGGCAACAUCAUCCAGGGCAAUGCGGACAGCGUGGACAAGAAGUUCUACGGCAGUCUGGACAAGCUGUACCGCUUCAUUGUGAACGAGGGCCA ((((((((.(....)))))..(((.(((((........))))).)))..((((((((((((((........)))))....((....))..))))).))))........))))........ (-38.54 = -38.76 + 0.22)

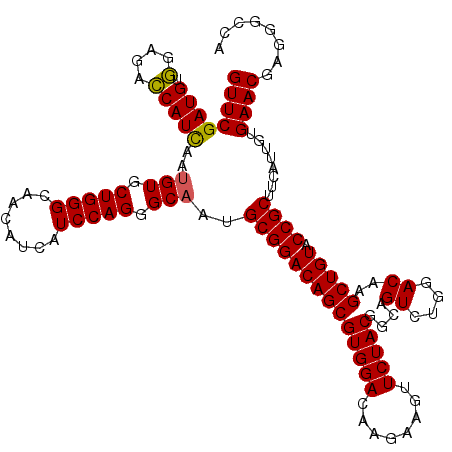
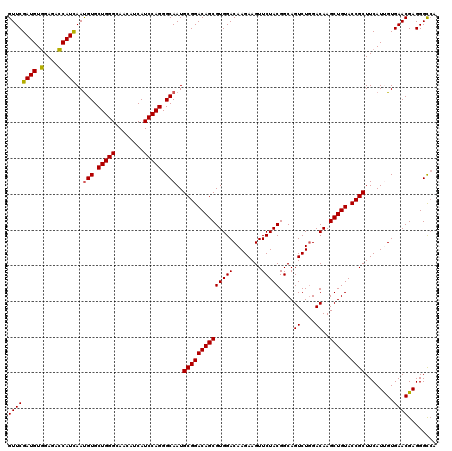
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:15:29 2006