
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 11,985,824 – 11,985,934 |
| Length | 110 |
| Max. P | 0.533522 |

| Location | 11,985,824 – 11,985,934 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 73.35 |
| Mean single sequence MFE | -45.55 |
| Consensus MFE | -22.58 |
| Energy contribution | -21.78 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.58 |
| Mean z-score | -1.62 |
| Structure conservation index | 0.50 |
| SVM decision value | -0.00 |
| SVM RNA-class probability | 0.533522 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 11985824 110 - 23771897 GGCCAGCGGAGC------CGGAAUGCUCAGUGGCAGUCAGCUGUCGGUGGCCACCAGUCAUGGGGUCAAGGAGGAUGCACUGAGUCUGUGCGGCAAGUUC---AAGGCUGGCUGCUCCA (((((((.((((------((.(..((((((((...(((..((.....(((((.(((....)))))))).))..))).))))))))...).))))....))---...)))))))...... ( -47.20) >DroVir_CAF1 4471 98 - 1 GGCCAGCGUC---------GG---GCUGAGCGGCAGUCAGCUGUCGGUGGCCACCUCCCA------CAAAGACGACGCGCUGAGCCUGUGCGGCAAAUUC---AAGAUGGGCUGCGCCC ((((((((.(---------((---(((((.(....)))))))(((.((((.......)))------)...)))..))))))).))).(((((((..(((.---..)))..))))))).. ( -43.70) >DroPse_CAF1 6718 116 - 1 GGCCAGUGGCGGCUGCUCAGGGCUGCUCAGCGGCAGCCAGCUGUCGGUGGCCACCAGCCACCUGGCCAAGGACGACACCCUGAGCCUGUCCAGCAAGUUC---AAGGUCGGAUGCUCCA (((((((((((((.(((...(((((((....)))))))))).)))(((....))).)))).))))))..(((((.((((.(((((.((.....)).))))---).))).)..)).))). ( -55.30) >DroWil_CAF1 4614 104 - 1 GGCCAGCGGC---------GGCUUACUCAGUGGCAGUCAAUUGUCGGUGGCCACCACCCAU---GCCAAGGAUGAUGUCCUGAGUUUAUCCAGUAAAUUC---AAAAUGGGUUGCGCCA (((((((((.---------(((.....((.((((((....)))))).))))).))..((((---....(((((...)))))((((((((...))))))))---...)))))))).))). ( -35.80) >DroMoj_CAF1 4621 101 - 1 GGCCAGCGCC---------GG---CCUCAGCGGCAGUCAGCUGUCGGUGGCCACGUCGCA------CAAGGACGACACGCUGAGCCUGUGCAGCAAGUUCAGCAAGAUGGGCUGCACCA ((((((((..---------((---((..(.((((((....)))))).)))))..((((..------......)))).))))).))).(((((((..(((......)))..))))))).. ( -48.70) >DroAna_CAF1 4208 107 - 1 GACCAUCGGCGG------AGGGCUGUUCAGUGGCAGUCAACUGUCGGUGGCCACCAGCCAU---GUAAAGGAGGACGCAUUGAGUUUGUGCGGCAAGUUC---AAGGCAGGCUGCUCCA (.((((((((((------..((((((......))))))..)))))))))).)..(((((.(---((...(((.(.(((((.......))))).)...)))---...))))))))..... ( -42.60) >consensus GGCCAGCGGC_________GG__UGCUCAGCGGCAGUCAGCUGUCGGUGGCCACCACCCAU___G_CAAGGACGACGCACUGAGCCUGUGCAGCAAGUUC___AAGAUGGGCUGCUCCA ...................(((..((((((((((((....)))))(((....))).......................)))))))....(((((..(((......)))..)))))))). (-22.58 = -21.78 + -0.80)

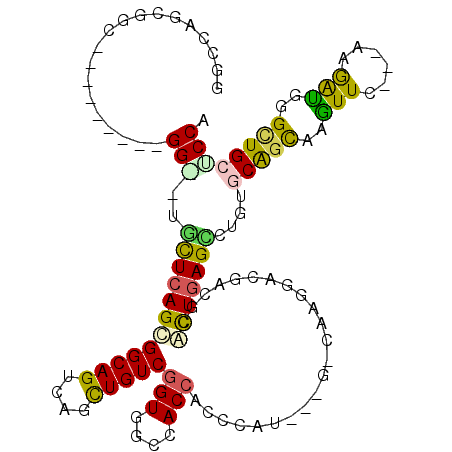
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:36 2006