
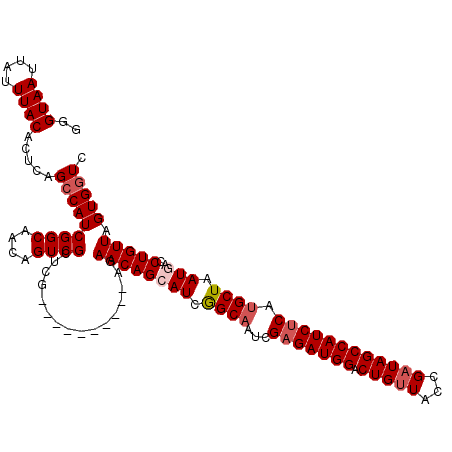
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 11,895,192 – 11,895,301 |
| Length | 109 |
| Max. P | 0.999773 |

| Location | 11,895,192 – 11,895,301 |
|---|---|
| Length | 109 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 92.48 |
| Mean single sequence MFE | -34.36 |
| Consensus MFE | -27.38 |
| Energy contribution | -28.42 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.808230 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 11895192 109 + 23771897 GGGUAAUUAUUUACACUCAGCCAUCGGCAACAGUCGCUCG----------AGAACAGAAUCGGCAAUCGAGAUGGACUGUUACCGAUAGCCAUCUCAUGCUAAUGACCUGUUAGUGUUC ............((((((((.(((..(((......((.((----------(........)))))....(((((((.(((((...)))))))))))).)))..)))..)))..))))).. ( -32.70) >DroSec_CAF1 71064 109 + 1 UGGUAAUUAUUUACACUCAGCCAUCGGCAACAGUCGCUCG----------AGAACAGCAUCGGCAAUCGAGAUGGACUGUUACCGUUAGCCAUCUCAUGCUAAUGACCUGUUAGUGGUC ..((((....)))).....(((((..(((((((((.((((----------(.....((....))..)))))...)))))))..(((((((........)))))))....))..))))). ( -34.00) >DroSim_CAF1 71649 109 + 1 UGGUAAUUAUUUACACUCAGCCAUCGGCAACAGUCGCUCG----------AGAACAGCAUCGGCAAUCGAGAUGGACUGUUACCGAUAGCCAUCUCAUGCUAAUGACCUGUUAGUGGUC ..((((....)))).((((((.((.(....).)).))).)----------))((((((((..(((...(((((((.(((((...)))))))))))).)))..)))..)))))....... ( -34.10) >DroEre_CAF1 72434 109 + 1 GGGUAAUUAUUUACACUCAGCCAUCGGCAACAGUCGCUCG----------AGAACAGGAUCGGCAAUCGAGAUGGACUGUUACCGAUAGCCAUCUCCUGCUAAUGACCUGUUAGUGGUC ..((((....)))).((((((.((.(....).)).))).)----------))((((((.((((((...(((((((.(((((...)))))))))))).))))...))))))))....... ( -35.10) >DroYak_CAF1 74021 119 + 1 GGGUAAUUAUUUACACUCAGCCAUCGGCAACAGUCGCUCGACUGCUCGAGAGAACAGCAUCAGCCAUGGAGAUGGACUGUUAGCGAUAGCCAUCGCAUGCUAAUGACCUGUUAGUGGUC ..((((....)))).....(((((..(((...(((((((((....))))).....((((((((((((....)))).)))...(((((....))))))))))..)))).)))..))))). ( -35.90) >consensus GGGUAAUUAUUUACACUCAGCCAUCGGCAACAGUCGCUCG__________AGAACAGCAUCGGCAAUCGAGAUGGACUGUUACCGAUAGCCAUCUCAUGCUAAUGACCUGUUAGUGGUC ..((((....)))).....(((((((((....))))................((((((((.((((...(((((((.(((((...)))))))))))).)))).)))..))))).))))). (-27.38 = -28.42 + 1.04)

| Location | 11,895,192 – 11,895,301 |
|---|---|
| Length | 109 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 92.48 |
| Mean single sequence MFE | -41.78 |
| Consensus MFE | -35.54 |
| Energy contribution | -36.54 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.64 |
| Structure conservation index | 0.85 |
| SVM decision value | 4.05 |
| SVM RNA-class probability | 0.999773 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 11895192 109 - 23771897 GAACACUAACAGGUCAUUAGCAUGAGAUGGCUAUCGGUAACAGUCCAUCUCGAUUGCCGAUUCUGUUCU----------CGAGCGACUGUUGCCGAUGGCUGAGUGUAAAUAAUUACCC ..(((((..((..((((....))))..))((((((((((((((((...(((((..((.......))..)----------)))).))))))))))))))))..)))))............ ( -43.00) >DroSec_CAF1 71064 109 - 1 GACCACUAACAGGUCAUUAGCAUGAGAUGGCUAACGGUAACAGUCCAUCUCGAUUGCCGAUGCUGUUCU----------CGAGCGACUGUUGCCGAUGGCUGAGUGUAAAUAAUUACCA ((((.......))))(((.((((....((((((.(((((((((((...(((((..((.......))..)----------)))).))))))))))).)))))).)))).)))........ ( -39.10) >DroSim_CAF1 71649 109 - 1 GACCACUAACAGGUCAUUAGCAUGAGAUGGCUAUCGGUAACAGUCCAUCUCGAUUGCCGAUGCUGUUCU----------CGAGCGACUGUUGCCGAUGGCUGAGUGUAAAUAAUUACCA ((((.......))))(((.((((....((((((((((((((((((...(((((..((.......))..)----------)))).)))))))))))))))))).)))).)))........ ( -43.70) >DroEre_CAF1 72434 109 - 1 GACCACUAACAGGUCAUUAGCAGGAGAUGGCUAUCGGUAACAGUCCAUCUCGAUUGCCGAUCCUGUUCU----------CGAGCGACUGUUGCCGAUGGCUGAGUGUAAAUAAUUACCC ((((.......)))).......((....(((((((((((((((((...(((((..((.......))..)----------)))).)))))))))))))))))....((((....)))))) ( -41.40) >DroYak_CAF1 74021 119 - 1 GACCACUAACAGGUCAUUAGCAUGCGAUGGCUAUCGCUAACAGUCCAUCUCCAUGGCUGAUGCUGUUCUCUCGAGCAGUCGAGCGACUGUUGCCGAUGGCUGAGUGUAAAUAAUUACCC ((((.......)))).......((((..((((((((.(((((((((((....)))(((...(((((((....)))))))..))))))))))).))))))))...))))........... ( -41.70) >consensus GACCACUAACAGGUCAUUAGCAUGAGAUGGCUAUCGGUAACAGUCCAUCUCGAUUGCCGAUGCUGUUCU__________CGAGCGACUGUUGCCGAUGGCUGAGUGUAAAUAAUUACCC ((((.......))))(((.((((....((((((((((((((((((....(((.....)))....((((............)))))))))))))))))))))).)))).)))........ (-35.54 = -36.54 + 1.00)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:14:07 2006