
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 11,449,054 – 11,449,186 |
| Length | 132 |
| Max. P | 0.970432 |

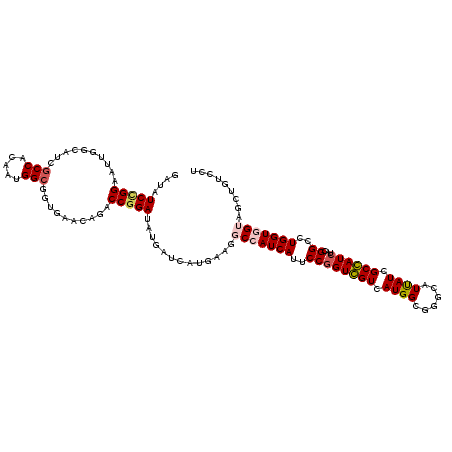
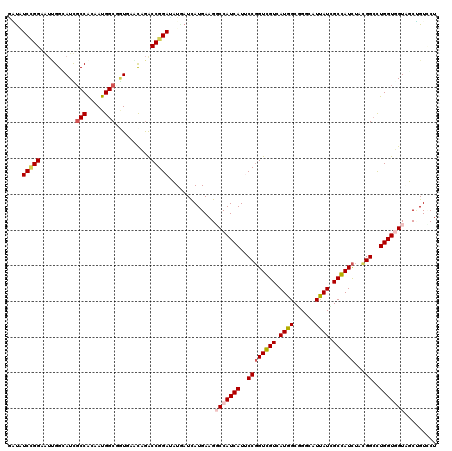
| Location | 11,449,054 – 11,449,174 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.33 |
| Mean single sequence MFE | -47.95 |
| Consensus MFE | -23.93 |
| Energy contribution | -24.35 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.50 |
| Structure conservation index | 0.50 |
| SVM decision value | 1.66 |
| SVM RNA-class probability | 0.970432 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 11449054 120 + 23771897 AGUUUCCGGUGUCGGAAUCGCCAAGAUGGCGGUCAAUCGACCGGAUAUGAUCAUGAAGGCCAUCAUUCCGGUCGUAAUGGCGGGAAUUAUUGCGAUCUACGGAUUGGUGGUAUCCGUCCU .......((...((((((((((((((((((((((....)))).(((...)))......))))))..((((((((((((((......))))))))))...)))))))))))).)))).)). ( -46.50) >DroGri_CAF1 276957 120 + 1 AAUGUCUGGCAUUGGCAUUGCCACAAUGGCUGUUGCCAGGCCGGAACUAAUUAUGAAGGCCAUCAUACCGGUGGUGAUGGCCGGCAUUAUAGCCAUCUAUGGCCUGGUUGUGGCUGUGCU ((((.....))))(((((.(((((((((((....)))((((((..............(((((((((.......)))))))))(((......))).....)))))).)))))))).))))) ( -55.20) >DroEre_CAF1 179623 120 + 1 GUUAUCCGGGGUUGGGAUCGCCACGAUGGCGGUGAACCGGCCGGAUAUGAUCAUGAAGGCCAUCAUUCCGGUCGUCAUGGCGGGGAUUAUCGCGAUCUAUGGCCUGGUGGUCUCGGUUCU .......((..((((((((((((..(((((((....))(((((((.(((((..........)))))))))))))))))(((..(((((.....)))))...)))))))))))))))..)) ( -51.30) >DroYak_CAF1 167317 120 + 1 GUUAUCCGGACUUGGCAUCGCCACCAUGGCCGUGAACCGACCGGACAUGAUCAUGAAGUCCAUCAUUCCGGUCGUCAUGGCGGGCAUCAUCGCGAUCUACGGCCUGGUGGUCUCGGUUCU .......(((((.((....(((((((.(((((((...((((((((.(((((..........)))))))))))))..((.((((......)))).)).)))))))))))))))).))))). ( -51.60) >DroMoj_CAF1 300199 120 + 1 UAUGUCAGGUAGAGGCAUUGCCGAAAUGGCCCUGAUCAAGCCGGAGCUUAUUAUGAAGGCCAUCAUACCGGUGGUCAUGGCUGGCAUUAUAGCCAUCUAUGGCUUGGUUGUGGCUGUCCU ......(((....(((...)))...((((((.(((((((((((.((...........(((((((.....)))))))(((((((......))))))))).))))))))))).))))))))) ( -44.80) >DroAna_CAF1 163809 120 + 1 GGCCUCCGGAAUCGGAAUUUCCGUAAUGGCGAUCAGCAGACCGGAUCUGAUCAUGAAGUCCAUCAUUCCUGUGGUUAUGGCGGGAAUUAUCGCCAUUUACGGCCUGGUGGUAGCUGUACU .(((..(((((.......)))))....)))((((((..........))))))....(((((((((...((((((..(((((((......)))))))))))))..))))))..)))..... ( -38.30) >consensus GAUAUCCGGAAUUGGCAUCGCCACAAUGGCGGUGAACAGACCGGAUAUGAUCAUGAAGGCCAUCAUUCCGGUCGUCAUGGCGGGCAUUAUCGCCAUCUACGGCCUGGUGGUAGCUGUCCU ....(((((..........(((.....)))..........))))).............(((((((..((((((((.((((......)))).))))))...))..)))))))......... (-23.93 = -24.35 + 0.42)

| Location | 11,449,054 – 11,449,174 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.33 |
| Mean single sequence MFE | -40.70 |
| Consensus MFE | -14.65 |
| Energy contribution | -14.84 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.36 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.739370 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 11449054 120 - 23771897 AGGACGGAUACCACCAAUCCGUAGAUCGCAAUAAUUCCCGCCAUUACGACCGGAAUGAUGGCCUUCAUGAUCAUAUCCGGUCGAUUGACCGCCAUCUUGGCGAUUCCGACACCGGAAACU .((((((((.......)))))).((((((..........))......((((((((((((.(......).))))).)))))))))))..))(((.....)))..(((((....)))))... ( -38.30) >DroGri_CAF1 276957 120 - 1 AGCACAGCCACAACCAGGCCAUAGAUGGCUAUAAUGCCGGCCAUCACCACCGGUAUGAUGGCCUUCAUAAUUAGUUCCGGCCUGGCAACAGCCAUUGUGGCAAUGCCAAUGCCAGACAUU .(((..(((((((..(((((......(((......)))((((((((((...)))..)))))))...............)))))(((....))).)))))))..))).............. ( -47.30) >DroEre_CAF1 179623 120 - 1 AGAACCGAGACCACCAGGCCAUAGAUCGCGAUAAUCCCCGCCAUGACGACCGGAAUGAUGGCCUUCAUGAUCAUAUCCGGCCGGUUCACCGCCAUCGUGGCGAUCCCAACCCCGGAUAAC ........((.((..(((((((.....(((........)))(((..(....)..))))))))))...)).)).(((((((..((((...((((.....)))).....))))))))))).. ( -36.90) >DroYak_CAF1 167317 120 - 1 AGAACCGAGACCACCAGGCCGUAGAUCGCGAUGAUGCCCGCCAUGACGACCGGAAUGAUGGACUUCAUGAUCAUGUCCGGUCGGUUCACGGCCAUGGUGGCGAUGCCAAGUCCGGAUAAC ....(((.(.((((((((((((..((((((........)))......((((((((((((.(......).))))).))))))))))..)))))).)))))))(((.....))))))..... ( -51.30) >DroMoj_CAF1 300199 120 - 1 AGGACAGCCACAACCAAGCCAUAGAUGGCUAUAAUGCCAGCCAUGACCACCGGUAUGAUGGCCUUCAUAAUAAGCUCCGGCUUGAUCAGGGCCAUUUCGGCAAUGCCUCUACCUGACAUA (((...(((.....((((((.....((((......))))(((((.(((...)))...)))))................)))))).....)))......(((...)))....)))...... ( -34.30) >DroAna_CAF1 163809 120 - 1 AGUACAGCUACCACCAGGCCGUAAAUGGCGAUAAUUCCCGCCAUAACCACAGGAAUGAUGGACUUCAUGAUCAGAUCCGGUCUGCUGAUCGCCAUUACGGAAAUUCCGAUUCCGGAGGCC ................((((....((((((........)))))).......((((((((((...(((.((((......))))...)))...))))).(((.....))))))))...)))) ( -36.10) >consensus AGAACAGACACCACCAGGCCAUAGAUCGCGAUAAUGCCCGCCAUGACCACCGGAAUGAUGGCCUUCAUGAUCAGAUCCGGCCGGUUCACCGCCAUUGUGGCAAUGCCAAUACCGGACAAC ..........((....((((.......(((........)))..........(((((((......)))).......)))))))........(((.....)))............))..... (-14.65 = -14.84 + 0.20)

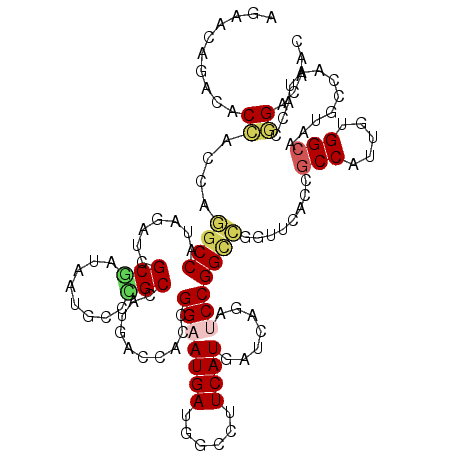

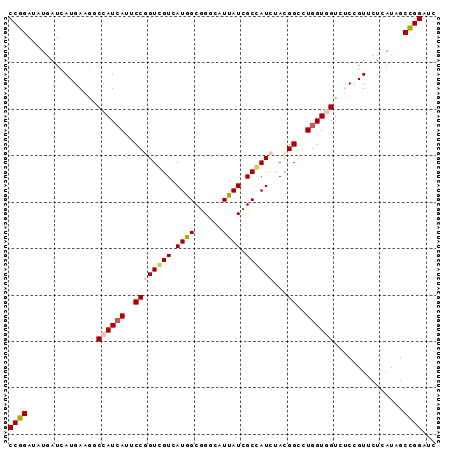
| Location | 11,449,094 – 11,449,186 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 92 |
| Reading direction | forward |
| Mean pairwise identity | 81.45 |
| Mean single sequence MFE | -32.67 |
| Consensus MFE | -18.29 |
| Energy contribution | -18.68 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.56 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.513764 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 11449094 92 + 23771897 CCGGAUAUGAUCAUGAAGGCCAUCAUUCCGGUCGUAAUGGCGGGAAUUAUUGCGAUCUACGGAUUGGUGGUAUCCGUCCUGAUAGCCGGAUC ((((.....((((.((..(((((((.((((((((((((((......))))))))))...)))).))))))).....)).))))..))))... ( -35.90) >DroEre_CAF1 179663 92 + 1 CCGGAUAUGAUCAUGAAGGCCAUCAUUCCGGUCGUCAUGGCGGGGAUUAUCGCGAUCUAUGGCCUGGUGGUCUCGGUUCUCAUAGCCGGAUC ((((.(((((..((..(((((((((....((((((.((.((((......)))).))..)))))))))))))))..))..))))).))))... ( -36.80) >DroWil_CAF1 195460 92 + 1 CCGGAUUUGAUUAUGAAAUCCAUAAUACCCGUUGUAAUGGCUGGCAUUAUAGCCAUAUAUGGUCUGGUUGUCUCCGUUCUCAUUGCUGGCUC (((((....((((((.....))))))..((((....(((((((......)))))))..)))))))))..(((..((.......))..))).. ( -21.80) >DroYak_CAF1 167357 92 + 1 CCGGACAUGAUCAUGAAGUCCAUCAUUCCGGUCGUCAUGGCGGGCAUCAUCGCGAUCUACGGCCUGGUGGUCUCGGUUCUGAUAGCCGGAUC ((((.....((((.((((.((((((....((((((.((.((((......)))).))..)))))))))))).))....))))))..))))... ( -31.80) >DroMoj_CAF1 300239 92 + 1 CCGGAGCUUAUUAUGAAGGCCAUCAUACCGGUGGUCAUGGCUGGCAUUAUAGCCAUCUAUGGCUUGGUUGUGGCUGUCCUCAUAGCCGGUUC ...(((((..((((((.((((((...((((..((((((((.((((......)))).)))))))))))).))))))....))))))..))))) ( -37.20) >DroAna_CAF1 163849 92 + 1 CCGGAUCUGAUCAUGAAGUCCAUCAUUCCUGUGGUUAUGGCGGGAAUUAUCGCCAUUUACGGCCUGGUGGUAGCUGUACUGAUUGCCGGAGC ((((....(((((...(((((((((...((((((..(((((((......)))))))))))))..))))))..)))....))))).))))... ( -32.50) >consensus CCGGAUAUGAUCAUGAAGGCCAUCAUUCCGGUCGUCAUGGCGGGCAUUAUCGCCAUCUACGGCCUGGUGGUCUCCGUUCUCAUAGCCGGAUC ((((...............((((((..((((((((.((((......)))).))))))...))..))))))...............))))... (-18.29 = -18.68 + 0.39)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:10:40 2006