
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 11,041,662 – 11,041,792 |
| Length | 130 |
| Max. P | 0.862306 |

| Location | 11,041,662 – 11,041,764 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 80.26 |
| Mean single sequence MFE | -32.25 |
| Consensus MFE | -20.79 |
| Energy contribution | -21.93 |
| Covariance contribution | 1.14 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.33 |
| Structure conservation index | 0.64 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
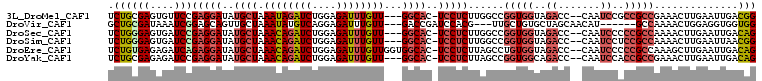
>3L_DroMel_CAF1 11041662 102 - 23771897 UCUGCGAGUGUUCCGAGGAUAUGCUAAAUAGAUCUGGAGAUUUGUU---GGCAC-UCCUCUUGGCCGGUGGUAGACC--CAAUCCGCCGCCGAAACUUGAAUUGACGG ....(((((.....(((((..((((((.((((((....))))))))---)))).-)))))(((((.(((((......--....)))))))))).)))))......... ( -35.40) >DroVir_CAF1 2639 96 - 1 GCUGCGAUAAAUCGGAGCAGUUGCUAAAUAUGUCAGGAGAUUUGUU---GACCGAUCCACG---UUGCUGUGCUAGCAACAU------GCCAAAACUGGAGGUGGUGG (((.(((....))).))).((((...((((.(((....))).))))---...))))(((((---((((((...)))))))..------.(((....)))..))))... ( -25.90) >DroSec_CAF1 2652 102 - 1 UCUGGGAGUGAUCCGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUU---GGCAC-UCCUCUUGGCCGGUGGUAGACC--CAAUCCCCCGCCAAAACUUGAAUUGACAG .((((((....)))(((((..((((((.((((((....))))))))---)))).-)))))(((((.((.((......--....)))).)))))............))) ( -35.50) >DroSim_CAF1 2636 102 - 1 UCUGGGAGUGAUCCGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUU---GGCAC-UCCUCUUGGCCGGUGGUAGACC--CAAUCCUCCGCCAAAACUUGAAUUAACGG .((((((....)))(((((..((((((.((((((....))))))))---)))).-)))))(((((.((.((......--....)).)))))))............))) ( -34.60) >DroEre_CAF1 2775 105 - 1 UCUGUGAGAGAUCAGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUUGGUGGCAC-UCCUCUUAGCCUGUGGUAGACC--CAAUCCCCCGCCAAAGCUUGAAUUGACAG .((((.((.....((((((..(((((((((((((....))))))))..))))).-)))))).(((..((((..((..--...))..))))....)))....)).)))) ( -29.60) >DroYak_CAF1 2708 102 - 1 UCUGCGAGAGAUCCGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUU---GGCAC-UCCUCUUAGCCGGUGGCAGACC--CAAUCCACCGCCGAAACUUGAAUUGACAG ....((((......(((((..((((((.((((((....))))))))---)))).-))))).....((((((..((..--...))..))))))...))))......... ( -32.50) >consensus UCUGCGAGAGAUCCGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUU___GGCAC_UCCUCUUGGCCGGUGGUAGACC__CAAUCCCCCGCCAAAACUUGAAUUGACAG .((((((....)))(((((..((((.((((((((....))))))))...))))..)))))......(((((..((.......))..)))))..............))) (-20.79 = -21.93 + 1.14)

| Location | 11,041,691 – 11,041,792 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | forward |
| Mean pairwise identity | 80.51 |
| Mean single sequence MFE | -29.65 |
| Consensus MFE | -18.99 |
| Energy contribution | -20.66 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.19 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.83 |
| SVM RNA-class probability | 0.862306 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
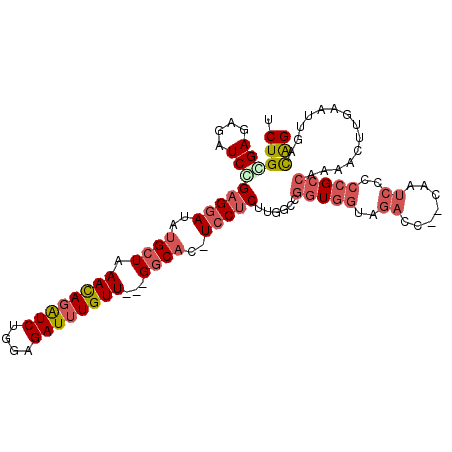
>3L_DroMel_CAF1 11041691 101 + 23771897 -GGUCUACCACCGGCCAAGAGGA-GUGCC---AACAAAUCUCCAGAUCUAUUUAGCAUAUCCUCGGAACACUCGCAGAUUCCGCUGGCGCAACCAAGACCCUCA---UC -(((((..(.(((((...(((((-((((.---.....(((....))).......)))).)))))(((((.......).))))))))).)......)))))....---.. ( -26.92) >DroVir_CAF1 2663 103 + 1 UGCUAGCACAGCAA---CGUGGAUCGGUC---AACAAAUCUCCUGACAUAUUUAGCAACUGCUCCGAUUUAUCGCAGCUCACGCUGGCACAGCCUGUGCCUUCAGUUUC (((((((..(((..---((((((((((((---(..........))))......(((....))).))))))).))..)))...)))))))....(((......))).... ( -27.10) >DroSec_CAF1 2681 101 + 1 -GGUCUACCACCGGCCAAGAGGA-GUGCC---AACAAAUCUCCAGAUCUGUUUAGCAUAUCCUCGGAUCACUCCCAGAUUCCGCUGGCGCAACCGCGACCUUCG---UC -((((.....(((((...(((((-((((.---((((.(((....))).))))..)))).)))))(((((.......))))).))))).((....))))))....---.. ( -33.50) >DroSim_CAF1 2665 101 + 1 -GGUCUACCACCGGCCAAGAGGA-GUGCC---AACAAAUCUCCAGAUCUGUUUAGCAUAUCCUCGGAUCACUCCCAGAUUCCGCUGGCGCAACCGCGACCCUCG---UC -((((.....(((((...(((((-((((.---((((.(((....))).))))..)))).)))))(((((.......))))).))))).((....))))))....---.. ( -33.10) >DroEre_CAF1 2804 104 + 1 -GGUCUACCACAGGCUAAGAGGA-GUGCCACCAACAAAUCUCCAGAUCUGUUUAGCAUAUCCUCUGAUCUCUCACAGAUUCCUCUGGCGCAGCCACGACCCUCA---UC -((((.......((((.((((((-((((....((((.(((....))).))))..)))).)))))).........((((....))))....))))..))))....---.. ( -27.50) >DroYak_CAF1 2737 101 + 1 -GGUCUGCCACCGGCUAAGAGGA-GUGCC---AACAAAUCUCCAGAUCUGUUUAGCAUAUCCUCGGAUCUCUCGCAGAUUCCUCUGGCACAGCCACGACCCUCA---UC -(((.(((((........(((((-((((.---((((.(((....))).))))..)))).)))))((((((.....))))))...)))))..)))..........---.. ( -29.80) >consensus _GGUCUACCACCGGCCAAGAGGA_GUGCC___AACAAAUCUCCAGAUCUGUUUAGCAUAUCCUCGGAUCACUCGCAGAUUCCGCUGGCGCAACCACGACCCUCA___UC .((((........((((.(((((.((((....((((.(((....))).))))..)))).)))))(((((.......)))))...))))........))))......... (-18.99 = -20.66 + 1.67)


| Location | 11,041,691 – 11,041,792 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 80.51 |
| Mean single sequence MFE | -38.15 |
| Consensus MFE | -25.47 |
| Energy contribution | -26.25 |
| Covariance contribution | 0.78 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.79 |
| SVM RNA-class probability | 0.851061 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
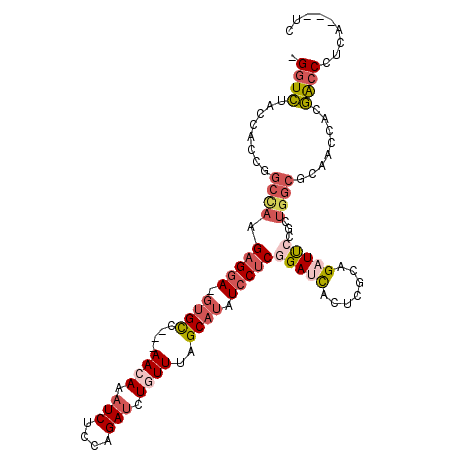
>3L_DroMel_CAF1 11041691 101 - 23771897 GA---UGAGGGUCUUGGUUGCGCCAGCGGAAUCUGCGAGUGUUCCGAGGAUAUGCUAAAUAGAUCUGGAGAUUUGUU---GGCAC-UCCUCUUGGCCGGUGGUAGACC- ..---....((((((.(..(.(((((.(((((........)))))(((((..((((((.((((((....))))))))---)))).-)))))))))))..).).)))))- ( -35.60) >DroVir_CAF1 2663 103 - 1 GAAACUGAAGGCACAGGCUGUGCCAGCGUGAGCUGCGAUAAAUCGGAGCAGUUGCUAAAUAUGUCAGGAGAUUUGUU---GACCGAUCCACG---UUGCUGUGCUAGCA ......(..(((((((..(((((((((....)))).)....((((((((....))).((((.(((....))).))))---..))))).))))---...)))))))..). ( -32.00) >DroSec_CAF1 2681 101 - 1 GA---CGAAGGUCGCGGUUGCGCCAGCGGAAUCUGGGAGUGAUCCGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUU---GGCAC-UCCUCUUGGCCGGUGGUAGACC- ((---(....)))..((((..((((.(((......(((....)))(((((..((((((.((((((....))))))))---)))).-)))))....))).)))).))))- ( -44.70) >DroSim_CAF1 2665 101 - 1 GA---CGAGGGUCGCGGUUGCGCCAGCGGAAUCUGGGAGUGAUCCGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUU---GGCAC-UCCUCUUGGCCGGUGGUAGACC- ((---(....)))..((((..((((.(((......(((....)))(((((..((((((.((((((....))))))))---)))).-)))))....))).)))).))))- ( -42.20) >DroEre_CAF1 2804 104 - 1 GA---UGAGGGUCGUGGCUGCGCCAGAGGAAUCUGUGAGAGAUCAGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUUGGUGGCAC-UCCUCUUAGCCUGUGGUAGACC- ..---....((((((....))((((.(((.((((.....)))).((((((..(((((((((((((....))))))))..))))).-))))))...))).)))).))))- ( -36.00) >DroYak_CAF1 2737 101 - 1 GA---UGAGGGUCGUGGCUGUGCCAGAGGAAUCUGCGAGAGAUCCGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUU---GGCAC-UCCUCUUAGCCGGUGGCAGACC- ..---....((((((.((((.((....(((.(((.....))))))(((((..((((((.((((((....))))))))---)))).-)))))...)))))).)).))))- ( -38.40) >consensus GA___UGAGGGUCGUGGCUGCGCCAGCGGAAUCUGCGAGAGAUCCGAGGAUAUGCUAAACAGAUCUGGAGAUUUGUU___GGCAC_UCCUCUUGGCCGGUGGUAGACC_ .........((((........((((.(((......(((....)))(((((..((((.((((((((....))))))))...))))..)))))....))).)))).)))). (-25.47 = -26.25 + 0.78)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:07:38 2006