
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 11,000,107 – 11,000,296 |
| Length | 189 |
| Max. P | 0.549262 |

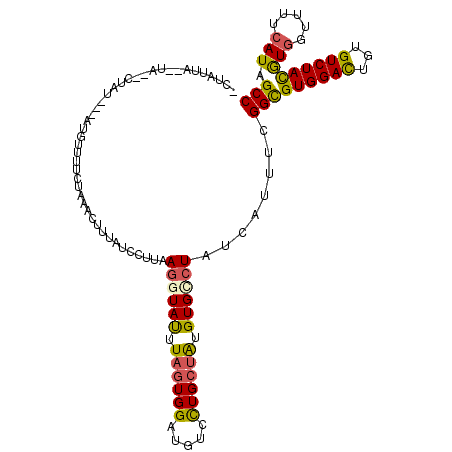
| Location | 11,000,107 – 11,000,216 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 79.67 |
| Mean single sequence MFE | -23.66 |
| Consensus MFE | -14.19 |
| Energy contribution | -13.75 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.60 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.523074 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 11000107 109 + 23771897 -CUAUUAUAUA--CUAU---AUGUUUUCUAAACUUUAUCCUUAAGGUAUUUAGUGGAUAUCCUGCUAUGUGCCUAUCAUUUUGGUGUGGAUUGUGUCUAUGUGGUUUUCAUAGCC -((((.....(--((((---(((...((((.(((.........((((((.(((..(.....)..))).))))))........))).)))).......))))))))....)))).. ( -22.03) >DroSec_CAF1 132064 110 + 1 -CUAUUA--AA--CUAAACUAAUUAGUCUAAACUUUAUUCUUAAGGUAUUUAGUGGAUGUCCUGCUAUGUGUCUAUCAUUUUGGCGUGGACUGUGUCUAUGUGGUUUUCAUUGCC -...(((--.(--((((.....))))).)))((((((....)))))).....(((((((((((((((.(((.....)))..))))).))))...))))))..(((.......))) ( -22.00) >DroSim_CAF1 130778 107 + 1 -CUCCUA--UA--CUAU---AUGUUUUCUAAACUUUAUCCUUAAGGUAUUUAGUGGAUGUCCUGCUAUGUGUCUAUCAUUUUGGCGUGGACUGUGUCUAUGUGGUUUUCAUUGCC -......--.(--((((---(((((..((((((((((....))))))..))))..)))(((((((((.(((.....)))..))))).)))).......))))))).......... ( -22.50) >DroEre_CAF1 134133 107 + 1 -UUUUUA--UA--UUAU---AUGUUUUCUAAACUUUAUCCUUAAGGUAUUUAGUGGAUGUAUUGCUAUGUGCCUAUCAUUUCGGCGUGGAUUGUGUCUACGUGGUUUUCAUAGCC -......--..--..((---(((((..((((((((((....))))))..))))..))))))).((((((.(((..........((((((((...)))))))))))...)))))). ( -26.00) >DroYak_CAF1 136533 109 + 1 -UUAUUA--CAUUUUAU---AUGUUACCUAAACUAUAUCCCUAAGUUAUUUAGUGGAUGUCCUGCUGUGUGCCUAUCACUUCGGUGUGGACUGCGUCUACGUGGUUUUCAUAGCC -.....(--(((.....---))))..........(((((((((((...))))).))))))...((((((.(((....((....))((((((...))))))..)))...)))))). ( -26.60) >DroPer_CAF1 129652 108 + 1 CUCACUA--UAUUCCAU---CUG--UAUUUAACCUGAUUCUUCAGUUACUUCGUGGACAUACUGAUGUGUGCGUAUCAUUUCGGCGUGGACUGUGUCUACAUUGUGUUUAUAGCC ....(((--((..(((.---...--........((((....)))).......(((((((((.....((.(((((.........))))).)))))))))))..)).)..))))).. ( -22.80) >consensus _CUAUUA__UA__CUAU___AUGUUUUCUAAACUUUAUCCUUAAGGUAUUUAGUGGAUGUCCUGCUAUGUGCCUAUCAUUUCGGCGUGGACUGUGUCUACGUGGUUUUCAUAGCC ...........................................((((((.((((((.....)))))).))))))........(((((((((...))))))(((.....))).))) (-14.19 = -13.75 + -0.44)

| Location | 11,000,176 – 11,000,296 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.39 |
| Mean single sequence MFE | -36.38 |
| Consensus MFE | -26.74 |
| Energy contribution | -27.22 |
| Covariance contribution | 0.48 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.549262 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 11000176 120 + 23771897 UCAUUUUGGUGUGGAUUGUGUCUAUGUGGUUUUCAUAGCCAAAAGUUUAAAGCAUCUGGGGGAUAUGUAUUUGUGGGUCUGGGAUGAGAGGCUCUAUAUGGCAUUGAUAGCCUCUCCAUU ......(((.((((((...))))))(.((((.(((..((((..(((((....(((((.(((..(((......)))..))).)))))..))))).....))))..))).)))).).))).. ( -29.80) >DroPse_CAF1 140588 120 + 1 UCAUUUCGGCGUGGACUGUGUCUACAUUGUGUUUAUAGCCAAGAACCUAAAAUUCUUGGGGGAUCUGUAUCUUUAUCCCAUGGAUCUGCGUCUGUAUAUGGCCCUGCUGAUCCUCCCACU ..........((((((...))))))...(((.......(((((((.......)))))))(((((..........)))))..(((((.(((((.......)))...)).)))))...))). ( -30.60) >DroSec_CAF1 132134 120 + 1 UCAUUUUGGCGUGGACUGUGUCUAUGUGGUUUUCAUUGCCAAGAGUUUAAAGCAUCUGGGGGAUAUGUAUUUGUGGGUAUGGGAUGAGAGGCUCUACAUGGCAUUGAUAGCCUCUCCACU ........((((((((...))))))))((((.(((.((((((((((((....(((((.(....(((......)))....).)))))..)))))))...))))).))).))))........ ( -37.00) >DroSim_CAF1 130845 120 + 1 UCAUUUUGGCGUGGACUGUGUCUAUGUGGUUUUCAUUGCCAAGAGUUUAAAGCAUCUGGGGGAUAUGUAUUUGUGGGUCUGGGAUGAGAGGCUCUAUAUGGCAUUGAUAGCCUCUCCACU ........((((((((...))))))))((((.(((.((((((((((((....(((((.(((..(((......)))..))).)))))..)))))))...))))).))).))))........ ( -41.30) >DroEre_CAF1 134200 120 + 1 UCAUUUCGGCGUGGAUUGUGUCUACGUGGUUUUCAUAGCCAAGAGUUUGAAGCAUCUGGGGGAUAUGUAUUUAUGGGCCUGGGAUGGCAGGCUUUACAUGGCCUUGAUCGCUUCGCCACU .......(((((((((...))))))(.(((..(((..((((((((((((...(((((.(((..((((....))))..))).))))).))))))))...))))..)))..))).))))... ( -42.80) >DroYak_CAF1 136602 120 + 1 UCACUUCGGUGUGGACUGCGUCUACGUGGUUUUCAUAGCCAAGAGUUUGAAGCAUCUGGGGGAUAUGUACUUGUGGUUCUGGGAUGAGAGGCUUUACAUGGCCUUGAUAGCUUCGCCGCU ......((((((((((...)))))).(((((.....))))).......(((((((((.((((.(((......))).)))).))))..((((((......))))))....))))))))).. ( -36.80) >consensus UCAUUUCGGCGUGGACUGUGUCUACGUGGUUUUCAUAGCCAAGAGUUUAAAGCAUCUGGGGGAUAUGUAUUUGUGGGUCUGGGAUGAGAGGCUCUACAUGGCAUUGAUAGCCUCUCCACU .......((.((((((...))))))(.((((.(((..(((((((((((....(((((.(((..((((....))))..))).)))))..)))))))...))))..))).)))).).))... (-26.74 = -27.22 + 0.48)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:07:14 2006