
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
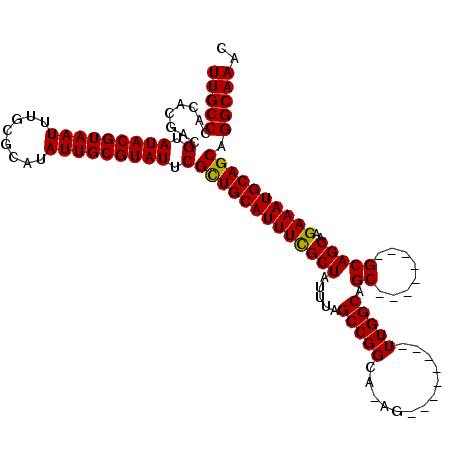
| Location | 10,702,406 – 10,702,509 |
| Length | 103 |
| Max. P | 0.999965 |

| Location | 10,702,406 – 10,702,509 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.99 |
| Mean single sequence MFE | -39.52 |
| Consensus MFE | -37.21 |
| Energy contribution | -36.85 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.50 |
| Structure conservation index | 0.94 |
| SVM decision value | 4.97 |
| SVM RNA-class probability | 0.999965 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 10702406 103 + 23771897 UUGCCCACACGACGUAUACGUAAUUUGCGCAUAUUGCGUAUUCGCUGCAUUUCGCUAUUUAGCCGGCA-AG--------UUGGCAGC--------GCAGCAAGAAAUGCAGCAGGCAAAC (((((........(.(((((((((........))))))))).)(((((((((((((.....(((((..-..--------)))))...--------..)))..)))))))))).))))).. ( -39.40) >DroSec_CAF1 36790 103 + 1 UUGCCCACACGACGUAUACGUAAUUUGCGCAUAUUGCGUAUUCGCUGCAUUUCGCUAUUUAGCCGGCA-AG--------UUGGCAGC--------GCAGCAAGAAAUGCAGCAGGCAAAC (((((........(.(((((((((........))))))))).)(((((((((((((.....(((((..-..--------)))))...--------..)))..)))))))))).))))).. ( -39.40) >DroSim_CAF1 38554 103 + 1 UUGCCCACACGACGUAUACGUAAUUUGCGCAUAUUGCGUAUUCGCUGCAUUUCGCUAUUUAGCCGGCA-AG--------UUGGCAGC--------GCAGCAAGAAAUGCAGCAGGCAAAC (((((........(.(((((((((........))))))))).)(((((((((((((.....(((((..-..--------)))))...--------..)))..)))))))))).))))).. ( -39.40) >DroEre_CAF1 40217 103 + 1 UUGCCCACACGACGUAUACGUAAUUUGCGCAUAUUGCGUAUUCGUUGCAUUUCGCUAUUUAGCCGGCA-AG--------UUGGCAGC--------GCAGCAAGAAAUGCAGCAGGCAAAC (((((........(.(((((((((........))))))))).)(((((((((((((.....(((((..-..--------)))))...--------..)))..)))))))))).))))).. ( -37.00) >DroYak_CAF1 49801 111 + 1 UUGCCCACACGACGUAUACGUAAUUUGCGCAUAUUGCGUAUUCGCUGCAUUUCGCUAUUUAGCCGGCA-AGUUGGCAAGUUGGCAGC--------GCAGCAAGAAAUGCAGCAGGCAAAC (((((........(.(((((((((........))))))))).)((((((((((((((.((.(((((..-..))))).)).)))).((--------...))..)))))))))).))))).. ( -43.10) >DroAna_CAF1 38308 112 + 1 UUGCCCACACGACGUAUACGUAAUUUGCGCAUAUUGCGUAUUCGUUGCAUUUUGCUAUUUAGCCGGCAAAG--------UUGGCAGCAGGGCACAGCAGCAAGAAAUGCAGCAGGCAAAC (((((........(.(((((((((........))))))))).)(((((((((((((.....((((((...)--------))))).((........))))))..))))))))).))))).. ( -38.80) >consensus UUGCCCACACGACGUAUACGUAAUUUGCGCAUAUUGCGUAUUCGCUGCAUUUCGCUAUUUAGCCGGCA_AG________UUGGCAGC________GCAGCAAGAAAUGCAGCAGGCAAAC (((((........(.(((((((((........))))))))).)(((((((((((((.....(((((.............))))).((........)))))..)))))))))).))))).. (-37.21 = -36.85 + -0.36)


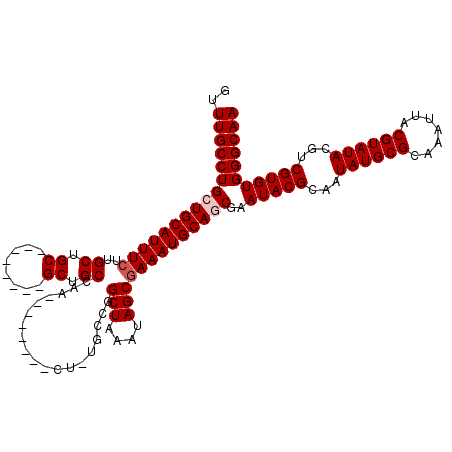
| Location | 10,702,406 – 10,702,509 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.99 |
| Mean single sequence MFE | -37.55 |
| Consensus MFE | -33.57 |
| Energy contribution | -34.07 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.84 |
| Structure conservation index | 0.89 |
| SVM decision value | 3.22 |
| SVM RNA-class probability | 0.998779 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 10702406 103 - 23771897 GUUUGCCUGCUGCAUUUCUUGCUGC--------GCUGCCAA--------CU-UGCCGGCUAAAUAGCGAAAUGCAGCGAAUACGCAAUAUGCGCAAAUUACGUAUACGUCGUGUGGGCAA ..((((((((((((((((..((((.--------...(((..--------..-....)))....))))))))))))))..(((((...((((((.......))))))...))))))))))) ( -39.70) >DroSec_CAF1 36790 103 - 1 GUUUGCCUGCUGCAUUUCUUGCUGC--------GCUGCCAA--------CU-UGCCGGCUAAAUAGCGAAAUGCAGCGAAUACGCAAUAUGCGCAAAUUACGUAUACGUCGUGUGGGCAA ..((((((((((((((((..((((.--------...(((..--------..-....)))....))))))))))))))..(((((...((((((.......))))))...))))))))))) ( -39.70) >DroSim_CAF1 38554 103 - 1 GUUUGCCUGCUGCAUUUCUUGCUGC--------GCUGCCAA--------CU-UGCCGGCUAAAUAGCGAAAUGCAGCGAAUACGCAAUAUGCGCAAAUUACGUAUACGUCGUGUGGGCAA ..((((((((((((((((..((((.--------...(((..--------..-....)))....))))))))))))))..(((((...((((((.......))))))...))))))))))) ( -39.70) >DroEre_CAF1 40217 103 - 1 GUUUGCCUGCUGCAUUUCUUGCUGC--------GCUGCCAA--------CU-UGCCGGCUAAAUAGCGAAAUGCAACGAAUACGCAAUAUGCGCAAAUUACGUAUACGUCGUGUGGGCAA ..(((((..(((((((((..((((.--------...(((..--------..-....)))....))))))))))))(((.(((((.(((........))).))))).)))...)..))))) ( -33.90) >DroYak_CAF1 49801 111 - 1 GUUUGCCUGCUGCAUUUCUUGCUGC--------GCUGCCAACUUGCCAACU-UGCCGGCUAAAUAGCGAAAUGCAGCGAAUACGCAAUAUGCGCAAAUUACGUAUACGUCGUGUGGGCAA ..((((((((((((((((..((((.--------((((.(((.........)-)).))))....))))))))))))))..(((((...((((((.......))))))...))))))))))) ( -38.70) >DroAna_CAF1 38308 112 - 1 GUUUGCCUGCUGCAUUUCUUGCUGCUGUGCCCUGCUGCCAA--------CUUUGCCGGCUAAAUAGCAAAAUGCAACGAAUACGCAAUAUGCGCAAAUUACGUAUACGUCGUGUGGGCAA ..(((((..((((((((.....((((((.....((((.((.--------...)).))))...)))))))))))))(((.(((((.(((........))).))))).)))...)..))))) ( -33.60) >consensus GUUUGCCUGCUGCAUUUCUUGCUGC________GCUGCCAA________CU_UGCCGGCUAAAUAGCGAAAUGCAGCGAAUACGCAAUAUGCGCAAAUUACGUAUACGUCGUGUGGGCAA ..((((((((((((((((..((.((........)).))...................(((....)))))))))))))..(((((...((((((.......))))))...))))))))))) (-33.57 = -34.07 + 0.50)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:03:10 2006