
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 10,293,723 – 10,293,869 |
| Length | 146 |
| Max. P | 0.985150 |

| Location | 10,293,723 – 10,293,831 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 81.93 |
| Mean single sequence MFE | -40.45 |
| Consensus MFE | -37.78 |
| Energy contribution | -37.75 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.06 |
| Mean z-score | -4.53 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.00 |
| SVM RNA-class probability | 0.985150 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
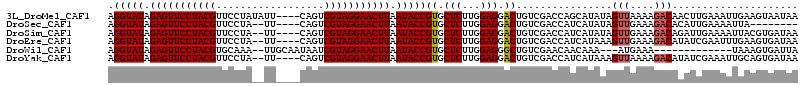
>3L_DroMel_CAF1 10293723 108 + 23771897 -GA-ACGAAAAAAACCGAAGUCUUUUUACCAUCAGCGAGGUAUAGAGUUCCUACGUUCC-UAUAUU----C--AGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCA -..-...........(((((((((....(((..((((.(((((.(((((((((((...(-(.....----.--)).))))))))))).))))).))))..))))))))).))).... ( -39.50) >DroPse_CAF1 181478 106 + 1 ----GUAUCU-AAAGCGAAGCCUCUUUACCAUCAGCGAGGUAUAGAGUUCCUACGUGCCGUA--UC----CAAAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCCAUCA ----......-...(.((((((.((...(((..((((.(((((.(((((((((((.((....--..----....))))))))))))).))))).))))..))))))))).))).... ( -38.20) >DroGri_CAF1 182337 111 + 1 AAACCGAAAG-AUAACGAAGUCUAUUUACCAUUAGCGAGGUAUAGAGUUCCUACGUGCA-AA--UUGCAAU--AAUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGACCA ....(....)-....((((((((.....(((..((((.(((((.((((((((((((((.-..--..)))..--...))))))))))).))))).))))..))).))))).))).... ( -41.40) >DroWil_CAF1 263780 108 + 1 --U-UUGAAA-UAAACGAAGCCUAUUUACCAUAAGCGAGGUAUAGAGUUCCUACGUGCA-AA--UUGCAAU--AAUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGAACA --.-......-....((((((((.....(((..((((.(((((.((((((((((((((.-..--..)))..--...))))))))))).))))).))))..))).))))).))).... ( -43.30) >DroAna_CAF1 179924 107 + 1 ----CUGAAG-AAACCGAAGCCUAUUUGCCAUCAGCGAGGUAUAGAGUUCCUACGUGCC-AC--UUAAACU--AGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGACCA ----......-....((((((((.....(((..((((.(((((.(((((((((((....-((--(......--)))))))))))))).))))).))))..))).))))).))).... ( -42.10) >DroPer_CAF1 181731 106 + 1 ----GUAUCU-AAAGCGAAGCCUCUUUACCAUCAGCGAGGUAUAGAGUUCCUACGUGCCGUA--UC----CAAAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCCAUCA ----......-...(.((((((.((...(((..((((.(((((.(((((((((((.((....--..----....))))))))))))).))))).))))..))))))))).))).... ( -38.20) >consensus ____CUAAAA_AAAACGAAGCCUAUUUACCAUCAGCGAGGUAUAGAGUUCCUACGUGCC_AA__UU____C__AGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGACCA ...............((((((((.....(((..((((.(((((.(((((((((((.....................))))))))))).))))).))))..))).))))).))).... (-37.78 = -37.75 + -0.03)



| Location | 10,293,723 – 10,293,831 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 81.93 |
| Mean single sequence MFE | -36.46 |
| Consensus MFE | -31.52 |
| Energy contribution | -31.47 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.40 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.975579 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 10293723 108 - 23771897 UGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACU--G----AAUAUA-GGAACGUAGGAACUCUAUACCUCGCUGAUGGUAAAAAGACUUCGGUUUUUUUCGU-UC- .((.(....).)).(((..(((..(((((..((((((((((.((--(----....))-)...))))))))))..)))))..)))..))).((((((((....))))))))...-..- ( -37.30) >DroPse_CAF1 181478 106 - 1 UGAUGGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUUUG----GA--UACGGCACGUAGGAACUCUAUACCUCGCUGAUGGUAAAGAGGCUUCGCUUU-AGAUAC---- .....((.(((((((((..(((..(((((..((((((((((...(((----..--..)))..))))))))))..)))))..)))..)))...)).)))))).....-......---- ( -36.00) >DroGri_CAF1 182337 111 - 1 UGGUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGAUU--AUUGCAA--UU-UGCACGUAGGAACUCUAUACCUCGCUAAUGGUAAAUAGACUUCGUUAU-CUUUCGGUUU .((((.........(((..(((..(((((..((((((((((...--..(((..--..-.)))))))))))))..)))))..)))..)))......))))(((....-....)))... ( -33.96) >DroWil_CAF1 263780 108 - 1 UGUUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGAUU--AUUGCAA--UU-UGCACGUAGGAACUCUAUACCUCGCUUAUGGUAAAUAGGCUUCGUUUA-UUUCAA-A-- ((..(((.((((..(((.((((..(((((..((((((((((...--..(((..--..-.)))))))))))))..)))))..)))).)))......)))))))..))-......-.-- ( -39.50) >DroAna_CAF1 179924 107 - 1 UGGUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACU--AGUUUAA--GU-GGCACGUAGGAACUCUAUACCUCGCUGAUGGCAAAUAGGCUUCGGUUU-CUUCAG---- .(..(((.((((..(((..(((..(((((..(((((((((((((--......)--))-....))))))))))..)))))..)))..)))......)))))))..).-......---- ( -36.00) >DroPer_CAF1 181731 106 - 1 UGAUGGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUUUG----GA--UACGGCACGUAGGAACUCUAUACCUCGCUGAUGGUAAAGAGGCUUCGCUUU-AGAUAC---- .....((.(((((((((..(((..(((((..((((((((((...(((----..--..)))..))))))))))..)))))..)))..)))...)).)))))).....-......---- ( -36.00) >consensus UGGUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGACU__A____AA__UA_GGCACGUAGGAACUCUAUACCUCGCUGAUGGUAAAUAGGCUUCGCUUU_AUUCAC____ .....((.((((..(((..(((..(((((..((((((((((.....................))))))))))..)))))..)))..)))......))))))................ (-31.52 = -31.47 + -0.06)

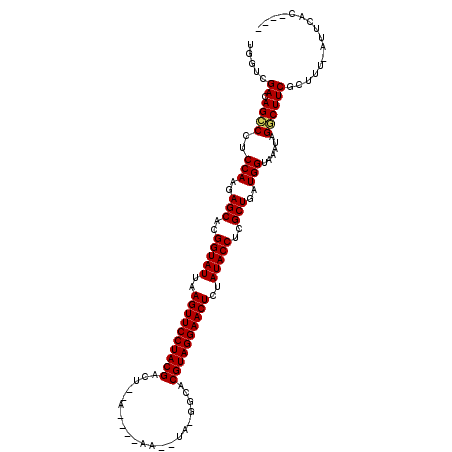
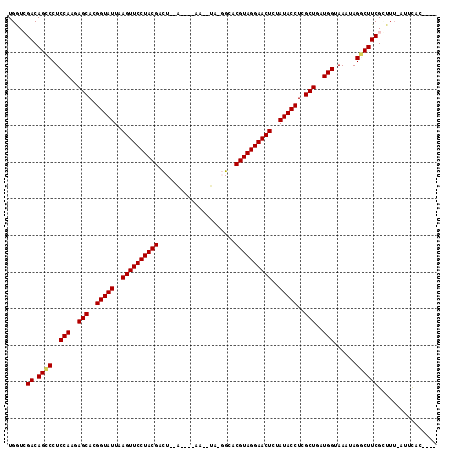
| Location | 10,293,758 – 10,293,869 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 83.48 |
| Mean single sequence MFE | -30.97 |
| Consensus MFE | -26.36 |
| Energy contribution | -26.22 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967150 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 10293758 111 + 23771897 AGGUAUAGAGUUCCUACGUUCCUAUAUU----CAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAGCAUAUAGUUAAAAGACAACUUGAAAUUGAAGUAAUAA .(((((.(((((((((((...((.....----.)).))))))))))).)))))((.(((...))).))((((....(((.....)))....))))((((.......))))..... ( -31.50) >DroSec_CAF1 168715 101 + 1 AGGUAUAGAGUUCCUACGUUCCUA--UU----CAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCAUAUAGUUGAAAGACACAUUGAAAAUUA-------- .(((((.(((((((((((......--..----....))))))))))).)))))(((.((((....((((((..........))))))..)))))))...........-------- ( -31.40) >DroSim_CAF1 175222 109 + 1 AGGUAUAGAGUUCCUACGUUCCUA--UU----CAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCAUAUAGUUGAAAGACAGAUUGAAAAUUACGUGAUAA .(((((.(((((((((((......--..----....))))))))))).)))))((.(((...))).))........((((..(((((..((......))...)))))..)))).. ( -31.90) >DroEre_CAF1 179279 109 + 1 AGGUAUAGAGUUCCUACGUUCCUA--UU----CAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCAUAAAGUUGAAAGACAUAUCGAAUUUGAAGUGAUAA .(((((.(((((((((((......--..----....))))))))))).)))))((.(((...))).))........(((((...(((....)))...(((....))).))))).. ( -31.70) >DroWil_CAF1 263813 97 + 1 AGGUAUAGAGUUCCUACGUGCAAA--UUGCAAUAAUCGUAGGAACUUAAUACCGUGCUCUUGGAGGGCUGUCGAACAACAAA---AUGAAA-------------UAAAGUGAUUA .(((((.((((((((((((((...--..))).....))))))))))).)))))..((((.....))))..............---......-------------........... ( -29.80) >DroYak_CAF1 177366 109 + 1 AGGUAUAGAGUUCCUACGUUCCUA--UU----CAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCAUAAAGUUAAAAGACAUAUCGAAAUUGCAGUGAUAA .(((((.(((((((((((......--..----....))))))))))).))))).(((..((.((....((((...................))))..)).))...)))....... ( -29.51) >consensus AGGUAUAGAGUUCCUACGUUCCUA__UU____CAGUCGUAGGAACUUAAUACCGUGCUCUUGGAGGACUGUCGACCAUCAUAUAGUUGAAAGACAUAUUGAAAUUGAAGUGAUAA .(((((.(((((((((((..................))))))))))).)))))((.(((...))).))............................................... (-26.36 = -26.22 + -0.14)
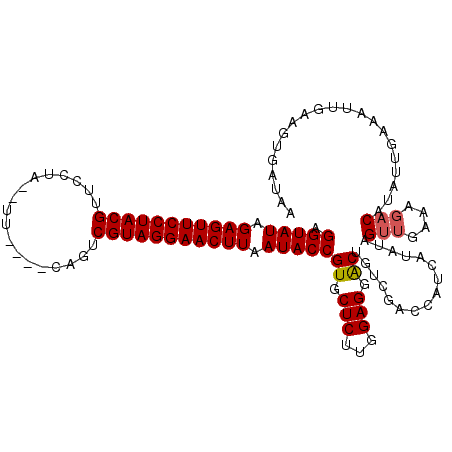

| Location | 10,293,758 – 10,293,869 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 83.48 |
| Mean single sequence MFE | -28.72 |
| Consensus MFE | -24.38 |
| Energy contribution | -25.52 |
| Covariance contribution | 1.14 |
| Combinations/Pair | 1.06 |
| Mean z-score | -3.16 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.64 |
| SVM RNA-class probability | 0.969166 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 10293758 111 - 23771897 UUAUUACUUCAAUUUCAAGUUGUCUUUUAACUAUAUGCUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUG----AAUAUAGGAACGUAGGAACUCUAUACCU .................(((((.....)))))...((((((..(.....)..))...)))).(((((..((((((((((.(((----....)))...))))))))))..))))). ( -28.80) >DroSec_CAF1 168715 101 - 1 --------UAAUUUUCAAUGUGUCUUUCAACUAUAUGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUG----AA--UAGGAACGUAGGAACUCUAUACCU --------............((((..(((.(.....).)))..))))((.(((...))).))(((((..((((((((((.((.----..--.))...))))))))))..))))). ( -28.80) >DroSim_CAF1 175222 109 - 1 UUAUCACGUAAUUUUCAAUCUGUCUUUCAACUAUAUGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUG----AA--UAGGAACGUAGGAACUCUAUACCU .......((..((((....(((((..(((.(.....).)))..)))))......))))..))(((((..((((((((((.((.----..--.))...))))))))))..))))). ( -30.60) >DroEre_CAF1 179279 109 - 1 UUAUCACUUCAAAUUCGAUAUGUCUUUCAACUUUAUGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUG----AA--UAGGAACGUAGGAACUCUAUACCU ....................((((..(((.(.....).)))..))))((.(((...))).))(((((..((((((((((.((.----..--.))...))))))))))..))))). ( -28.90) >DroWil_CAF1 263813 97 - 1 UAAUCACUUUA-------------UUUCAU---UUUGUUGUUCGACAGCCCUCCAAGAGCACGGUAUUAAGUUCCUACGAUUAUUGCAA--UUUGCACGUAGGAACUCUAUACCU ...........-------------......---.....(((((.............))))).(((((..((((((((((.....(((..--...)))))))))))))..))))). ( -27.22) >DroYak_CAF1 177366 109 - 1 UUAUCACUGCAAUUUCGAUAUGUCUUUUAACUUUAUGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUG----AA--UAGGAACGUAGGAACUCUAUACCU ..............(((((.((((............)))))))))..((.(((...))).))(((((..((((((((((.((.----..--.))...))))))))))..))))). ( -28.00) >consensus UUAUCACUUCAAUUUCAAUAUGUCUUUCAACUAUAUGAUGGUCGACAGUCCUCCAAGAGCACGGUAUUAAGUUCCUACGACUG____AA__UAGGAACGUAGGAACUCUAUACCU ....................((((..(((.(.....).)))..))))((.(((...))).))(((((..((((((((((.(((........)))...))))))))))..))))). (-24.38 = -25.52 + 1.14)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:59:59 2006