
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 10,170,997 – 10,171,089 |
| Length | 92 |
| Max. P | 0.949285 |

| Location | 10,170,997 – 10,171,089 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | forward |
| Mean pairwise identity | 80.74 |
| Mean single sequence MFE | -35.07 |
| Consensus MFE | -19.44 |
| Energy contribution | -19.67 |
| Covariance contribution | 0.22 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.89 |
| SVM RNA-class probability | 0.875188 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 10170997 92 + 23771897 ----------GUCAUUGUUGUG-UUG-UGCUGCUGUUGUUGCCACUGCUGGUGGCUUUUAAGUCAUGACAAUCAGCACGACCAACAGCCAGCGGCAAUGGCAAU ----------((((((((((.(-(((-(((((..(((((((((((.....)))))...........))))))))))))))))))).(((...)))))))))... ( -38.10) >DroPse_CAF1 59148 88 + 1 -------GAUUUCAUUGUUGCCAUUGUUGCUGCUGCUGUUG---CUUUUGGUGGCUUUUUAGUCAUGACAAUCAGCACGGCCAACAGCCAACGGCU------AU -------............(((.(((..((((..(((((.(---((.(((((((((....))))))..)))..))))))))...))))))).))).------.. ( -26.50) >DroSec_CAF1 47781 102 + 1 GGGAAUGGCUGUCAUUGUUGCG-UUG-UGCUGCUGUUGCUGCCACUACUGGUGGCUUUUUAGUCAUGACAAUCAACACGACCAACAGCCAGCGGCAAUGGCAAU .........(((((((((((((-...-..).(((((((..(((((.....)))))......(((.((........)).))))))))))..)))))))))))).. ( -36.10) >DroSim_CAF1 51719 102 + 1 GGGAAUGGCUGUCAUUGUUGCG-UUG-UGCUGCUGUUGUUGCCACUACUGGUGGCUUUUUAGUCAUGACAAUCAGCACGACCAACAGCCAGCGGCAAUGGCAAU .........(((((((((((.(-(((-(((((..(((((((((((.....)))))...........))))))))))))))))))).(((...)))))))))).. ( -38.90) >DroYak_CAF1 50857 103 + 1 GGGCAUGGCUGUCAUUGCUGUG-CUGCUGCUGCUGUUGUUGCCAUUGGUGGUGGCUUUUUAGUCAUGACAAUCAGCACGACCAACAGCCAGCGGCUAUGGCAAU .((((....))))(((((((((-((((((..(((((((.(((.....((.((((((....)))))).)).....)))....)))))))))))))).)))))))) ( -43.30) >DroPer_CAF1 59043 88 + 1 -------GAUCUCAUUGUUGCCAUUGUUGGUGCUGCUGUUG---CUUUUGGUGGCUUUUUAGUCAUGACAAUCAGCACGGCCAACAGCCAACGGCU------AU -------.......((((((...((((((((..((((((((---....(.((((((....)))))).)))).)))))..)))))))).))))))..------.. ( -27.50) >consensus _______GCUGUCAUUGUUGCG_UUG_UGCUGCUGUUGUUGCCACUACUGGUGGCUUUUUAGUCAUGACAAUCAGCACGACCAACAGCCAGCGGCAAUGGCAAU ............................(((((((((((((.........((((((....)))))).....((.....)).)))))).)))))))......... (-19.44 = -19.67 + 0.22)



| Location | 10,170,997 – 10,171,089 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 80.74 |
| Mean single sequence MFE | -31.47 |
| Consensus MFE | -22.15 |
| Energy contribution | -23.35 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.94 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.39 |
| SVM RNA-class probability | 0.949285 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 10170997 92 - 23771897 AUUGCCAUUGCCGCUGGCUGUUGGUCGUGCUGAUUGUCAUGACUUAAAAGCCACCAGCAGUGGCAACAACAGCAGCA-CAA-CACAACAAUGAC---------- .((((((((((.(.(((((...((((((((.....).)))))))....))))).).))))))))))...........-...-............---------- ( -34.30) >DroPse_CAF1 59148 88 - 1 AU------AGCCGUUGGCUGUUGGCCGUGCUGAUUGUCAUGACUAAAAAGCCACCAAAAG---CAACAGCAGCAGCAACAAUGGCAACAAUGAAAUC------- ..------.((((((((((((((....((((..(((...((.((....)).)).))).))---))....)))))))..)))))))............------- ( -25.90) >DroSec_CAF1 47781 102 - 1 AUUGCCAUUGCCGCUGGCUGUUGGUCGUGUUGAUUGUCAUGACUAAAAAGCCACCAGUAGUGGCAGCAACAGCAGCA-CAA-CGCAACAAUGACAGCCAUUCCC ..(((((((((.(.(((((.(((((((((........)))))))))..))))).).)))))))))((....)).((.-...-.))................... ( -34.90) >DroSim_CAF1 51719 102 - 1 AUUGCCAUUGCCGCUGGCUGUUGGUCGUGCUGAUUGUCAUGACUAAAAAGCCACCAGUAGUGGCAACAACAGCAGCA-CAA-CGCAACAAUGACAGCCAUUCCC .((((((((((.(.(((((.((((((((((.....).)))))))))..))))).).))))))))))........((.-...-.))................... ( -34.50) >DroYak_CAF1 50857 103 - 1 AUUGCCAUAGCCGCUGGCUGUUGGUCGUGCUGAUUGUCAUGACUAAAAAGCCACCACCAAUGGCAACAACAGCAGCAGCAG-CACAGCAAUGACAGCCAUGCCC (((((....((.((((((((((((((((((.....).))))))......((((.......))))..)))))))..)))).)-)...)))))............. ( -34.40) >DroPer_CAF1 59043 88 - 1 AU------AGCCGUUGGCUGUUGGCCGUGCUGAUUGUCAUGACUAAAAAGCCACCAAAAG---CAACAGCAGCACCAACAAUGGCAACAAUGAGAUC------- ..------.((((((((((((((....((((..(((...((.((....)).)).))).))---)).))))))).....)))))))............------- ( -24.80) >consensus AUUGCCAUAGCCGCUGGCUGUUGGUCGUGCUGAUUGUCAUGACUAAAAAGCCACCAGAAGUGGCAACAACAGCAGCA_CAA_CGCAACAAUGACAGC_______ ............((((.(((((((((((((.....).))))))......(((((.....)))))..))))))))))............................ (-22.15 = -23.35 + 1.20)

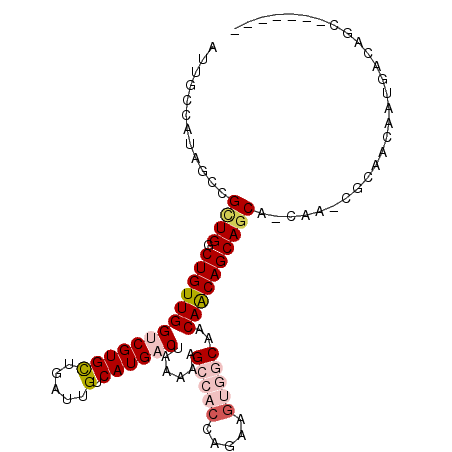
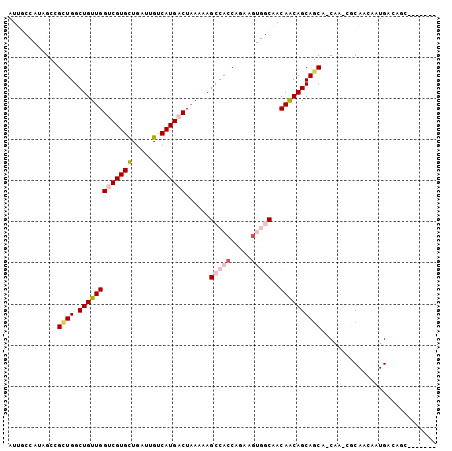
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:58:37 2006