
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 10,100,151 – 10,100,252 |
| Length | 101 |
| Max. P | 0.603413 |

| Location | 10,100,151 – 10,100,252 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 80.79 |
| Mean single sequence MFE | -32.65 |
| Consensus MFE | -19.83 |
| Energy contribution | -20.95 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.603413 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 10100151 101 - 23771897 ---------CGAGUGUGUGGUUAUCUGAGUUGAUUUAUUGGCCAGGUCAAAGUCGCAGCCGAGUUUGGGCCAUAACUCAAAGCGA----GCUACUUGGACCACAAAUAUUUGCU ---------(((((((((((((...(((((((......(((((....((((.(((....))).))))))))))))))))...(((----(...)))))))))))..)))))).. ( -30.50) >DroPse_CAF1 42634 101 - 1 -------UGUGUGUGUGUGG----GCUAGUUGAUUUAUUGGCCAGGCCAAAGCCACAGCACAGCUC--GCCAUAACUCAAAUACACGCCGCCCCUUGGACCACAAAUAUUUGCU -------.((((((..((.(----((.(((((.....(((((...))))).((....)).))))).--)))...))....))))))((......(((.....)))......)). ( -26.20) >DroSec_CAF1 39083 101 - 1 ---------CGAGUGUGUGGUUAUCUGAGUUGAUUUAUUGGCCAGGCCAAAGUCGCAGCCGGGUUUGGGCUAUAACUCAAAGCGA----GCUACUUGGACCACAAAUAUUUGCU ---------(((((((((((((...(((((((((((..((((...)))))))))..((((.......))))..))))))...(((----(...)))))))))))..)))))).. ( -29.30) >DroSim_CAF1 39888 101 - 1 ---------CGAGUGUGUGGUUAUCUGAGUUGAUUUAUUGGCCAGGCCAAAGUCGCAGCCGGGUUUGGGCCAUAACUCAAAGCGA----GCUACUUGGACCACAAAUAUUUGCU ---------(((((((((((((...(((((((......((((((((((...((....))..))))).))))))))))))...(((----(...)))))))))))..)))))).. ( -31.00) >DroEre_CAF1 43448 100 - 1 ---------UGAGUGUGUGGUUGUCUGAGUUGAUUUAUUGGCCAGGCCA-AGUCGCAGCCGGGCUUGGCCCAUAACUCAAAGCGA----GCUACUUGGACCACAAAUAUUUGCU ---------.((((((...((.(((..((((((.((((.(((((((((.-.((....))..))))))))).)))).))).((...----.)))))..))).))..))))))... ( -37.30) >DroAna_CAF1 36850 108 - 1 ACACACGCAUAGAUGUGU--GUAUUUGAGUUGAUUUAUUGGCCCGGCCAAAGCCACACUCGGCCCUUGGCCAUAACUCAAAGCGG----GCUACUUGGUCCGCAAAUAUUUGCU ..(((((((....)))))--)).(((((((((......(((((.((((............))))...))))))))))))))((((----(((....)))))))........... ( -41.60) >consensus _________CGAGUGUGUGGUUAUCUGAGUUGAUUUAUUGGCCAGGCCAAAGUCGCAGCCGGGCUUGGGCCAUAACUCAAAGCGA____GCUACUUGGACCACAAAUAUUUGCU .........(((((((((((((...(((((((......((((((((((...((....))..))))).)))))))))))).(((......))).....)))))))..)))))).. (-19.83 = -20.95 + 1.12)

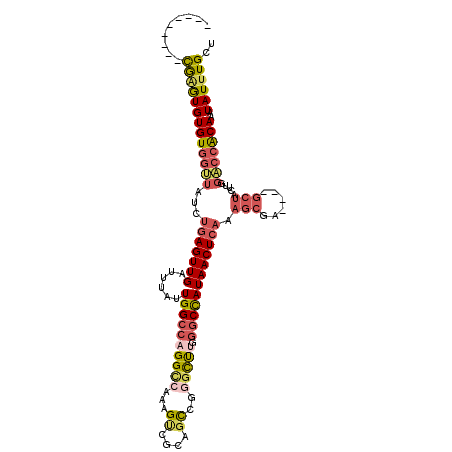
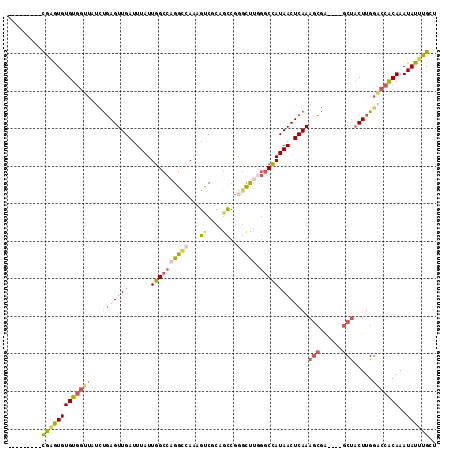
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:58:11 2006