
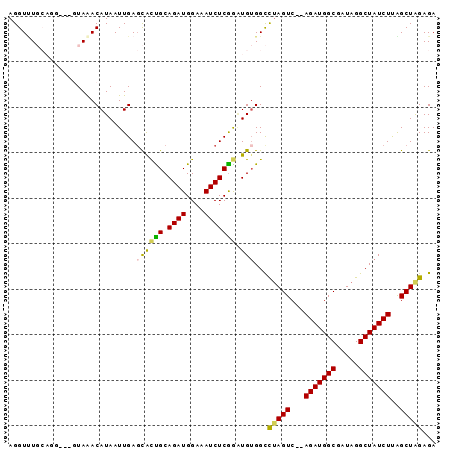
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 9,848,992 – 9,849,140 |
| Length | 148 |
| Max. P | 0.917981 |

| Location | 9,848,992 – 9,849,084 |
|---|---|
| Length | 92 |
| Sequences | 5 |
| Columns | 94 |
| Reading direction | forward |
| Mean pairwise identity | 83.85 |
| Mean single sequence MFE | -27.02 |
| Consensus MFE | -18.86 |
| Energy contribution | -18.46 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.916831 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9848992 92 + 23771897 AGGUUUGCAGGUUUGUAAACAUAAUUGAGCAUUGCAGAUGGAAAUCUCGGUUGUGGCCUAGUC--AGAUGGCGAUAGGCUAUCUUAGCUAGAGA ..((((((......))))))(((((((((.(((.........))))))))))))...(((((.--(((((((.....)))))))..)))))... ( -26.30) >DroSec_CAF1 10004 88 + 1 AGGUUUGCAGG----UAAACAUAAUUGAGCACCGCAGAUGGAAAUCUCGGAUGUGGCCUAGUC--AGAUGGCGGUAGGCUAUCUUAGCUAGAGA ..(((((....----)))))........((((((.((((....))))))).)))...(((((.--(((((((.....)))))))..)))))... ( -26.80) >DroSim_CAF1 9868 87 + 1 AGGU-UGCAGG----UAAACAUAAUUGAGCACCGCAGAUGGAAAUCUCGGAUGUGGCCUAGUC--AGAUGGCGAUAGGCUAUCUUAGCUAGAGA .(((-..((..----....((....))....(((.((((....))))))).))..)))((((.--(((((((.....)))))))..)))).... ( -27.60) >DroYak_CAF1 10788 84 + 1 AGGUUU--------GUAGACAUAAUUGAUCGCUGCAGAUGGAAAUCUCGGAUGUGGCUAAGUC--AGAUGGCGAUAGGCUAUCUUGGCUGGAGA .((((.--------.((....))...))))((..((..(((.....)))..))..))..((((--(((((((.....)))))).)))))..... ( -22.90) >DroAna_CAF1 9457 86 + 1 AGGUCU--------GUAAACAUAAUUGAGUGCUGCAGAUGGAGAUCUCAAGUGGGGUCUAGUCGGAGAUGGCGAUAGGCUAUCUCGGCUGGAGA ..((((--------(((.((........))..)))))))..(((((((....)))))))(((((.(((((((.....))))))))))))..... ( -31.50) >consensus AGGUUUGCAGG___GUAAACAUAAUUGAGCACUGCAGAUGGAAAUCUCGGAUGUGGCCUAGUC__AGAUGGCGAUAGGCUAUCUUAGCUAGAGA ...................((((.(((((..((......))....))))).))))..(((((...(((((((.....)))))))..)))))... (-18.86 = -18.46 + -0.40)


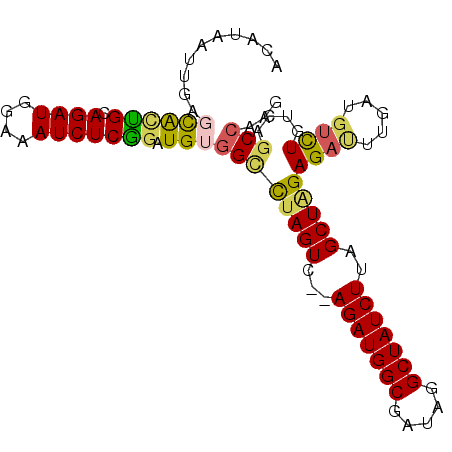
| Location | 9,849,010 – 9,849,104 |
|---|---|
| Length | 94 |
| Sequences | 5 |
| Columns | 96 |
| Reading direction | forward |
| Mean pairwise identity | 86.39 |
| Mean single sequence MFE | -30.22 |
| Consensus MFE | -20.82 |
| Energy contribution | -21.34 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.749684 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9849010 94 + 23771897 ACAUAAUUGAGCAUUGCAGAUGGAAAUCUCGGUUGUGGCCUAGUC--AGAUGGCGAUAGGCUAUCUUAGCUAGAGAUUUGAUUUCUGUCAUCGAAG ......((((.....(((((...((((((((((((((((((((((--....)))..))))))))...)))).)))))))....)))))..)))).. ( -27.30) >DroSec_CAF1 10018 94 + 1 ACAUAAUUGAGCACCGCAGAUGGAAAUCUCGGAUGUGGCCUAGUC--AGAUGGCGGUAGGCUAUCUUAGCUAGAGAUUUGAUGUCUGUCAGCCAAG ..........((((((.((((....))))))).)))((((((((.--(((((((.....)))))))..)))))((((.....))))....)))... ( -30.70) >DroSim_CAF1 9881 94 + 1 ACAUAAUUGAGCACCGCAGAUGGAAAUCUCGGAUGUGGCCUAGUC--AGAUGGCGAUAGGCUAUCUUAGCUAGAGAUUUGAUGUCUGUUAGCCAAG ..........((((((.((((....))))))).)))((((((((.--(((((((.....)))))))..)))))((((.....))))....)))... ( -30.70) >DroYak_CAF1 10798 94 + 1 ACAUAAUUGAUCGCUGCAGAUGGAAAUCUCGGAUGUGGCUAAGUC--AGAUGGCGAUAGGCUAUCUUGGCUGGAGAUUUGAUGUCUUUGAGCCCAG .............(((.((((....))))))).((.((((.((((--(((((((.....)))))).)))))((((((.....)))))).)))))). ( -29.60) >DroAna_CAF1 9467 96 + 1 ACAUAAUUGAGUGCUGCAGAUGGAGAUCUCAAGUGGGGUCUAGUCGGAGAUGGCGAUAGGCUAUCUCGGCUGGAGACUUGAUGCUUUUGAGCCAAG .....((..(((.((.(((....(((((((....)))))))...(.((((((((.....)))))))).)))).)))))..))(((....))).... ( -32.80) >consensus ACAUAAUUGAGCACUGCAGAUGGAAAUCUCGGAUGUGGCCUAGUC__AGAUGGCGAUAGGCUAUCUUAGCUAGAGAUUUGAUGUCUGUCAGCCAAG ...............(((((((.(((((((((......))((((...(((((((.....)))))))..)))))))))))..)))))))........ (-20.82 = -21.34 + 0.52)

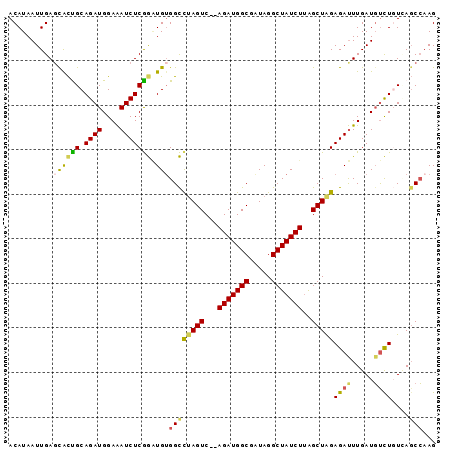
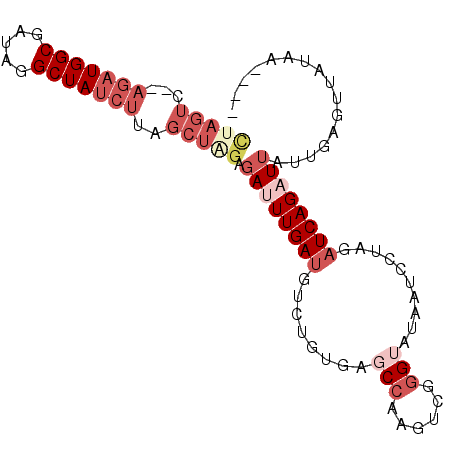
| Location | 9,849,049 – 9,849,140 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 97 |
| Reading direction | forward |
| Mean pairwise identity | 75.12 |
| Mean single sequence MFE | -25.80 |
| Consensus MFE | -11.93 |
| Energy contribution | -14.40 |
| Covariance contribution | 2.47 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.46 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.917981 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9849049 91 + 23771897 CUAGUC--AGAUGGCGAUAGGCUAUCUUAGCUAGAGAUUUGAUUUCUGUCAUCGAAGUCGGGUAUAAUCCUAGAUCAGAUUAUUGAGUUAUAA---- (((((.--(((((((.....)))))))..))))).((((..((.((((((..((....))((......))..)).))))..))..))))....---- ( -23.90) >DroSec_CAF1 10057 90 + 1 CUAGUC--AGAUGGCGGUAGGCUAUCUUAGCUAGAGAUUUGAUGUCUGUCAGCCAAGUAGGGUAUAAUCCUAGAUCAGAU-AUUGAGUUAUAA---- (((((.--(((((((.....)))))))..))))).((((..(((((((((.......((((((...)))))))).)))))-))..))))....---- ( -29.11) >DroSim_CAF1 9920 91 + 1 CUAGUC--AGAUGGCGAUAGGCUAUCUUAGCUAGAGAUUUGAUGUCUGUUAGCCAAGUCGGGUAUAAUCCUAGAUCAGAUUAUUGAGUUAUUA---- (((((.--(((((((.....)))))))..))))).((((..(((((((........((((((......))).))))))).)))..))))....---- ( -23.90) >DroEre_CAF1 10320 86 + 1 -----------UGGCGAUAGGCUAUCUUAGCUGGAGAUUUGAUGUCUGCGAGCCAAGUCGGGUAUAAUCCUAGAUCAGAUUAAUAAGUUGGAACUUG -----------((((....(((((...)))))(.((((.....)))).)..)))).((((((......))).)))........(((((....))))) ( -16.90) >DroYak_CAF1 10837 94 + 1 UAAGUC--AGAUGGCGAUAGGCUAUCUUGGCUGGAGAUUUGAUGUCUUUGAGCCCAGUUGGGUAUAAUCCUAGAUCAGCUU-UUAAGUUUAAUCUUG ..((((--(((((((.....)))))).))))).((((((.(((......((((....(((((......)))))....))))-....))).)))))). ( -27.30) >DroAna_CAF1 9506 78 + 1 CUAGUCGGAGAUGGCGAUAGGCUAUCUCGGCUGGAGACUUGAUGCUUUUGAGCCAAGUUUGGG----------CUCAGAUUAAGG---CAA------ (((((((.(((((((.....))))))))))))))........((((((((((((.......))----------))))))....))---)).------ ( -33.70) >consensus CUAGUC__AGAUGGCGAUAGGCUAUCUUAGCUAGAGAUUUGAUGUCUGUGAGCCAAGUCGGGUAUAAUCCUAGAUCAGAUUAUUGAGUUAUAA____ (((((...(((((((.....)))))))..))))).((((((((........(((......)))..........))))))))................ (-11.93 = -14.40 + 2.47)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:56:27 2006