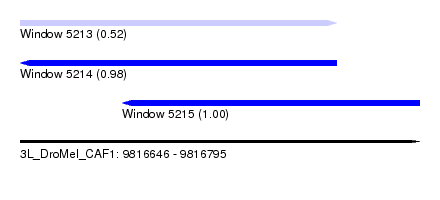
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 9,816,646 – 9,816,795 |
| Length | 149 |
| Max. P | 0.999007 |

| Location | 9,816,646 – 9,816,764 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.87 |
| Mean single sequence MFE | -30.96 |
| Consensus MFE | -23.68 |
| Energy contribution | -24.68 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.76 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.522630 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
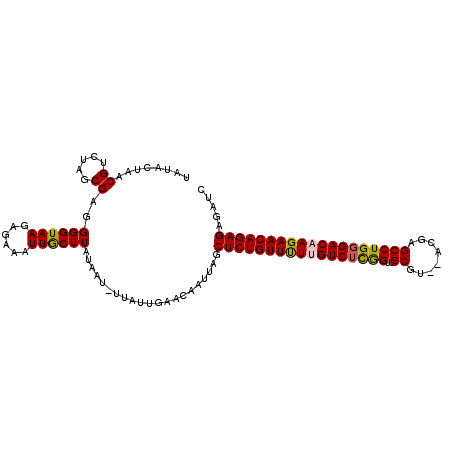
>3L_DroMel_CAF1 9816646 118 + 23771897 CGGGGUCUUAGCAUCUGCUUUUCGAUAUCUCGCAC--UCAGAUCUCUCUGUUCUUGUCCCAGGCUAGUAUAUGCAACCAAGACAAAAACAGAGCCAAUUGUUCAAUAAUGUUAUAAGUAA .(((((((.(((....)))...(((....)))...--..))))).(((((((.(((((....((........))......))))).)))))))))......................... ( -23.20) >DroSec_CAF1 110722 115 + 1 CGGGGUCUUAGCAUCUGCUCUUCGAGAUCUCGCAC--UCAGAUCCCUCUGUUCUUGUCCCAGGCUCGU--ACGCAACCGAGACAAAAACAGAGCUAAGUGUUCAAUAA-AUUAUAAGCAA .(((((((((((....)))....))))))))((((--(.((....(((((((.((((((...((....--..))....).))))).))))))))).))))).......-........... ( -32.00) >DroSim_CAF1 114057 115 + 1 CGGGGUCUUAGCAUCUGCUCUUCGAGAUCUCGCAC--UCAGAUCCCUCUGUUCUUGUCCCAGGCUCGU--ACGCAACCGAGACAAAAACAGAGCUAAUUGUUCAAUAA-AUUAUAAGCAA .(((((((((((....)))....))))))))((..--........(((((((.((((((...((....--..))....).))))).))))))).(((((........)-))))...)).. ( -30.00) >DroEre_CAF1 113599 113 + 1 CGGGGUCUUAGCAUCUGCACUUCGAGAUCUCGCGC--UCGGU--UCUCUGUUCUGGUCCCAGGCUCAU--GCGCAACCGAGACAAGAACAGAGUUAAAUGUUUGACAA-AUUACAAGCAA (((((((((.((....)).....))))))))).((--((.((--(((.(((..((((.....((....--..)).))))..)))))))).))))....((((((....-....)))))). ( -32.70) >DroYak_CAF1 123637 115 + 1 CGGGGUCUUAGCAUCUGCACUUCGAGAUCUCGCAGUGUCAGU--UCUCUGUUCUGGUCACAGGCUCAU--ACGCAACUGAGACGAGAACAGAGGUAAAUGUUUAACAA-AUUACAAGCAA ..........((..(((((((.(((....))).)))).))).--((((((((((.(((.(((((....--..))..))).))).))))))))))..............-.......)).. ( -36.90) >consensus CGGGGUCUUAGCAUCUGCUCUUCGAGAUCUCGCAC__UCAGAUCUCUCUGUUCUUGUCCCAGGCUCGU__ACGCAACCGAGACAAAAACAGAGCUAAAUGUUCAAUAA_AUUAUAAGCAA ((((((((((((....)))....))))))))).............(((((((.(((((....((........))......))))).)))))))........................... (-23.68 = -24.68 + 1.00)


| Location | 9,816,646 – 9,816,764 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.87 |
| Mean single sequence MFE | -37.28 |
| Consensus MFE | -31.08 |
| Energy contribution | -31.88 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.31 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.980588 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9816646 118 - 23771897 UUACUUAUAACAUUAUUGAACAAUUGGCUCUGUUUUUGUCUUGGUUGCAUAUACUAGCCUGGGACAAGAACAGAGAGAUCUGA--GUGCGAGAUAUCGAAAAGCAGAUGCUAAGACCCCG .........................((((((((((((((((..(..((........)))..))))))))))))))(((((((.--...(((....))).....))))).))......)). ( -34.40) >DroSec_CAF1 110722 115 - 1 UUGCUUAUAAU-UUAUUGAACACUUAGCUCUGUUUUUGUCUCGGUUGCGU--ACGAGCCUGGGACAAGAACAGAGGGAUCUGA--GUGCGAGAUCUCGAAGAGCAGAUGCUAAGACCCCG ((((((.....-........((((((((((((((((((((((((..((..--....)))))))))))))))))))....))))--)))(((....)))..)))))).............. ( -41.30) >DroSim_CAF1 114057 115 - 1 UUGCUUAUAAU-UUAUUGAACAAUUAGCUCUGUUUUUGUCUCGGUUGCGU--ACGAGCCUGGGACAAGAACAGAGGGAUCUGA--GUGCGAGAUCUCGAAGAGCAGAUGCUAAGACCCCG ((((((.....-................((((((((((((((((..((..--....))))))))))))))))))(((((((..--.....)))))))...)))))).............. ( -38.10) >DroEre_CAF1 113599 113 - 1 UUGCUUGUAAU-UUGUCAAACAUUUAACUCUGUUCUUGUCUCGGUUGCGC--AUGAGCCUGGGACCAGAACAGAGA--ACCGA--GCGCGAGAUCUCGAAGUGCAGAUGCUAAGACCCCG ...((((..((-((((...((......(((((((((.(((((((..((..--....))))))))).))))))))).--..(((--((....)..))))..))))))))..))))...... ( -36.30) >DroYak_CAF1 123637 115 - 1 UUGCUUGUAAU-UUGUUAAACAUUUACCUCUGUUCUCGUCUCAGUUGCGU--AUGAGCCUGUGACCAGAACAGAGA--ACUGACACUGCGAGAUCUCGAAGUGCAGAUGCUAAGACCCCG ...((((((..-...............(((((((((.(((.(((..((..--....))))).))).))))))))).--.(((.((((.(((....))).))))))).))).)))...... ( -36.30) >consensus UUGCUUAUAAU_UUAUUGAACAAUUAGCUCUGUUUUUGUCUCGGUUGCGU__ACGAGCCUGGGACAAGAACAGAGAGAUCUGA__GUGCGAGAUCUCGAAGAGCAGAUGCUAAGACCCCG ...((((....................(((((((((((((((((..((........)))))))))))))))))))..(((((......(((....))).....)))))..))))...... (-31.08 = -31.88 + 0.80)

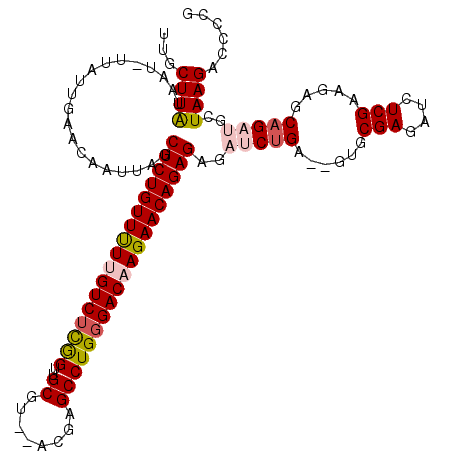
| Location | 9,816,684 – 9,816,795 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 87.98 |
| Mean single sequence MFE | -32.64 |
| Consensus MFE | -29.66 |
| Energy contribution | -29.54 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.51 |
| Structure conservation index | 0.91 |
| SVM decision value | 3.32 |
| SVM RNA-class probability | 0.999007 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9816684 111 - 23771897 UAUACUAACGUCUAGCGAGGGGUAAGAGAAAUUACUUAUAACAUUAUUGAACAAUUGGCUCUGUUUUUGUCUUGGUUGCAUAUACUAGCCUGGGACAAGAACAGAGAGAUC .........((((.....(..(((((........)))))..)................((((((((((((((..(..((........)))..)))))))))))))))))). ( -30.50) >DroSec_CAF1 110760 108 - 1 UAUACUAACGUCUAGCGAGGGGUAAGAGAAAUUGCUUAUAAU-UUAUUGAACACUUAGCUCUGUUUUUGUCUCGGUUGCGU--ACGAGCCUGGGACAAGAACAGAGGGAUC ....((((.((.(((..((..(((((........)))))..)-)..))).))..))))(((((((((((((((((..((..--....)))))))))))))))))))..... ( -33.40) >DroSim_CAF1 114095 108 - 1 UAUACUAACGUCUAGCGAGGGGUAAGAGAAAUUGCUUAUAAU-UUAUUGAACAAUUAGCUCUGUUUUUGUCUCGGUUGCGU--ACGAGCCUGGGACAAGAACAGAGGGAUC ....((.((.(((....))).)).))...(((((.(((....-....))).)))))..(((((((((((((((((..((..--....)))))))))))))))))))..... ( -33.30) >DroEre_CAF1 113637 106 - 1 UAUACUAACGUCUAGCGAGGGGUAAGAGAAAUUGCUUGUAAU-UUGUCAAACAUUUAACUCUGUUCUUGUCUCGGUUGCGC--AUGAGCCUGGGACCAGAACAGAGA--AC ..((((..((.....))...)))).((.((((((....))))-)).))..........(((((((((.(((((((..((..--....))))))))).))))))))).--.. ( -34.60) >DroYak_CAF1 123677 106 - 1 UAUACUAACGUCUAGCGAGGGGUAAGAGAAAUUGCUUGUAAU-UUGUUAAACAUUUACCUCUGUUCUCGUCUCAGUUGCGU--AUGAGCCUGUGACCAGAACAGAGA--AC .........((.(((((((((((((......))))))....)-)))))).))......(((((((((.(((.(((..((..--....))))).))).))))))))).--.. ( -31.40) >consensus UAUACUAACGUCUAGCGAGGGGUAAGAGAAAUUGCUUAUAAU_UUAUUGAACAAUUAGCUCUGUUUUUGUCUCGGUUGCGU__ACGAGCCUGGGACAAGAACAGAGAGAUC ........((.....))..((((((......)))))).....................(((((((((((((((((..((........)))))))))))))))))))..... (-29.66 = -29.54 + -0.12)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:36 2006