
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 9,815,091 – 9,815,681 |
| Length | 590 |
| Max. P | 0.999977 |

| Location | 9,815,091 – 9,815,211 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.08 |
| Mean single sequence MFE | -44.00 |
| Consensus MFE | -41.72 |
| Energy contribution | -41.00 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.07 |
| Structure conservation index | 0.95 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.595895 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815091 120 + 23771897 AAGGCCUGUGGUGAAUGCUUCUGCAGAUCGGUGGCCAGCGGCACCAGAGGAUUGUGCUGUUGGUGUUGCGUCUGCGACAGAGACUGGUGAUAGGUGAGCGGAUCUAGCAGUGACUCCAGC .....(((..((.(.(((((((((..(((.((.((((((((((((((.((......)).))))))))))((((.(....))))))))).)).)))..)))))...)))).).))..))). ( -45.70) >DroSec_CAF1 109206 120 + 1 AAGGCCUGUGGUGAAUGUUUUUGCAGAUCGUUGGCCAGCGGCACCAGAGGAUUGUGCUGUUGGUGUUGCGUUUGCGACAGAUUCUGGUGAUAGGUGAGCGGAUCUAGCAGUGACUCCAGC ...((((((.(.((((.(..(((((((.(((..(((((((((((.........)))))))))))...)))))))))).).))))...).))))))..(((((((.......)).))).)) ( -40.40) >DroSim_CAF1 112541 120 + 1 AAGGCCUGUGGUGAAUGUUUUUGCAGAUCGUUGGCCAGCGGCACCAGAGGAUUGUGCUGUUGGUGUUGCGUUUGCGACAGAUUCUGGUGAUAGGUGAGCGGAUCUAGCAGUGACUCCAGC ...((((((.(.((((.(..(((((((.(((..(((((((((((.........)))))))))))...)))))))))).).))))...).))))))..(((((((.......)).))).)) ( -40.40) >DroEre_CAF1 112048 120 + 1 AAGGCCUGUGGUGAAUGCUUCUGCAGAUCGGUGGCCAGCGGCACUAGAGGAUUGUGCUGCUGGUGUUGCGUCUGCGACAGAUGUUGGCGAUAGGUGAGCGGAUCGAGCAGUGACUCCAGC ...((((((.((.((((((..(((((((.....(((((((((((.........))))))))))).....)))))))..)).)))).)).))))))..((((((((.....))).))).)) ( -47.80) >DroYak_CAF1 122128 120 + 1 AAGGCCUGUGGUGAAUGCUUCUGCAGAUCGGUGGCCAGCGGCACUAGGGGAUUGUGCUGCUGGUGUUGCGUCUGCGACAAAUGCUGGUGAUAGGUGAGCGGAUCAAGCAGUGACUCCAGC .....(((..((.(.(((((((((..(((.((.((((((((((((((.((......)).))))))))..(((...)))....)))))).)).)))..)))))...)))).).))..))). ( -45.70) >consensus AAGGCCUGUGGUGAAUGCUUCUGCAGAUCGGUGGCCAGCGGCACCAGAGGAUUGUGCUGUUGGUGUUGCGUCUGCGACAGAUGCUGGUGAUAGGUGAGCGGAUCUAGCAGUGACUCCAGC .....(((..((.(.(((((((((..(((....(((((((((((.........)))))))))))(((((....)))))..............)))..)))))...)))).).))..))). (-41.72 = -41.00 + -0.72)

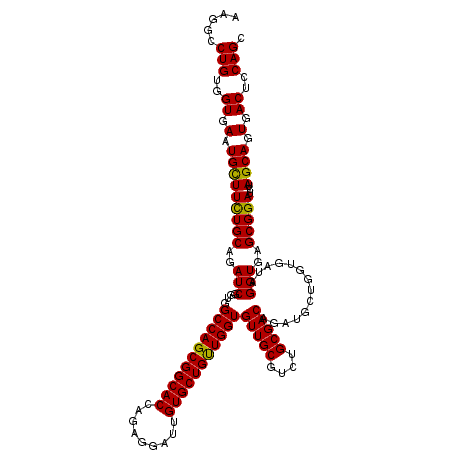
| Location | 9,815,131 – 9,815,251 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.92 |
| Mean single sequence MFE | -32.09 |
| Consensus MFE | -28.23 |
| Energy contribution | -28.51 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.565128 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815131 120 - 23771897 UGCAGCGCAUCCACACGCUGAUCAUGUCUAACAUGAGUGUGCUGGAGUCACUGCUAGAUCCGCUCACCUAUCACCAGUCUCUGUCGCAGACGCAACACCAACAGCACAAUCCUCUGGUGC .((((.(..(((((((((...(((((.....)))))))))).))))..).)))).................((((((....(((.((................))))).....)))))). ( -32.09) >DroSec_CAF1 109246 120 - 1 UGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGGGUGUGCUGGAGUCACUGCUAGAUCCGCUCACCUAUCACCAGAAUCUGUCGCAAACGCAACACCAACAGCACAAUCCUCUGGUGC .((((.(..((((((((((...((((.....)))))))))).))))..).)))).................(((((((...(((.((................)))))....))))))). ( -32.99) >DroSim_CAF1 112581 120 - 1 UGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGUGCUGGAGUCACUGCUAGAUCCGCUCACCUAUCACCAGAAUCUGUCGCAAACGCAACACCAACAGCACAAUCCUCUGGUGC .((((.(..(((((((((...(((((.....)))))))))).))))..).)))).................(((((((...(((.((................)))))....))))))). ( -33.29) >DroEre_CAF1 112088 120 - 1 UGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGUGCUGGAGUCACUGCUCGAUCCGCUCACCUAUCGCCAACAUCUGUCGCAGACGCAACACCAGCAGCACAAUCCUCUAGUGC ..(((((........))))).(((((.....)))))(((((((((.((...(((.((((..(....)..))))......((((...)))).))))).)))))).)))............. ( -30.20) >DroYak_CAF1 122168 120 - 1 UGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGUGCUGGAGUCACUGCUUGAUCCGCUCACCUAUCACCAGCAUUUGUCGCAGACGCAACACCAGCAGCACAAUCCCCUAGUGC ..(((((........))))).(((((.....)))))(((((((((......((((((((..(....)..))))..))))..(((.((....)).))))))))).)))............. ( -31.90) >consensus UGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGUGCUGGAGUCACUGCUAGAUCCGCUCACCUAUCACCAGAAUCUGUCGCAGACGCAACACCAACAGCACAAUCCUCUGGUGC .((((.(..(((((((((...(((((.....)))))))))).))))..).)))).................((((((....(((.((................))))).....)))))). (-28.23 = -28.51 + 0.28)


| Location | 9,815,171 – 9,815,291 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.42 |
| Mean single sequence MFE | -49.60 |
| Consensus MFE | -46.90 |
| Energy contribution | -47.34 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.02 |
| Mean z-score | -2.91 |
| Structure conservation index | 0.95 |
| SVM decision value | 4.72 |
| SVM RNA-class probability | 0.999942 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815171 120 + 23771897 AGACUGGUGAUAGGUGAGCGGAUCUAGCAGUGACUCCAGCACACUCAUGUUAGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACUAAAUCCAUCUG (((.(((...(((...(((((((((.((((((..((((.(((.((((((.....)))))...).)))))))..)))))))))))..((((((....)))))))))).)))...)))))). ( -51.10) >DroSec_CAF1 109286 120 + 1 AUUCUGGUGAUAGGUGAGCGGAUCUAGCAGUGACUCCAGCACACCCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACCAAAUCCAUCUG ....(((((((.((((...(((((((((..(((((.((((.((((((((((((......)))))))).)).)).)))).)).)))...)))))))))..))))))))))).......... ( -51.70) >DroSim_CAF1 112621 120 + 1 AUUCUGGUGAUAGGUGAGCGGAUCUAGCAGUGACUCCAGCACACUCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGGUCCAGCGCCGUUACCAAAUCCAUCUG ....(((((((.(((..((.(((((.((((((..((((.(((.((((((.....)))))...).)))))))..)))))))))))..))((((....)))))))))))))).......... ( -50.00) >DroEre_CAF1 112128 120 + 1 AUGUUGGCGAUAGGUGAGCGGAUCGAGCAGUGACUCCAGCACACUCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACCAAAUCCAUCUG ....(((.....(((((.((((((..((((((..((((.(((.((((((.....)))))...).)))))))..)))))).))))..((((((....)))))))))))))....))).... ( -48.00) >DroYak_CAF1 122208 120 + 1 AUGCUGGUGAUAGGUGAGCGGAUCAAGCAGUGACUCCAGCACACUCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACCAAAUCCAUCUG ....(((((((.(((..((.((((..((((((..((((.(((.((((((.....)))))...).)))))))..)))))).))))..))((((....)))))))))))))).......... ( -47.20) >consensus AUGCUGGUGAUAGGUGAGCGGAUCUAGCAGUGACUCCAGCACACUCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACCAAAUCCAUCUG ....(((.....(((((.(((((((.((((((..((((.(((.((((((.....)))))...).)))))))..)))))))))))..((((((....)))))))))))))....))).... (-46.90 = -47.34 + 0.44)



| Location | 9,815,171 – 9,815,291 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.42 |
| Mean single sequence MFE | -46.62 |
| Consensus MFE | -45.18 |
| Energy contribution | -45.26 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.10 |
| Structure conservation index | 0.97 |
| SVM decision value | 5.17 |
| SVM RNA-class probability | 0.999977 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815171 120 - 23771897 CAGAUGGAUUUAGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCUAACAUGAGUGUGCUGGAGUCACUGCUAGAUCCGCUCACCUAUCACCAGUCU ..(((((....(((...(((((((....))))))).(((((((((.(..(((((((((...(((((.....)))))))))).))))..).)))).))))).)))...)))))........ ( -46.70) >DroSec_CAF1 109286 120 - 1 CAGAUGGAUUUGGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGGGUGUGCUGGAGUCACUGCUAGAUCCGCUCACCUAUCACCAGAAU ..(((((((((((((..(((((((....))))))).((.((.(((((((((((......(((...))).....))))))))))).))))..))))))))))).))............... ( -48.70) >DroSim_CAF1 112621 120 - 1 CAGAUGGAUUUGGUAACGGCGCUGGACCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGUGCUGGAGUCACUGCUAGAUCCGCUCACCUAUCACCAGAAU ........((((((...(((((((....))))))).(((((((((.(..(((((((((...(((((.....)))))))))).))))..).)))).)))))............)))))).. ( -47.10) >DroEre_CAF1 112128 120 - 1 CAGAUGGAUUUGGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGUGCUGGAGUCACUGCUCGAUCCGCUCACCUAUCGCCAACAU ..(.(((....(((...(((((((....))))))).((((.((((.(..(((((((((...(((((.....)))))))))).))))..).))))..)))).....))).....))).).. ( -45.50) >DroYak_CAF1 122208 120 - 1 CAGAUGGAUUUGGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGUGCUGGAGUCACUGCUUGAUCCGCUCACCUAUCACCAGCAU ..(((((....(((...(((((((....))))))).((((.((((.(..(((((((((...(((((.....)))))))))).))))..).))))..)))).)))...)))))........ ( -45.10) >consensus CAGAUGGAUUUGGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGUGCUGGAGUCACUGCUAGAUCCGCUCACCUAUCACCAGAAU ..(((((((((((((..(((((((....))))))).((.((.((((((((.((.....)).(((((.....))))))))))))).))))..))))))))))).))............... (-45.18 = -45.26 + 0.08)

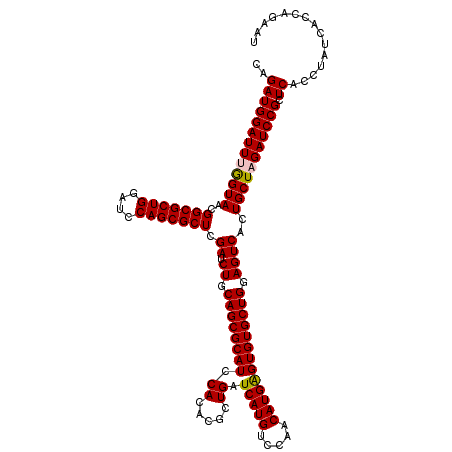
| Location | 9,815,211 – 9,815,331 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -40.64 |
| Consensus MFE | -40.22 |
| Energy contribution | -40.42 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.99 |
| SVM decision value | 2.62 |
| SVM RNA-class probability | 0.995856 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815211 120 + 23771897 ACACUCAUGUUAGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACUAAAUCCAUCUGGAAGUAGUCCAUCGUCUUGUCCAACACCUUCUUACAGCCC .......((((.((((.(((((((((......))))))..(((((.((((((....))))))))..((((..(((....)))..))))..)))))).)))).)))).............. ( -37.20) >DroSec_CAF1 109326 120 + 1 ACACCCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACCAAAUCCAUCUGGAAGUAGUCCAUCGUCUUGUCCAACACCUUCUUGCAGCCC .......(((((((((......(((.((((((..(((.(((((((.((((((....))))))))...........)))))..))).)))))))))..))))))))).............. ( -41.50) >DroSim_CAF1 112661 120 + 1 ACACUCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGGUCCAGCGCCGUUACCAAAUCCAUCUGGAAGUAGUCCAUCGUCUUGUCCAACACCUUCUUGCAGCCC .......(((((((((......(((.((((((..(((.(((((((.((((((....))))))))...........)))))..))).)))))))))..))))))))).............. ( -41.50) >DroEre_CAF1 112168 120 + 1 ACACUCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACCAAAUCCAUCUGGAAGUAGUCCAUCGUCUUGUCCAACACCUUCUUGCAGCCC .......(((((((((......(((.((((((..(((.(((((((.((((((....))))))))...........)))))..))).)))))))))..))))))))).............. ( -41.50) >DroYak_CAF1 122248 120 + 1 ACACUCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACCAAAUCCAUCUGGAAGUAGUCCAUCGUCUUGUCCAACACCUUCUUGCAGCCC .......(((((((((......(((.((((((..(((.(((((((.((((((....))))))))...........)))))..))).)))))))))..))))))))).............. ( -41.50) >consensus ACACUCAUGUUGGACAUGAUCAGCGUGUGGAUGCGCUGCAGAUCGAGCGCUGGAUCCAGCGCCGUUACCAAAUCCAUCUGGAAGUAGUCCAUCGUCUUGUCCAACACCUUCUUGCAGCCC .......(((((((((......(((.((((((..(((.(((((((.((((((....))))))))...........)))))..))).)))))))))..))))))))).............. (-40.22 = -40.42 + 0.20)


| Location | 9,815,211 – 9,815,331 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -44.48 |
| Consensus MFE | -44.72 |
| Energy contribution | -44.40 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.84 |
| Structure conservation index | 1.01 |
| SVM decision value | 2.55 |
| SVM RNA-class probability | 0.995200 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815211 120 - 23771897 GGGCUGUAAGAAGGUGUUGGACAAGACGAUGGACUACUUCCAGAUGGAUUUAGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCUAACAUGAGUGU .............((((((((((.((.(((.(((((..(((....)))..))))...(((((((....)))))))).)))..(((((........))))).)).))))))))))...... ( -42.80) >DroSec_CAF1 109326 120 - 1 GGGCUGCAAGAAGGUGUUGGACAAGACGAUGGACUACUUCCAGAUGGAUUUGGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGGGUGU .....(((.....((((((((((.((.(((.(((((..(((....)))..))))...(((((((....)))))))).)))..(((((........))))).)).))))))))))...))) ( -44.90) >DroSim_CAF1 112661 120 - 1 GGGCUGCAAGAAGGUGUUGGACAAGACGAUGGACUACUUCCAGAUGGAUUUGGUAACGGCGCUGGACCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGU .....(((.....((((((((((.((.(((.(((((..(((....)))..))))...(((((((....)))))))).)))..(((((........))))).)).))))))))))...))) ( -44.90) >DroEre_CAF1 112168 120 - 1 GGGCUGCAAGAAGGUGUUGGACAAGACGAUGGACUACUUCCAGAUGGAUUUGGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGU .....(((.....((((((((((.((.(((.(((((..(((....)))..))))...(((((((....)))))))).)))..(((((........))))).)).))))))))))...))) ( -44.90) >DroYak_CAF1 122248 120 - 1 GGGCUGCAAGAAGGUGUUGGACAAGACGAUGGACUACUUCCAGAUGGAUUUGGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGU .....(((.....((((((((((.((.(((.(((((..(((....)))..))))...(((((((....)))))))).)))..(((((........))))).)).))))))))))...))) ( -44.90) >consensus GGGCUGCAAGAAGGUGUUGGACAAGACGAUGGACUACUUCCAGAUGGAUUUGGUAACGGCGCUGGAUCCAGCGCUCGAUCUGCAGCGCAUCCACACGCUGAUCAUGUCCAACAUGAGUGU .............((((((((((.((.(((.(((((..(((....)))..))))...(((((((....)))))))).)))..(((((........))))).)).))))))))))...... (-44.72 = -44.40 + -0.32)


| Location | 9,815,331 – 9,815,451 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.50 |
| Mean single sequence MFE | -43.92 |
| Consensus MFE | -40.54 |
| Energy contribution | -41.58 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.49 |
| Structure conservation index | 0.92 |
| SVM decision value | 3.80 |
| SVM RNA-class probability | 0.999623 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815331 120 + 23771897 GGUGAAUUCUCAUCCGACCGGCCGCAGCAGCUGUAGAAUCCACUUACAAAGGCCGAGGGAUGGAAGUACUGCAUCCGGAUGGAGUUCAUCAGACAUAUCUUGUGCAGCAGGUCGAACCAC (((((((((.(((((...(((((((....))(((((.......)))))..)))))..(((((.(.....).)))))))))))))))))))........((((.....))))......... ( -41.90) >DroSec_CAF1 109446 120 + 1 GGUGAAUUCUCAUCCGACCGGCCGCAGCAGCUAUAGAAGCCACUGACAAAGGCCGAGGGAUGGAAGUACUGCAUCCGGAUGGAGUUCAUCAGACAGAUCUUGUGCAGCAAGUCGAACCAC (((((((((.(((((...(((((.(((..(((.....)))..))).....)))))..(((((.(.....).))))))))))))))))))).....((.((((.....))))))....... ( -46.70) >DroSim_CAF1 112781 120 + 1 GGUGAAUUCUCAUCCGACCGGCCGCAGCAGCUAUAGAAGCCACUGACAAAGGCCGAGGGAUGGAAGUACUGCAUCCGGAUGGAGUUCAUCAGACAGAUCUUGUGCAGCAAGUCGAACCAC (((((((((.(((((...(((((.(((..(((.....)))..))).....)))))..(((((.(.....).))))))))))))))))))).....((.((((.....))))))....... ( -46.70) >DroEre_CAF1 112288 120 + 1 GGCGAAUUCUCAUCCGACCGGCCGCAGCAGCUGUAGAAGCCACUGACAAAGGCCGAGGGAUGGAAGUACUGCAUCCGGAUGGAGUUCAUCAGACAGAUCUUGUGCAGCAAGUCAAACCAC ((.((((((.(((((...(((((.(((..(((.....)))..))).....)))))..(((((.(.....).))))))))))))))))........((.((((.....))))))...)).. ( -44.20) >DroYak_CAF1 122368 120 + 1 GGUGAAUUCUCAUCCGACCGGCCACAACAACUGUAGAAGCCACUGACAAAGGCCGAGGGAUGGAAGUACUGCAUCCGGAUGGAGUUCAUCAGACAGAUCUUGUGCAGCAAGUCGAACCAC (((((((((.(((((...((((((((.....)))((......))......)))))..(((((.(.....).))))))))))))))))))).....((.((((.....))))))....... ( -40.10) >consensus GGUGAAUUCUCAUCCGACCGGCCGCAGCAGCUGUAGAAGCCACUGACAAAGGCCGAGGGAUGGAAGUACUGCAUCCGGAUGGAGUUCAUCAGACAGAUCUUGUGCAGCAAGUCGAACCAC (((((((((.(((((...(((((.(((..(((.....)))..))).....)))))..(((((.(.....).))))))))))))))))))).....((.((((.....))))))....... (-40.54 = -41.58 + 1.04)

| Location | 9,815,331 – 9,815,451 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.50 |
| Mean single sequence MFE | -42.92 |
| Consensus MFE | -41.10 |
| Energy contribution | -41.06 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.84 |
| SVM RNA-class probability | 0.997327 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815331 120 - 23771897 GUGGUUCGACCUGCUGCACAAGAUAUGUCUGAUGAACUCCAUCCGGAUGCAGUACUUCCAUCCCUCGGCCUUUGUAAGUGGAUUCUACAGCUGCUGCGGCCGGUCGGAUGAGAAUUCACC (((((((((..(((((((..(((....)))((((.....))))....)))))))..))(((((.((((((.......((((...)))).((....))))))))..))))).))).)))). ( -38.90) >DroSec_CAF1 109446 120 - 1 GUGGUUCGACUUGCUGCACAAGAUCUGUCUGAUGAACUCCAUCCGGAUGCAGUACUUCCAUCCCUCGGCCUUUGUCAGUGGCUUCUAUAGCUGCUGCGGCCGGUCGGAUGAGAAUUCACC (((((((((..(((((((.....((((...((((.....)))))))))))))))..))(((((.((((((...(.(((..(((.....)))..))))))))))..))))).))).)))). ( -45.70) >DroSim_CAF1 112781 120 - 1 GUGGUUCGACUUGCUGCACAAGAUCUGUCUGAUGAACUCCAUCCGGAUGCAGUACUUCCAUCCCUCGGCCUUUGUCAGUGGCUUCUAUAGCUGCUGCGGCCGGUCGGAUGAGAAUUCACC (((((((((..(((((((.....((((...((((.....)))))))))))))))..))(((((.((((((...(.(((..(((.....)))..))))))))))..))))).))).)))). ( -45.70) >DroEre_CAF1 112288 120 - 1 GUGGUUUGACUUGCUGCACAAGAUCUGUCUGAUGAACUCCAUCCGGAUGCAGUACUUCCAUCCCUCGGCCUUUGUCAGUGGCUUCUACAGCUGCUGCGGCCGGUCGGAUGAGAAUUCGCC ((((.......(((((((.....((((...((((.....))))))))))))))).((((((((.((((((...(.(((..(((.....)))..))))))))))..))))).))).)))). ( -42.60) >DroYak_CAF1 122368 120 - 1 GUGGUUCGACUUGCUGCACAAGAUCUGUCUGAUGAACUCCAUCCGGAUGCAGUACUUCCAUCCCUCGGCCUUUGUCAGUGGCUUCUACAGUUGUUGUGGCCGGUCGGAUGAGAAUUCACC (((((((((..(((((((.....((((...((((.....)))))))))))))))..))(((((.((((((.....(((..(((.....)))..))).))))))..))))).))).)))). ( -41.70) >consensus GUGGUUCGACUUGCUGCACAAGAUCUGUCUGAUGAACUCCAUCCGGAUGCAGUACUUCCAUCCCUCGGCCUUUGUCAGUGGCUUCUACAGCUGCUGCGGCCGGUCGGAUGAGAAUUCACC (((((((((..(((((((.....((((...((((.....)))))))))))))))..))(((((.((((((.....(((..(((.....)))..))).))))))..))))).))).)))). (-41.10 = -41.06 + -0.04)


| Location | 9,815,451 – 9,815,571 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 99.33 |
| Mean single sequence MFE | -37.78 |
| Consensus MFE | -37.70 |
| Energy contribution | -37.38 |
| Covariance contribution | -0.32 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.44 |
| Structure conservation index | 1.00 |
| SVM decision value | 3.45 |
| SVM RNA-class probability | 0.999229 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815451 120 + 23771897 UCGCGCUCCUCCACGCAGUUCGUUGUCUGCACAAUCAGCGAGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCU ..(((........))).((((((((..........))))))))....(((((((((((...((((((.((((.((....))...))))..))))))..)))))))..))))......... ( -38.20) >DroSec_CAF1 109566 120 + 1 UCGCGCUCCUCCACGCAGUUCGUUGUCUGCACAAUCAGCGAGCGAUCGUUGUGUACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCU ..((((..((((.(((((...(((((..(((((....((((....)))))))))))))))))))))))..(((((..(((.....))).)))))......))))................ ( -36.40) >DroSim_CAF1 112901 120 + 1 UCGCGCUCCUCCACGCAGUUCGUUGUCUGCACAAUCAGCGAGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCU ..(((........))).((((((((..........))))))))....(((((((((((...((((((.((((.((....))...))))..))))))..)))))))..))))......... ( -38.20) >DroEre_CAF1 112408 120 + 1 UCGCGCUCCUCCACGCAGUUCGUUGUCUGCACAAUCAGCGAGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCU ..(((........))).((((((((..........))))))))....(((((((((((...((((((.((((.((....))...))))..))))))..)))))))..))))......... ( -38.20) >DroYak_CAF1 122488 120 + 1 UCGCGCUCCUCCACGCAGUUUGUUGUCUGCACAAUCAGCGAGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCU ..((((..((((.(((((.((((.....(((((....((((....))))))))))))).)))))))))..(((((..(((.....))).)))))......))))................ ( -37.90) >consensus UCGCGCUCCUCCACGCAGUUCGUUGUCUGCACAAUCAGCGAGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCU ..(((........))).((((((((..........))))))))....(((((((((((...((((((.((((.((....))...))))..))))))..)))))))..))))......... (-37.70 = -37.38 + -0.32)

| Location | 9,815,491 – 9,815,611 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 99.67 |
| Mean single sequence MFE | -39.00 |
| Consensus MFE | -39.16 |
| Energy contribution | -39.00 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.44 |
| Structure conservation index | 1.00 |
| SVM decision value | 4.06 |
| SVM RNA-class probability | 0.999779 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815491 120 + 23771897 AGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCUGUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAA .((((((((((((((((....((((((.((((.((....))...))))..)))))).((((((.......))))))...)))))))))))..))))).......((((......)))).. ( -39.40) >DroSec_CAF1 109606 120 + 1 AGCGAUCGUUGUGUACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCUGUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAA .((((((((((((((((....((((((.((((.((....))...))))..)))))).((((((.......))))))...)))))))))))..))))).......((((......)))).. ( -37.40) >DroSim_CAF1 112941 120 + 1 AGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCUGUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAA .((((((((((((((((....((((((.((((.((....))...))))..)))))).((((((.......))))))...)))))))))))..))))).......((((......)))).. ( -39.40) >DroEre_CAF1 112448 120 + 1 AGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCUGUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAA .((((((((((((((((....((((((.((((.((....))...))))..)))))).((((((.......))))))...)))))))))))..))))).......((((......)))).. ( -39.40) >DroYak_CAF1 122528 120 + 1 AGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCUGUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAA .((((((((((((((((....((((((.((((.((....))...))))..)))))).((((((.......))))))...)))))))))))..))))).......((((......)))).. ( -39.40) >consensus AGCGAUCGUUGUGCACAAUCUGUGGGAGAAGAGGCGAAAGUAAAUCUUAGUCUCAUCUUUGUGCAAUUAACAUAAAUUCUGUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAA .((((((((((((((((....((((((.((((.((....))...))))..)))))).((((((.......))))))...)))))))))))..))))).......((((......)))).. (-39.16 = -39.00 + -0.16)

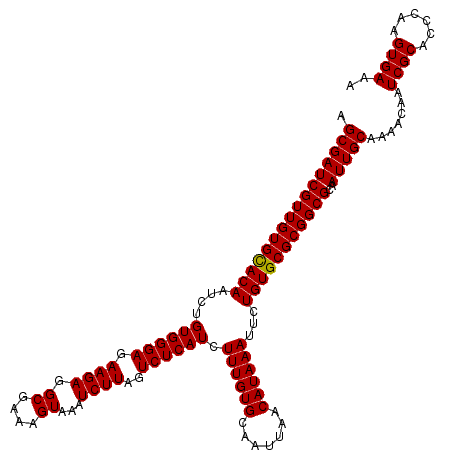

| Location | 9,815,531 – 9,815,651 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.83 |
| Mean single sequence MFE | -33.02 |
| Consensus MFE | -31.00 |
| Energy contribution | -30.60 |
| Covariance contribution | -0.40 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.31 |
| Structure conservation index | 0.94 |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.644650 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815531 120 - 23771897 UUUAACGCAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCACAGAAUUUAUGUUAAUUGCACAAAGAUGAGACUAAGAUUUA .((..((((.......))))..))..((((((....(((....).....((((((((((.....))))))))))))(((...(((....)))...))).......))))))......... ( -31.30) >DroSec_CAF1 109646 120 - 1 UUUAUCACAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCACAGAAUUUAUGUUAAUUGCACAAAGAUGAGACUAAGAUUUA ((((((...((((((.....((((((((........)))))))).))))((((((((((.....))))))))))))(((...(((....)))...))).....))))))........... ( -32.60) >DroSim_CAF1 112981 120 - 1 UUUAUCACAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCACAGAAUUUAUGUUAAUUGCACAAAGAUGAGACUAAGAUUUA ((((((...((((((.....((((((((........)))))))).))))((((((((((.....))))))))))))(((...(((....)))...))).....))))))........... ( -32.60) >DroEre_CAF1 112488 120 - 1 UUUAUCGCAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCACAGAAUUUAUGUUAAUUGCACAAAGAUGAGACUAAGAUUUA ((((((((...((((.....((((((((........)))))))).))))((((((((((.....))))))))))))(((...(((....)))...))).....))))))........... ( -33.60) >DroYak_CAF1 122568 120 - 1 UUUAUCGCAGCCAAGAUGUGGGAAUUGUCUCAGCCAGUGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCACAGAAUUUAUGUUAAUUGCACAAAGAUGAGACUAAGAUUUA ....(((((.......)))))(((((((((((((.((((....))))..((((((((((.....))))))))))))(((...(((....)))...))).......))))))...))))). ( -35.00) >consensus UUUAUCGCAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCACAGAAUUUAUGUUAAUUGCACAAAGAUGAGACUAAGAUUUA .....((((.......)))).(((((((((((....(((....).....((((((((((.....))))))))))))(((...(((....)))...))).......))))))...))))). (-31.00 = -30.60 + -0.40)

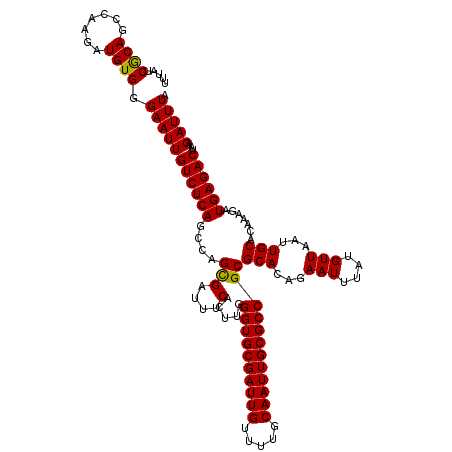
| Location | 9,815,571 – 9,815,681 |
|---|---|
| Length | 110 |
| Sequences | 4 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 93.39 |
| Mean single sequence MFE | -25.48 |
| Consensus MFE | -23.46 |
| Energy contribution | -23.28 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.92 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.730093 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815571 110 + 23771897 GUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAAUCGCUGGCUGAGACAAUUCCCACAUCUUGGCUGCGUUAAAAA-AU------AAGAAAACGUAACGCCAAGAAAUCGC ..(((((((((.((((.....)))).)).....((((....)))).))))..............(((((((((((((.....-..------.....))))))..)))))))...))) ( -28.20) >DroSec_CAF1 109686 110 + 1 GUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAAUCGCUGGCUGAGACAAUUCCCACAUCUUGGCUGUGAUAAAAA-AA------AAUAAAACGUAACGCCAAGAAAUCGC ......((((...((((.......((((......)))).(((((.(((..(((...........)))..)))))))).....-..------........)))))))).......... ( -24.70) >DroSim_CAF1 113021 111 + 1 GUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAAUCGCUGGCUGAGACAAUUCCCACAUCUUGGCUGUGAUAAAAAAAA------AAUAAAACCUAACGCCAAGAAAUCGC ..(((((((((.((((.....)))).)).....((((....)))).))))..............(((((((.((...........------...........)))))))))...))) ( -23.55) >DroYak_CAF1 122608 116 + 1 GUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAAUCACUGGCUGAGACAAUUCCCACAUCUUGGCUGCGAUAAAAA-AAAUAAACUAAAAAACGUAACGCCAAGAAAUCGC ..(((((((((.((((.....)))).)).....((((....)))).))))..............(((((((((((.......-...............))))..)))))))...))) ( -25.45) >consensus GUGCGCGGCGCAAUUGCAAAACAAUCGCACCCAAGUGAAAUCGCUGGCUGAGACAAUUCCCACAUCUUGGCUGCGAUAAAAA_AA______AAUAAAACGUAACGCCAAGAAAUCGC ......(((((....))......(((((.....((((....))))(((..(((...........)))..))))))))...........................))).......... (-23.46 = -23.28 + -0.19)



| Location | 9,815,571 – 9,815,681 |
|---|---|
| Length | 110 |
| Sequences | 4 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 93.39 |
| Mean single sequence MFE | -35.88 |
| Consensus MFE | -33.52 |
| Energy contribution | -33.34 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.20 |
| SVM RNA-class probability | 0.990188 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9815571 110 - 23771897 GCGAUUUCUUGGCGUUACGUUUUCUU------AU-UUUUUAACGCAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCAC (((...(((((((....((((.....------..-.....))))..)))))))....((((((((........)))))))).....((((((((((.....)))))))))).))).. ( -38.00) >DroSec_CAF1 109686 110 - 1 GCGAUUUCUUGGCGUUACGUUUUAUU------UU-UUUUUAUCACAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCAC (((...(((((((.............------..-...........)))))))....((((((((........)))))))).....((((((((((.....)))))))))).))).. ( -34.97) >DroSim_CAF1 113021 111 - 1 GCGAUUUCUUGGCGUUAGGUUUUAUU------UUUUUUUUAUCACAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCAC (((...(((((((....(((......------........)))...)))))))....((((((((........)))))))).....((((((((((.....)))))))))).))).. ( -35.54) >DroYak_CAF1 122608 116 - 1 GCGAUUUCUUGGCGUUACGUUUUUUAGUUUAUUU-UUUUUAUCGCAGCCAAGAUGUGGGAAUUGUCUCAGCCAGUGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCAC (((...(((((((....((......((......)-)......))..)))))))..(((((....))))).(.((((....)))).)((((((((((.....)))))))))).))).. ( -35.00) >consensus GCGAUUUCUUGGCGUUACGUUUUAUU______UU_UUUUUAUCACAGCCAAGAUGUGGGAAUUGUCUCAGCCAGCGAUUUCACUUGGGUGCGAUUGUUUUGCAAUUGCGCCGCGCAC (((...(((((((.................................)))))))....((((((((........)))))))).....((((((((((.....)))))))))).))).. (-33.52 = -33.34 + -0.19)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:28 2006