
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 9,807,370 – 9,807,486 |
| Length | 116 |
| Max. P | 0.993721 |

| Location | 9,807,370 – 9,807,461 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 83.90 |
| Mean single sequence MFE | -25.00 |
| Consensus MFE | -17.56 |
| Energy contribution | -17.42 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.78 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.50 |
| SVM RNA-class probability | 0.761428 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9807370 91 + 23771897 UGGAUAACGAA-------------AA-UCUCCAUGCACCCAUACUAUAUACACAUAUU-UGGAGCAUGUGCAUAUAGUAUGGCUUAGAUUGGAGA---CCAGA-ACUAUA ...........-------------..-((((((.....(((((((((((.(((((...-......))))).))))))))))).......))))))---.....-...... ( -26.10) >DroPse_CAF1 98963 108 + 1 UGGAUAACGAAGGAGGAAGAGAUGAU-UCUCUAUGUACCCAUACUAUAUACACAUAUUUUGGAGCAUGUGCAUAUAGUAUGGCUAAGAUAGCAGAAGUUGAGA-ACUAUA ....((((...(((.(.(((((....-))))).).).))((((((((((.(((((..........))))).))))))))))((((...))))....))))...-...... ( -25.40) >DroEre_CAF1 103852 91 + 1 UGGAUAACGAA-------------AU-UCUCCACGUACCCAUACUAUAUACACAUAUU-UGGAGCAUGUGCAUAUAGUAUGGCUAAGAUAGGAGA---CCAUA-ACUAUA ...........-------------..-(((((...((.(((((((((((.(((((...-......))))).))))))))))).)).....)))))---.....-...... ( -24.90) >DroYak_CAF1 114517 91 + 1 UGGAUAACGAA-------------AA-UCUCCAUGUACCCAUACUAUAUACACAUAUU-UGGAGCAUGUGCAUAUAGUAUGGCCUAGAUAGGAGA---CCAGA-ACUAUA ...........-------------..-(((((((.((.(((((((((((.(((((...-......))))).)))))))))))..)).)).)))))---.....-...... ( -25.50) >DroAna_CAF1 109087 93 + 1 UGGAUAACGAA-------------AUGGCCCUAUGUACCUAUACUAUAUACACAUAUU-UGGAGCAUGUGCAUAUAGUAUGGCUAAGAUGGCGGA---UCAGAAACUAUA ...........-------------.(((((((((.((.(((((((((((.(((((...-......))))).))))))))))).))..)))).)).---)))......... ( -22.70) >DroPer_CAF1 100050 108 + 1 UGGAUAACGAAGGAGGAAGAGAUGAU-UCUCUAUGUACCCAUACUAUAUACACAUAUUUUGGAGCAUGUGCAUAUAGUAUGGCUAAGAUAGCAGAAGUUGAGA-ACUAUA ....((((...(((.(.(((((....-))))).).).))((((((((((.(((((..........))))).))))))))))((((...))))....))))...-...... ( -25.40) >consensus UGGAUAACGAA_____________AU_UCUCCAUGUACCCAUACUAUAUACACAUAUU_UGGAGCAUGUGCAUAUAGUAUGGCUAAGAUAGCAGA___CCAGA_ACUAUA ......................................(((((((((((.(((((..........))))).)))))))))))............................ (-17.56 = -17.42 + -0.14)


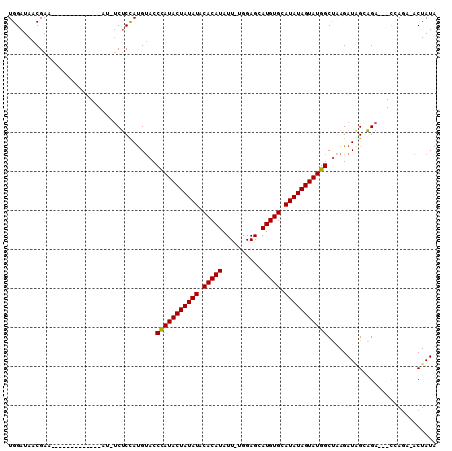
| Location | 9,807,370 – 9,807,461 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 83.90 |
| Mean single sequence MFE | -31.62 |
| Consensus MFE | -25.91 |
| Energy contribution | -25.92 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.15 |
| Mean z-score | -5.60 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.97 |
| SVM RNA-class probability | 0.984435 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9807370 91 - 23771897 UAUAGU-UCUGG---UCUCCAAUCUAAGCCAUACUAUAUGCACAUGCUCCA-AAUAUGUGUAUAUAGUAUGGGUGCAUGGAGA-UU-------------UUCGUUAUCCA ......-...((---((((((.....(.((((((((((((((((((.....-..)))))))))))))))))).)...))))))-))-------------........... ( -34.50) >DroPse_CAF1 98963 108 - 1 UAUAGU-UCUCAACUUCUGCUAUCUUAGCCAUACUAUAUGCACAUGCUCCAAAAUAUGUGUAUAUAGUAUGGGUACAUAGAGA-AUCAUCUCUUCCUCCUUCGUUAUCCA ......-.......((((.((((..((.((((((((((((((((((........)))))))))))))))))).)).)))))))-)......................... ( -30.70) >DroEre_CAF1 103852 91 - 1 UAUAGU-UAUGG---UCUCCUAUCUUAGCCAUACUAUAUGCACAUGCUCCA-AAUAUGUGUAUAUAGUAUGGGUACGUGGAGA-AU-------------UUCGUUAUCCA ......-.....---(((((...(.((.((((((((((((((((((.....-..)))))))))))))))))).)).).)))))-..-------------........... ( -33.70) >DroYak_CAF1 114517 91 - 1 UAUAGU-UCUGG---UCUCCUAUCUAGGCCAUACUAUAUGCACAUGCUCCA-AAUAUGUGUAUAUAGUAUGGGUACAUGGAGA-UU-------------UUCGUUAUCCA ......-...((---(((((......(.((((((((((((((((((.....-..)))))))))))))))))).)....)))))-))-------------........... ( -32.50) >DroAna_CAF1 109087 93 - 1 UAUAGUUUCUGA---UCCGCCAUCUUAGCCAUACUAUAUGCACAUGCUCCA-AAUAUGUGUAUAUAGUAUAGGUACAUAGGGCCAU-------------UUCGUUAUCCA ..........((---...(((......(((((((((((((((((((.....-..)))))))))))))))).)))......)))...-------------.))........ ( -27.60) >DroPer_CAF1 100050 108 - 1 UAUAGU-UCUCAACUUCUGCUAUCUUAGCCAUACUAUAUGCACAUGCUCCAAAAUAUGUGUAUAUAGUAUGGGUACAUAGAGA-AUCAUCUCUUCCUCCUUCGUUAUCCA ......-.......((((.((((..((.((((((((((((((((((........)))))))))))))))))).)).)))))))-)......................... ( -30.70) >consensus UAUAGU_UCUGA___UCUCCUAUCUUAGCCAUACUAUAUGCACAUGCUCCA_AAUAUGUGUAUAUAGUAUGGGUACAUAGAGA_AU_____________UUCGUUAUCCA ...............(((.((((...(.((((((((((((((((((........)))))))))))))))))).)..)))))))........................... (-25.91 = -25.92 + 0.00)


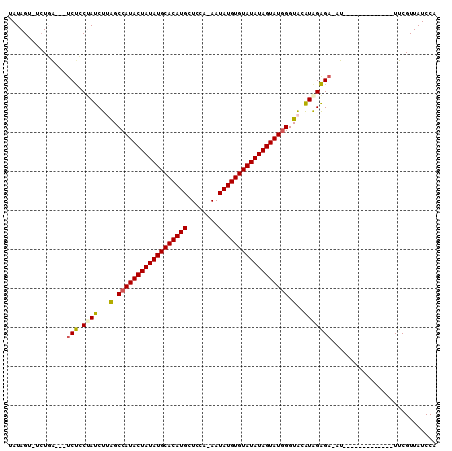
| Location | 9,807,395 – 9,807,486 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | forward |
| Mean pairwise identity | 86.33 |
| Mean single sequence MFE | -23.41 |
| Consensus MFE | -16.09 |
| Energy contribution | -16.28 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.13 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.540304 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9807395 91 + 23771897 CAUACUAUAUACACAUAUU-UGGAGCAUGUGCAUAUAGUAUGGCUUAGAUUGGAGA---CCAGA-ACUAUAUGCUAUAUGCUAUAUAGUGCUAGAU ..((((((((...((....-)).((((((((((((((((.((((((......))).---)))..-))))))))).)))))))))))))))...... ( -31.30) >DroPse_CAF1 99001 93 + 1 CAUACUAUAUACACAUAUUUUGGAGCAUGUGCAUAUAGUAUGGCUAAGAUAGCAGAAGUUGAGA-ACUAUAUAGUAAA--CCAUAUAGCGAUAGAU ((((((((((.(((((..........))))).))))))))))((((...))))....((((...-.((((((.(....--).)))))))))).... ( -20.50) >DroEre_CAF1 103877 89 + 1 CAUACUAUAUACACAUAUU-UGGAGCAUGUGCAUAUAGUAUGGCUAAGAUAGGAGA---CCAUA-ACUAUAUGCCAAG--CCAUAUAGUGCUAGAU ..((((((((...((....-))(.((.((.(((((((((((((((........)).---)))).-))))))))))).)--))))))))))...... ( -26.10) >DroYak_CAF1 114542 89 + 1 CAUACUAUAUACACAUAUU-UGGAGCAUGUGCAUAUAGUAUGGCCUAGAUAGGAGA---CCAGA-ACUAUAUGCUAAA--UCAUAUAGUGUUAGAU ((((((((((.(((((...-......))))).))))))))))..((((...(....---)....-((((((((.....--.)))))))).)))).. ( -24.10) >DroAna_CAF1 109113 88 + 1 UAUACUAUAUACACAUAUU-UGGAGCAUGUGCAUAUAGUAUGGCUAAGAUGGCGGA---UCAGAAACUAUAUACUAAAU--C--AUAGCGUUAGAU ((((((((((.(((((...-......))))).))))))))))((((.(((......---..................))--)--.))))....... ( -17.96) >DroPer_CAF1 100088 93 + 1 CAUACUAUAUACACAUAUUUUGGAGCAUGUGCAUAUAGUAUGGCUAAGAUAGCAGAAGUUGAGA-ACUAUAUAGUAAA--CCAUAUAGCGAUAGAU ((((((((((.(((((..........))))).))))))))))((((...))))....((((...-.((((((.(....--).)))))))))).... ( -20.50) >consensus CAUACUAUAUACACAUAUU_UGGAGCAUGUGCAUAUAGUAUGGCUAAGAUAGCAGA___CCAGA_ACUAUAUACUAAA__CCAUAUAGCGAUAGAU ((((((((((.(((((..........))))).))))))))))........................((((((..........))))))........ (-16.09 = -16.28 + 0.20)

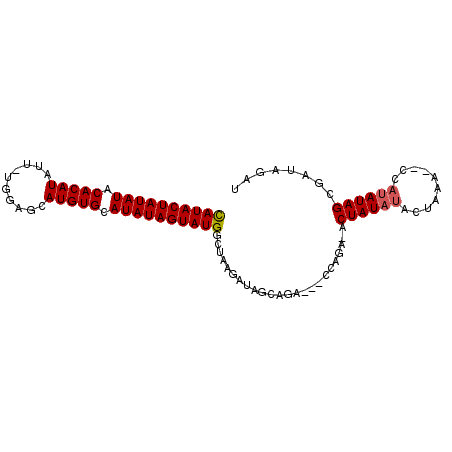
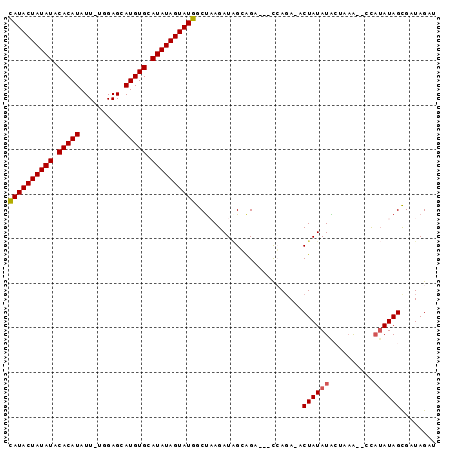
| Location | 9,807,395 – 9,807,486 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 96 |
| Reading direction | reverse |
| Mean pairwise identity | 86.33 |
| Mean single sequence MFE | -29.02 |
| Consensus MFE | -24.58 |
| Energy contribution | -24.83 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.08 |
| Mean z-score | -6.03 |
| Structure conservation index | 0.85 |
| SVM decision value | 2.42 |
| SVM RNA-class probability | 0.993721 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9807395 91 - 23771897 AUCUAGCACUAUAUAGCAUAUAGCAUAUAGU-UCUGG---UCUCCAAUCUAAGCCAUACUAUAUGCACAUGCUCCA-AAUAUGUGUAUAUAGUAUG ..((((.(((((((.((.....)))))))))-.))))---..............(((((((((((((((((.....-..))))))))))))))))) ( -31.60) >DroPse_CAF1 99001 93 - 1 AUCUAUCGCUAUAUGG--UUUACUAUAUAGU-UCUCAACUUCUGCUAUCUUAGCCAUACUAUAUGCACAUGCUCCAAAAUAUGUGUAUAUAGUAUG .......(((((((((--....)))))))))-...........((((...))))(((((((((((((((((........))))))))))))))))) ( -30.50) >DroEre_CAF1 103877 89 - 1 AUCUAGCACUAUAUGG--CUUGGCAUAUAGU-UAUGG---UCUCCUAUCUUAGCCAUACUAUAUGCACAUGCUCCA-AAUAUGUGUAUAUAGUAUG ..(((..((((((((.--.....))))))))-..)))---..............(((((((((((((((((.....-..))))))))))))))))) ( -28.60) >DroYak_CAF1 114542 89 - 1 AUCUAACACUAUAUGA--UUUAGCAUAUAGU-UCUGG---UCUCCUAUCUAGGCCAUACUAUAUGCACAUGCUCCA-AAUAUGUGUAUAUAGUAUG .......((((((((.--.....))))))))-...((---(((.......)))))((((((((((((((((.....-..)))))))))))))))). ( -30.40) >DroAna_CAF1 109113 88 - 1 AUCUAACGCUAU--G--AUUUAGUAUAUAGUUUCUGA---UCCGCCAUCUUAGCCAUACUAUAUGCACAUGCUCCA-AAUAUGUGUAUAUAGUAUA .......((((.--(--((((((..........))))---......))).)))).((((((((((((((((.....-..)))))))))))))))). ( -22.50) >DroPer_CAF1 100088 93 - 1 AUCUAUCGCUAUAUGG--UUUACUAUAUAGU-UCUCAACUUCUGCUAUCUUAGCCAUACUAUAUGCACAUGCUCCAAAAUAUGUGUAUAUAGUAUG .......(((((((((--....)))))))))-...........((((...))))(((((((((((((((((........))))))))))))))))) ( -30.50) >consensus AUCUAACACUAUAUGG__UUUAGCAUAUAGU_UCUGA___UCUCCUAUCUUAGCCAUACUAUAUGCACAUGCUCCA_AAUAUGUGUAUAUAGUAUG .......((((((((........)))))))).......................(((((((((((((((((........))))))))))))))))) (-24.58 = -24.83 + 0.25)

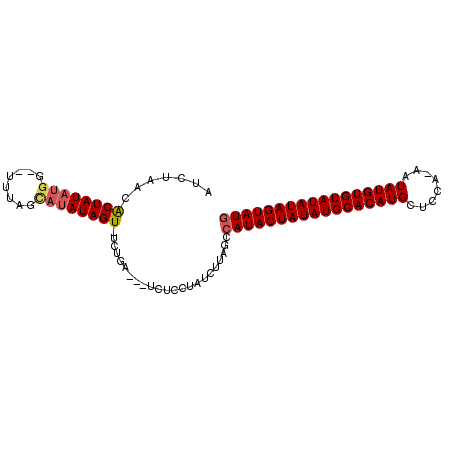
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:55:10 2006