
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 9,609,062 – 9,609,222 |
| Length | 160 |
| Max. P | 0.977943 |

| Location | 9,609,062 – 9,609,182 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.94 |
| Mean single sequence MFE | -44.27 |
| Consensus MFE | -38.07 |
| Energy contribution | -38.35 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.50 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.80 |
| SVM RNA-class probability | 0.977943 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
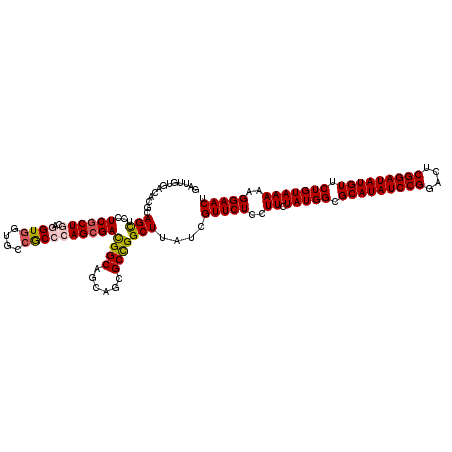
>3L_DroMel_CAF1 9609062 120 + 23771897 UUGCGAAACGUCGAACGUAAACUCCAUAGUGCGACCGGCCAGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAAGAGAACGAUAAGCCGGCGCUGCUGCCGUCGCU ..((((.(((.....)))........(((((((.(((((..(((((((((((..(..((((((((....))))))))..)..))))))).)))).....)))))))).))))...)))). ( -45.50) >DroVir_CAF1 4787 120 + 1 UUGGGAUACGUCGAAUGUGAAUUCCAUGGUGCGGCCGGCCAGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAGGAGAACGAUAAGCCGGCGCUGCUGCCAUCGCU .((((((((((...))))..))))))(((((((((((((..(((((((((((..(..((((((((....))))))))..)..))))))).)))).....))))))..))).))))..... ( -45.00) >DroGri_CAF1 4977 120 + 1 CUGCGAGACAUCGAAUGUGAACUCCAUGGUGCGGCCGGCUAGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAGGAGAACGAUAAGCCGGCGCUGCUGCCAUCGCU ..((((.((((...))))........((((((((((((((.(((((((((((..(..((((((((....))))))))..)..))))))).))))....)))))))..))).)))))))). ( -47.40) >DroSec_CAF1 4785 120 + 1 UUGUGAAACGUCGAACGUAAACUCCAUAGUGCGACCGGCCAGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAAGAGAACGAUAAGCCGGCGCUGCUGCCGUCGCU ..((((.(((.....)))........(((((((.(((((..(((((((((((..(..((((((((....))))))))..)..))))))).)))).....)))))))).))))...)))). ( -43.60) >DroWil_CAF1 5716 120 + 1 UUGACUCACAUCGAAUGUGAAUUCCAUGGUGCGUCCAGCCAGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAAGAGAACGAUAAGCCAGCGCUGCUGCCGUCGCU .....((((((...)))))).......((((((..((((..(((((((((((..(..((((((((....))))))))..)..))))))).))))....(((....))))))).)).)))) ( -37.00) >DroMoj_CAF1 4766 120 + 1 UUGCGACACGUCGAAGGUGAACUCCAUGGUGCGGCCGGCCAGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAGGAGAACGAUAAGCCGGCGCUGCUGCCAUCGCU ..(((((((.......))).......(((((((((((((..(((((((((((..(..((((((((....))))))))..)..))))))).)))).....))))))..))).)))))))). ( -47.10) >consensus UUGCGAAACGUCGAAUGUGAACUCCAUGGUGCGGCCGGCCAGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAAGAGAACGAUAAGCCGGCGCUGCUGCCAUCGCU ..((((.(((.....)))........(((((((.(((((..(((((((((((..(..((((((((....))))))))..)..))))))).)))).....)))))))).))))...)))). (-38.07 = -38.35 + 0.28)


| Location | 9,609,062 – 9,609,182 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.94 |
| Mean single sequence MFE | -46.22 |
| Consensus MFE | -38.80 |
| Energy contribution | -37.92 |
| Covariance contribution | -0.89 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967601 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9609062 120 - 23771897 AGCGACGGCAGCAGCGCCGGCUUAUCGUUCUCUUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACUGGCCGGUCGCACUAUGGAGUUUACGUUCGACGUUUCGCAA .((((((..(((.(((((((((....((((.((((.(((((.(((((((((....))))))))).))))).)))))))).)))))).)))(((....)))....)))...))..)))).. ( -47.90) >DroVir_CAF1 4787 120 - 1 AGCGAUGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACUGGCCGGCCGCACCAUGGAAUUCACAUUCGACGUAUCCCAA .(((((((..((.(.(((((((....(((((..((.(((((.(((((((((....))))))))).))))).)).))))).)))))))))).)))).((((....))))..)))....... ( -42.90) >DroGri_CAF1 4977 120 - 1 AGCGAUGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACUAGCCGGCCGCACCAUGGAGUUCACAUUCGAUGUCUCGCAG .((((.((((.(.(((((((((....(((((..((.(((((.(((((((((....))))))))).))))).)).))))).)))))).)))......((((....))))).)))))))).. ( -48.70) >DroSec_CAF1 4785 120 - 1 AGCGACGGCAGCAGCGCCGGCUUAUCGUUCUCUUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACUGGCCGGUCGCACUAUGGAGUUUACGUUCGACGUUUCACAA .((((((((......)))((((....((((.((((.(((((.(((((((((....))))))))).))))).)))))))).)))).)))))........((..(((.....)))...)).. ( -46.10) >DroWil_CAF1 5716 120 - 1 AGCGACGGCAGCAGCGCUGGCUUAUCGUUCUCUUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACUGGCUGGACGCACCAUGGAAUUCACAUUCGAUGUGAGUCAA ...(((..((...((((..(((....((((.((((.(((((.(((((((((....))))))))).))))).)))))))).)))..).)))..(((.((((....)))).))))).))).. ( -44.30) >DroMoj_CAF1 4766 120 - 1 AGCGAUGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACUGGCCGGCCGCACCAUGGAGUUCACCUUCGACGUGUCGCAA .(((((((..((.(.(((((((....(((((..((.(((((.(((((((((....))))))))).))))).)).))))).)))))))))).)).(((((.....)))))....))))).. ( -47.40) >consensus AGCGACGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACUGGCCGGCCGCACCAUGGAGUUCACAUUCGACGUGUCGCAA .(((.(((.....(((((((((....(((((..((.(((((.(((((((((....))))))))).)))))))..))))).)))))).)))...(((.......)))))).)))....... (-38.80 = -37.92 + -0.89)
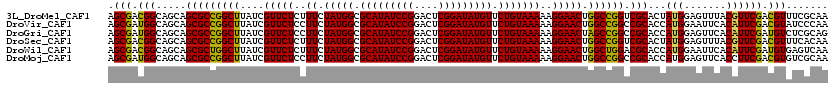
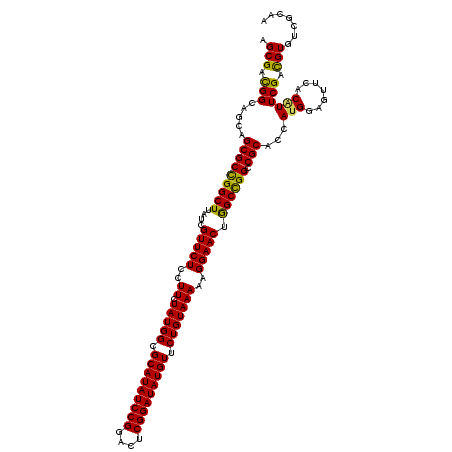
| Location | 9,609,102 – 9,609,222 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.78 |
| Mean single sequence MFE | -48.23 |
| Consensus MFE | -40.24 |
| Energy contribution | -40.05 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.47 |
| SVM RNA-class probability | 0.956868 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
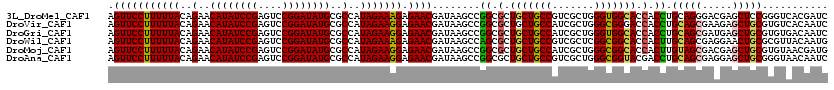
>3L_DroMel_CAF1 9609102 120 + 23771897 AGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAAGAGAACGAUAAGCCGGCGCUGCUGCCGUCGCUGGGUGGCACCACCUGCAGGGACGAGCUCCGGGUCACGAUC .(((((((((((..(..((((((((....))))))))..)..))))))).))))(((...((((.(((.....((((.(((((((....))))..))).))))))).)))))))...... ( -49.10) >DroVir_CAF1 4827 120 + 1 AGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAGGAGAACGAUAAGCCGGCGCUGCUGCCAUCGCUGGGCGGGACCACCUGCAGCGAAGAGCUGCGUGUCACAAUC .(((((((((((..(..((((((((....))))))))..)..))))))).))))........(((((.((.(((.(((((..(((((....)))))))))).).)).)))))))...... ( -47.70) >DroGri_CAF1 5017 120 + 1 AGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAGGAGAACGAUAAGCCGGCGCUGCUGCCAUCGCUGGGUGGCACCACCUGCAGCGAUGAGCUGCGUGUGACAAUC .(((((((((((..(..((((((((....))))))))..)..))))))).)))).........((((.((.((((((((((((((....))))..)))))))).)).))))))....... ( -50.90) >DroWil_CAF1 5756 120 + 1 AGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAAGAGAACGAUAAGCCAGCGCUGCUGCCGUCGCUCGGCGGCACCACUUGCAGCGAGGAACUGCGCGUUACAAUG .(((((((((((..(..((((((((....))))))))..)..))))))).)))).....((((((((((((((((((....)))))))......)))))).....))).))......... ( -47.10) >DroMoj_CAF1 4806 120 + 1 AGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAGGAGAACGAUAAGCCGGCGCUGCUGCCAUCGCUGGGCGGCACCACUUGUAGCGACGAGCUGCGUGUAACGAUG .(((((((((((..(..((((((((....))))))))..)..))))))).)))).........(((((((((((.......)))))))......(((((.....)))))))))....... ( -44.20) >DroAna_CAF1 7998 120 + 1 AGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAGGAGAACGAUAAGCCGGCGCUGCUGCCGUCGCUGGGCGGUACGACCUGCAGCGAGGAGCUGCGGGUAACAAUC .(((((((((((..(..((((((((....))))))))..)..))))))).))))..........((.(((((((.......)))))))))(((((((((.....)))))))))....... ( -50.40) >consensus AGUUCCUUUUUACAGAACAUAUCCGAGUCCGGAUAUGCGCCAUAGAAGGAGAACGAUAAGCCGGCGCUGCUGCCAUCGCUGGGCGGCACCACCUGCAGCGACGAGCUGCGUGUAACAAUC .(((((((((((..(..((((((((....))))))))..)..))))))).))))........((.(.(((((((.......))))))).).)).(((((.....)))))........... (-40.24 = -40.05 + -0.19)
| Location | 9,609,102 – 9,609,222 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.78 |
| Mean single sequence MFE | -46.41 |
| Consensus MFE | -37.76 |
| Energy contribution | -38.02 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.27 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.712713 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
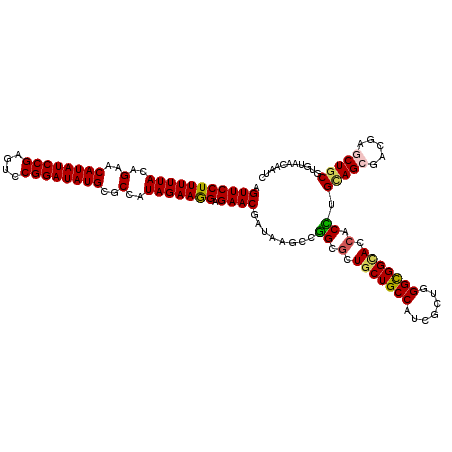
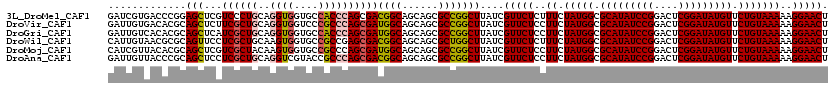
>3L_DroMel_CAF1 9609102 120 - 23771897 GAUCGUGACCCGGAGCUCGUCCCUGCAGGUGGUGCCACCCAGCGACGGCAGCAGCGCCGGCUUAUCGUUCUCUUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACU ....((((.((((.(((((((.(((..((((....))))))).)))).....))).)))).)))).((((.((((.(((((.(((((((((....))))))))).))))).)))))))). ( -49.30) >DroVir_CAF1 4827 120 - 1 GAUUGUGACACGCAGCUCUUCGCUGCAGGUGGUCCCGCCCAGCGAUGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACU (((.......(((.(((((((((((..((((....)))))))))).)).))).))).......)))....(((((.(((((.(((((((((....))))))))).)))))..)))))... ( -44.64) >DroGri_CAF1 5017 120 - 1 GAUUGUCACACGCAGCUCAUCGCUGCAGGUGGUGCCACCCAGCGAUGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACU (((.(((...(((.(((((((((((..((((....))))))))))))..))).)))..)))..)))....(((((.(((((.(((((((((....))))))))).)))))..)))))... ( -50.00) >DroWil_CAF1 5756 120 - 1 CAUUGUAACGCGCAGUUCCUCGCUGCAAGUGGUGCCGCCGAGCGACGGCAGCAGCGCUGGCUUAUCGUUCUCUUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACU ....((((.(((((((.....))))).((((.(((.((((.....)))).))).)))).)))))).((((.((((.(((((.(((((((((....))))))))).))))).)))))))). ( -49.90) >DroMoj_CAF1 4806 120 - 1 CAUCGUUACACGCAGCUCGUCGCUACAAGUGGUGCCGCCCAGCGAUGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACU ...........((.((.(((((((....(((....)))..))))))))).))(((....)))........(((((.(((((.(((((((((....))))))))).)))))..)))))... ( -39.70) >DroAna_CAF1 7998 120 - 1 GAUUGUUACCCGCAGCUCCUCGCUGCAGGUCGUACCGCCCAGCGACGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACU (((.(((...(((.(((((((((((..((.....))...)))))).)).))).)))..)))..)))....(((((.(((((.(((((((((....))))))))).)))))..)))))... ( -44.90) >consensus GAUUGUGACACGCAGCUCCUCGCUGCAGGUGGUGCCGCCCAGCGACGGCAGCAGCGCCGGCUUAUCGUUCUCCUUCUAUGGCGCAUAUCCGGACUCGGAUAUGUUCUGUAAAAAGGAACU .............(((...((((((..((((....))))))))))((((......)))))))....(((((..((.(((((.(((((((((....))))))))).)))))))..))))). (-37.76 = -38.02 + 0.25)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:53:37 2006