
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
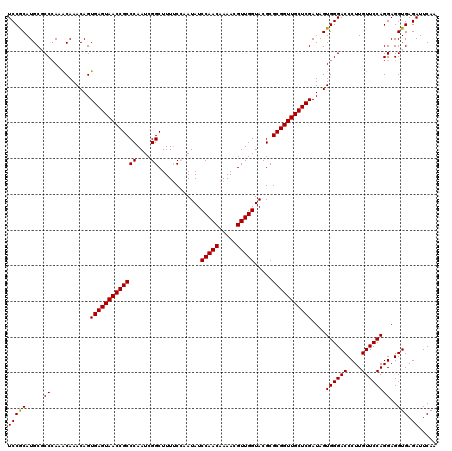
| Location | 9,125,420 – 9,125,641 |
| Length | 221 |
| Max. P | 0.996397 |

| Location | 9,125,420 – 9,125,540 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.67 |
| Mean single sequence MFE | -36.83 |
| Consensus MFE | -33.94 |
| Energy contribution | -33.72 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.22 |
| SVM RNA-class probability | 0.931716 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9125420 120 - 23771897 UCCGCAUGCGCCCAAGCAAACAGUGAGUAACCGCCCAAUCGGCUUUUCCAAUAUCCAACAAAACGUUGGUACGCGCGGUUGCUCGAUAGUGGGACCCUUGUUCCAGGAGGUGAGAUUCAA (((((.(((......))).....(((((((((((.(....((.....)).....(((((.....)))))...).)))))))))))...)))))(((((((...)))).)))......... ( -38.20) >DroPse_CAF1 527 120 - 1 UCCGCAUGCUCCCAAACAAACAGUGAGUAACCGCCCAAUCGGCUUUUCCAAUAUCCAACAAAACGUUGGUACGCGCGGUUGCUCGAUAGUGGGACCCUUGUUCCAGGAGGUGAGAUUCAA ..(.(((.((((...........(((((((((((.(....((.....)).....(((((.....)))))...).)))))))))))....((((((....))))))))))))).)...... ( -37.30) >DroWil_CAF1 453 120 - 1 UCCACAUGCGCCCAAACAAACAGUGAGUAACCGCCCAAUCGGCUUUUCAAAUAUCCAACAAAACGUUGGUACGCGCGGUUGCUCGAUAGUGGGACCCUUGUUCCAGGAGGUGAGAUUCAA ........(((((..........(((((((((((((....))............(((((.....))))).....)))))))))))....((((((....)))))).).))))........ ( -34.80) >DroMoj_CAF1 616 120 - 1 UCCGCAUGCGCCCAAACAAACAGUGAGUAACCGCCCAAUCGGCUUUACAAAUAUCCAACAAAACGUUGGUACGCGCGGUUGCUCGAUAGUGGGACCCUUGUUCCAGGAGGUGAGAUUCAA ........(((((..........(((((((((((((....))............(((((.....))))).....)))))))))))....((((((....)))))).).))))........ ( -34.80) >DroAna_CAF1 550 120 - 1 UCCACAUGCGCCAAAGCAAACAGUGAGUAACCGCCCAAUCGGCUUUUCCAAUAUCCAACAAAACGUUGGUACGCGCGGUUGCUCGAUAGUGGGACCCUUGUUCCAGGAGGUGAGAUUCAA (((((.(((......))).....(((((((((((.(....((.....)).....(((((.....)))))...).)))))))))))...)))))(((((((...)))).)))......... ( -38.60) >DroPer_CAF1 527 120 - 1 UCCGCAUGCUCCCAAACAAACAGUGAGUAACCGCCCAAUCGGCUUUUCCAAUAUCCAACAAAACGUUGGUACGCGCGGUUGCUCGAUAGUGGGACCCUUGUUCCAGGAGGUGAGAUUCAA ..(.(((.((((...........(((((((((((.(....((.....)).....(((((.....)))))...).)))))))))))....((((((....))))))))))))).)...... ( -37.30) >consensus UCCGCAUGCGCCCAAACAAACAGUGAGUAACCGCCCAAUCGGCUUUUCCAAUAUCCAACAAAACGUUGGUACGCGCGGUUGCUCGAUAGUGGGACCCUUGUUCCAGGAGGUGAGAUUCAA (((((.....((...........(((((((((((((....))............(((((.....))))).....)))))))))))....((((((....))))))))..))).))..... (-33.94 = -33.72 + -0.22)


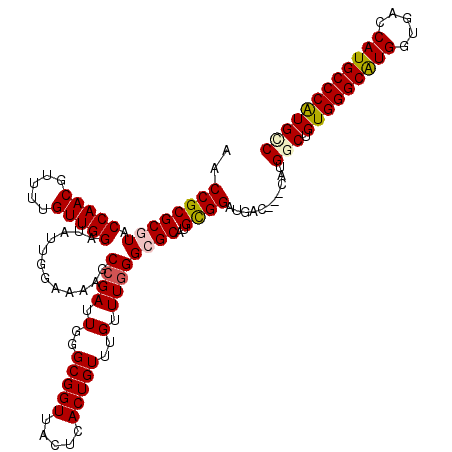
| Location | 9,125,460 – 9,125,577 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.50 |
| Mean single sequence MFE | -47.18 |
| Consensus MFE | -40.75 |
| Energy contribution | -40.83 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.53 |
| SVM RNA-class probability | 0.961551 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9125460 117 + 23771897 AACCGCGCGUACCAACGUUUUGUUGGAUAUUGGAAAAGCCGAUUGGGCGGUUACUCACUGUUUGCUUGGGCGCAUGCGGAUGUC---CAUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGC ..((((((((.(((((.....)))))............((((.(..(((((.....)))))..).))))))))..)))).....---....((..(((((((((...)))))))))..)) ( -48.30) >DroPse_CAF1 567 117 + 1 AACCGCGCGUACCAACGUUUUGUUGGAUAUUGGAAAAGCCGAUUGGGCGGUUACUCACUGUUUGUUUGGGAGCAUGCGGAUGAC---CAUGGCUGUGGGCAUGGUGACCAUGCCCAUGCC ((((((.((..(((((.....)))))..(((((.....))))))).))))))..(((((((.((((....)))).)))).))).---...(((.((((((((((...))))))))))))) ( -47.50) >DroWil_CAF1 493 117 + 1 AACCGCGCGUACCAACGUUUUGUUGGAUAUUUGAAAAGCCGAUUGGGCGGUUACUCACUGUUUGUUUGGGCGCAUGUGGAUGAC---CAUGGCUGUGGGCAUGAUGUCCAUGCCCAUGCC ....((((...(((((.....)))))............((((.(..(((((.....)))))..).))))))))..((((....)---)))(((.(((((((((.....)))))))))))) ( -45.70) >DroMoj_CAF1 656 117 + 1 AACCGCGCGUACCAACGUUUUGUUGGAUAUUUGUAAAGCCGAUUGGGCGGUUACUCACUGUUUGUUUGGGCGCAUGCGGAUGAC---CAUGGCUGUGGGCAUGAUGACCAUGCCCAUGUC ..((((((((.(((((.....)))))............((((.(..(((((.....)))))..).))))))))..)))).....---...(((.(((((((((.....)))))))))))) ( -44.20) >DroAna_CAF1 590 120 + 1 AACCGCGCGUACCAACGUUUUGUUGGAUAUUGGAAAAGCCGAUUGGGCGGUUACUCACUGUUUGCUUUGGCGCAUGUGGAUGCCCGCCGUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGC ..((((((...(((((.....)))))..........(((((....(((((.((..((((((..((....))))).)))..)).))))).)))))))((((((((...)))))))))))). ( -49.90) >DroPer_CAF1 567 117 + 1 AACCGCGCGUACCAACGUUUUGUUGGAUAUUGGAAAAGCCGAUUGGGCGGUUACUCACUGUUUGUUUGGGAGCAUGCGGAUGAC---CAUGGCUGUGGGCAUGGUGACCAUGCCCAUGCC ((((((.((..(((((.....)))))..(((((.....))))))).))))))..(((((((.((((....)))).)))).))).---...(((.((((((((((...))))))))))))) ( -47.50) >consensus AACCGCGCGUACCAACGUUUUGUUGGAUAUUGGAAAAGCCGAUUGGGCGGUUACUCACUGUUUGUUUGGGCGCAUGCGGAUGAC___CAUGGCUGUGGGCAUGGUGACCAUGCCCAUGCC ..((((((((.(((((.....)))))............((((.(..(((((.....)))))..).))))))))..))))...........(((.(((((((((.....)))))))))))) (-40.75 = -40.83 + 0.09)

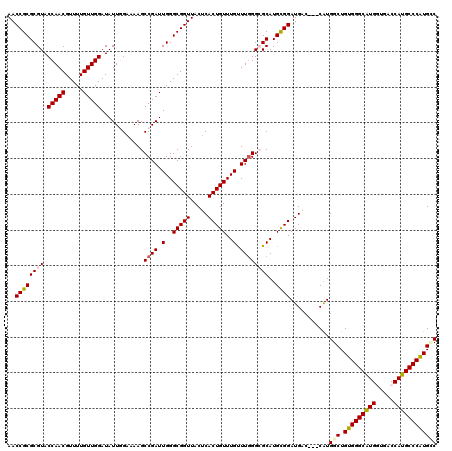
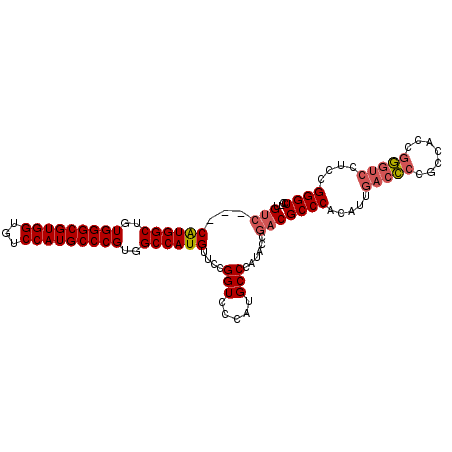
| Location | 9,125,540 – 9,125,641 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 93.29 |
| Mean single sequence MFE | -46.67 |
| Consensus MFE | -41.58 |
| Energy contribution | -42.17 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.69 |
| SVM RNA-class probability | 0.996397 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 9125540 101 + 23771897 UGUC---CAUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGCCAUGUUCCGGUCCCAUGCCCAUACCGACGCCCACAUUGACCCCGCCACCGGGUCCUCCGGGUCC- .(((---((((((..(((((((((...)))))))))..))))))....(((.....)))......)))((((.....(((((.......)))))....))))..- ( -46.30) >DroSec_CAF1 655 101 + 1 UGUC---CAUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGCCAUGUUCCGGUCCCAUGCCCAUACCGACGCCCACAUUGACCCCGCCACCGGGUCCUCCGGGUCC- .(((---((((((..(((((((((...)))))))))..))))))....(((.....)))......)))((((.....(((((.......)))))....))))..- ( -46.30) >DroSim_CAF1 655 101 + 1 UGUC---CAUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGCCAUGUUCCGGUCCCAUGCCCAUACCGACGCCCACAUUGACCCCGCCACCGGGUCCUCCGGGUCC- .(((---((((((..(((((((((...)))))))))..))))))....(((.....)))......)))((((.....(((((.......)))))....))))..- ( -46.30) >DroEre_CAF1 660 101 + 1 UGUC---CAUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGCCAUGUUCCGGUCCCAUGCCCAUACCGACGCCCACAUUGACCCCGCCACCGGGUCCACCGGGUCC- .(((---((((((..(((((((((...)))))))))..))))))....(((.....)))......)))((((.....(((((.......)))))....))))..- ( -46.30) >DroYak_CAF1 640 101 + 1 UGUC---CAUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGCCAUGCUCCGGUCCCAUGCCCAUACCGACGCCCACAUUGACCCCGCCACCGGGUCCUCCGGGUCC- .(((---((((((..(((((((((...)))))))))..))))))....(((.....)))......)))((((.....(((((.......)))))....))))..- ( -46.10) >DroAna_CAF1 670 102 + 1 UGCCCGCCGUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGCCAUGCUCCGGUCCCAUGCCCAUGCCCACGCCCACAUUGGGUCCGCCUCCCA---UUCCGGGUCCC .(((((..((((.(((((((((((.(..((((.(((..((...))..)))...)))))))))))))))).))))..((((.......))))---...)))))... ( -48.70) >consensus UGUC___CAUGGCUGUGGGCGUGGUGUCCAUGCCCGUGGCCAUGUUCCGGUCCCAUGCCCAUACCGACGCCCACAUUGACCCCGCCACCGGGUCCUCCGGGUCC_ .(((...((((((..(((((((((...)))))))))..))))))....(((.....)))......)))((((.....(((((.......)))))....))))... (-41.58 = -42.17 + 0.59)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:50:17 2006