
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 8,958,329 – 8,958,566 |
| Length | 237 |
| Max. P | 0.940729 |

| Location | 8,958,329 – 8,958,449 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.67 |
| Mean single sequence MFE | -49.58 |
| Consensus MFE | -25.27 |
| Energy contribution | -24.75 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.48 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.552325 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8958329 120 - 23771897 GGUCAGCUCCGCUGUGUUGGCCACCGCACACGUCCAGGGCAAGAAACUAGAUCGCAGGGAGCUGGCUUCCAUUUCGGCCAUGUGUGAGCAGUACAUUGGAACGCACAGUGGUGGUAUGGA ((((((((((.((((((((.((...((....))...)).)))..........)))))))))))))))(((((..(.((((((((((..(((....)))...)))))).)))).).))))) ( -45.60) >DroVir_CAF1 28131 120 - 1 GGUUAGCGCCGCUGUGCUGGCCACUGGACACGUGCAGGGCAUGCAGCUGGAUCGCAAGCAGCUAGCCUCGAUUUCAGCUAGCUGUGAGCAAUAUAUUGGCACACAUAGCGGCGGCAUGGA ......((((((((((.(((((((..(...(((((...)))))...)..)...((..(((((((((..........)))))))))..)).......)))).))))))))))))....... ( -52.80) >DroGri_CAF1 41011 120 - 1 AGUGAGUGCCGCUGUGCUGGCCACGGCCAAAGUGCAGGGCAUGAGGCUGGACCGCAAGCAGUUGGCAUCGAUUUCGGCCAGUUGUGAGCAGUACAUUGGCACACACAGCGGUGGCAUGGA .(((..((((((((((.(((((((((((...((((...))))..))))).((.((..(((..((((..((....))))))..)))..)).))....)))).)))))))))))).)))... ( -52.30) >DroWil_CAF1 28035 120 - 1 GGUCAGCUCUGCAGUAUUGGCCACAGCCAAUGUGCAGGGCAUGCAGUUGGAUCGUAAGGAGCUGGCCUCCAUAUCGGCCAGGUGUGAGCAAUAUAUUGGCACGCAUAGCGGAGGAAUGGA .....((((((((.(((((((....)))))))))))))))...(((((...((....))))))).(((((.((((((((((((((.....)))).))))).)).)))..)))))...... ( -47.70) >DroMoj_CAF1 17309 120 - 1 GGUCAGCGCCGCUGUACUGGCUACAGGACAUACUCAGGGCAUGCCGUUGGAUCGCAAGCAGCUGGCCUCGAUUUCGGCAAGCUGUGAGCAAUAUAUUGGCACCCACAGCGGUGGCAUGGA ..(((((((((((((.(((....)))..........(((..(((((....((.((..((((((.(((........))).))))))..)).))....)))))))))))))))).)).))). ( -49.60) >DroAna_CAF1 14894 120 - 1 GGUUAGCUCGGCAGUUCUGGCCACCGCCCAUGUCCAGGGCAUGCAGCUGGAUCGCCGGGAGCUGGCCUCUAUCUCCGCCAAGUGCGAGCAAUACAUUGGCACUCACAGUGGAGGCAUGGA (((((((((.((((((((((.((.......)).))))))).)))..((((....)))))))))))))(((((((((((..(((((.((.......)).)))))....))))))..))))) ( -49.50) >consensus GGUCAGCGCCGCUGUGCUGGCCACAGCACAUGUGCAGGGCAUGCAGCUGGAUCGCAAGCAGCUGGCCUCGAUUUCGGCCAGGUGUGAGCAAUACAUUGGCACACACAGCGGUGGCAUGGA ......(((((((((....((((..............(((.....))).....((..(.(.(((((..........))))).).)..)).......))))....)))))))))....... (-25.27 = -24.75 + -0.52)

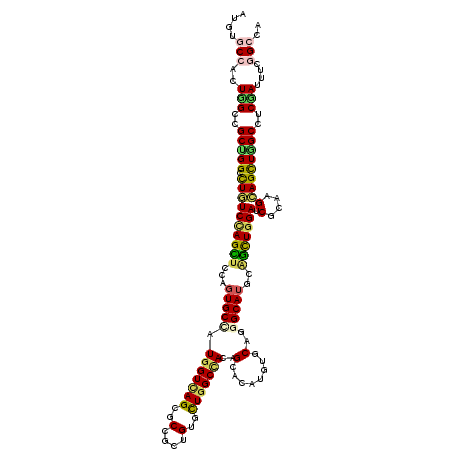
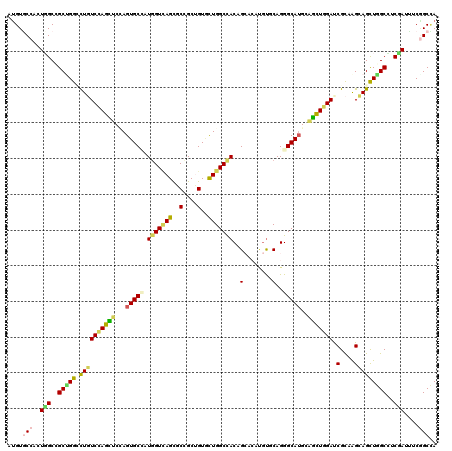
| Location | 8,958,369 – 8,958,489 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.44 |
| Mean single sequence MFE | -50.12 |
| Consensus MFE | -27.77 |
| Energy contribution | -27.42 |
| Covariance contribution | -0.35 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.608377 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8958369 120 - 23771897 AUGUGCCCUUGGCGGCAGGACUCUCCAGUUCGAGUGCCAUGGUCAGCUCCGCUGUGUUGGCCACCGCACACGUCCAGGGCAAGAAACUAGAUCGCAGGGAGCUGGCUUCCAUUUCGGCCA ...((((((.((((...(((((....)))))..((((..((((((((........))))))))..)))).)))).))))))........(..((..(((((....)))))....))..). ( -47.20) >DroVir_CAF1 28171 120 - 1 AUGUGCCACUUGCUGCUGGCUUGUCCAGCUCUAGUGCGUUGGUUAGCGCCGCUGUGCUGGCCACUGGACACGUGCAGGGCAUGCAGCUGGAUCGCAAGCAGCUAGCCUCGAUUUCAGCUA ...........((((((.((..((((((((...((((..((((((((((....))))))))))(((.(....).))).))))..)))))))).)).))))))((((..........)))) ( -49.80) >DroGri_CAF1 41051 120 - 1 AUGUGCCACUUGCCGCUGGCCUGUCCAGCUCUAGUGCGCUAGUGAGUGCCGCUGUGCUGGCCACGGCCAAAGUGCAGGGCAUGAGGCUGGACCGCAAGCAGUUGGCAUCGAUUUCGGCCA ..((((((...((.(((.((..((((((((((((....))))...(((((.(((..(((((....)))..))..))))))))..)))))))).)).))).))))))))((....)).... ( -53.70) >DroWil_CAF1 28075 120 - 1 AUGUUCCUCUGGCAGCUGGUCUCUCUAGCUCCAGUGCCAUGGUCAGCUCUGCAGUAUUGGCCACAGCCAAUGUGCAGGGCAUGCAGUUGGAUCGUAAGGAGCUGGCCUCCAUAUCGGCCA ..((((((((((.((((((.....))))))))))..(((..((..((((((((.(((((((....)))))))))))))))..))...)))......)))))).((((........)))). ( -55.50) >DroMoj_CAF1 17349 120 - 1 ACGUUCCUUUGGCCGCCGGACUGUCCAGUUCCAGUGCUUUGGUCAGCGCCGCUGUACUGGCUACAGGACAUACUCAGGGCAUGCCGUUGGAUCGCAAGCAGCUGGCCUCGAUUUCGGCAA .((.(((..((((.(((.((.(((((.((.(((((((..((((....))))..)))))))..)).)))))...))..)))..))))..))).))......(((((........))))).. ( -45.90) >DroAna_CAF1 14934 120 - 1 AUGUGCCAUUGGCCGCUGGCUUGUCCAGCUCGAGUGCCAUGGUUAGCUCGGCAGUUCUGGCCACCGCCCAUGUCCAGGGCAUGCAGCUGGAUCGCCGGGAGCUGGCCUCUAUCUCCGCCA .........((((...((((..(((((((((((((((....))..)))))((((((((((.((.......)).))))))).))))))))))).))))((((.(((...))).)))))))) ( -48.60) >consensus AUGUGCCACUGGCCGCUGGCCUGUCCAGCUCCAGUGCCAUGGUCAGCGCCGCUGUGCUGGCCACAGCACAUGUGCAGGGCAUGCAGCUGGAUCGCAAGCAGCUGGCCUCGAUUUCGGCCA ....(((..(((..(((((.((((((((((...(((((.(((((((..(....)..)))))))..(........)..)))))..))))))).(....)))))))))..)))....))).. (-27.77 = -27.42 + -0.35)

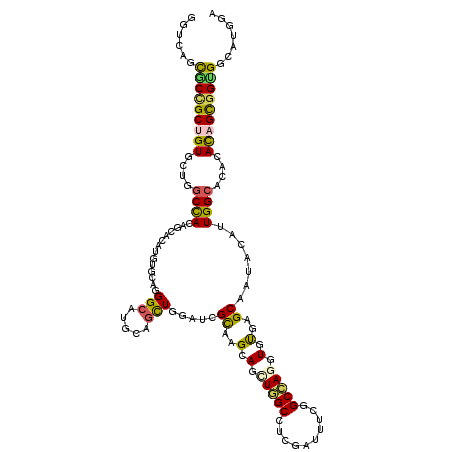
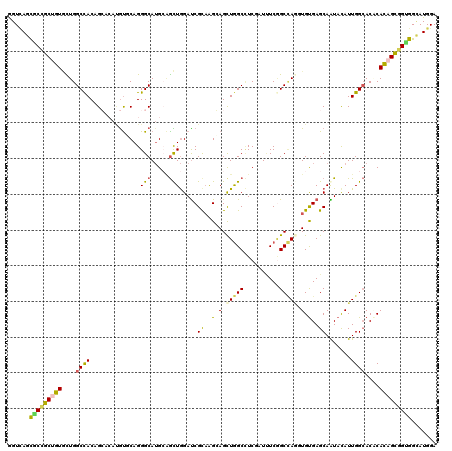
| Location | 8,958,449 – 8,958,566 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 74.70 |
| Mean single sequence MFE | -48.50 |
| Consensus MFE | -28.68 |
| Energy contribution | -31.05 |
| Covariance contribution | 2.37 |
| Combinations/Pair | 1.46 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.59 |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.940729 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8958449 117 - 23771897 GUGCGGCAUCAAAGGAAUACAGGAGAGUCUGGGCAGCCAGUGGAAGCCCAUUGGCAUGCGCAUUGCUGUGGACGGUAAUGUGCCCUUGGCGGCAGGACUCUCCAGUUCGAGUGCCAU ....(((((....(((.....(((((((((..((.(((((.((..(((....)))..(((((((((((....)))))))))))))))))).)).)))))))))..)))..))))).. ( -57.00) >DroVir_CAF1 28251 117 - 1 AUGCGGCAUCAAGGGCAUACAGGAGGAGCUGGAGAAGAAGUGGCGUCCCAUUGGCAUGCGCAUUGCACUUAGCGGCAAUGUGCCACUUGCUGCUGGCUUGUCCAGCUCUAGUGCGUU (((((((.......))........(((((((((.(((.((..(((((.....)))..(((((((((........))))))))).....))..))..))).)))))))))..))))). ( -48.00) >DroGri_CAF1 41131 117 - 1 GUGCGGCAUCAAGGGUGUACAGGAGGAGCUGGGCGAUAAGUGGCGGCCAGUUGGCAUGCGUGUUGCACUCUGUGGCAAUGUGCCACUUGCCGCUGGCCUGUCCAGCUCUAGUGCGCU (((((((((.....))))......(((((((((((........)(((((((.((((.(((..((((.(.....)))))..)))....))))))))))).))))))))))..))))). ( -55.80) >DroWil_CAF1 28155 117 - 1 CUGUGGCAUCAAGGGUGUCCAGGAACAUCUGGGGAGUAAGUGGCAGCCCCGGGGCAUGCGAAUUGCCGUUAUCGGCAAUGUUCCUCUGGCAGCUGGUCUCUCUAGCUCCAGUGCCAU ..((((((....((....))((((((.(((((((.((.....))..)))))))........(((((((....)))))))))))))((((.((((((.....)))))))))))))))) ( -49.90) >DroMoj_CAF1 17429 117 - 1 GUGUGGCAUCAAAGGCAUACAGGAAGAGCUGAAUGACAAGUGGCGCCCAAUUGGUAUGCGCAUUGCUCUCAGUGGCAACGUUCCUUUGGCCGCCGGACUGUCCAGUUCCAGUGCUUU (((((.(......).)))))..((((.((((..........((((.((((..((...(((..(((((......)))))))).)).)))).))))(((((....))))))))).)))) ( -37.70) >DroAna_CAF1 15014 117 - 1 GUGCGGCAUUAAGGGAGUCCAGGAACAGCUGGCCAGCAAGUGGAGCCCCAUUGGGAUGCGUAUCGCUGUAGACGGGAAUGUGCCAUUGGCCGCUGGCUUGUCCAGCUCGAGUGCCAU ....((((((..((..(..((((..((((.((((((..(((((....))))).((.(((((.((.(((....)))))))))))).)))))))))).))))..)..))..)))))).. ( -42.60) >consensus GUGCGGCAUCAAGGGAAUACAGGAAGAGCUGGGCAACAAGUGGCGGCCCAUUGGCAUGCGCAUUGCUCUCAGCGGCAAUGUGCCACUGGCCGCUGGCCUGUCCAGCUCCAGUGCCAU ....(((((....(.....)......((((((((((..((((((.(((....)))..((((((((((......)))))))))).....))))))...))))))))))...))))).. (-28.68 = -31.05 + 2.37)

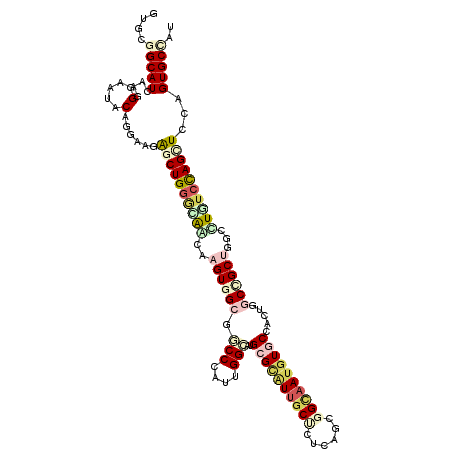
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:49:17 2006