
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 8,575,548 – 8,575,698 |
| Length | 150 |
| Max. P | 0.682950 |

| Location | 8,575,548 – 8,575,658 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 74.93 |
| Mean single sequence MFE | -26.34 |
| Consensus MFE | -17.82 |
| Energy contribution | -18.18 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.13 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.31 |
| SVM RNA-class probability | 0.682950 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8575548 110 - 23771897 GAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCUACCAGAACCUGAAA-AGUG--AAAAGCCAUUAAU-UAGUUUGGA-UCACUUUA---ACCAUG ((((((((((....)))))))))).(((((..((....)).))))).((.........(((...))).((-((((--(....(((.....-.....))).-))))))).---.))... ( -29.90) >DroVir_CAF1 302 110 - 1 GAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCAGUCAGCGGACCAUCAACCAAAACCUGCAA--UCA--AUAUGU--A-UAUAUAGUU-AAGUUCUAUUUAAUUUACUUU ((((((((((....)))))))))).(((((.(((....)))))))).((((....(((.......((((.--...--...)))--)-......)))-..))))............... ( -20.32) >DroEre_CAF1 297 111 - 1 GAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGGGGACCAUCAACCAGAACCUGCAG-AGUG--AUAAGCCAGUAAU-UAGUUUGGAUGCUCUAUA---CACAUU ((((((((((....))))))))))((((((..((....)).))))))(((.((((...(((...)))(((-(.((--((.........))-)).)))))))).)))...---...... ( -32.20) >DroWil_CAF1 309 102 - 1 GAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGAACACCGGUGAGAGGGCCAUCAACCAAAACCUGCGGUAAUG--CAAAAC---AAAU-UAGUAUUC-------AUA---UUUAUU ((((((((((....)))))))))).((.((.((.....)).)).)).((........))......((((....))--))....---....-........-------...---...... ( -21.90) >DroYak_CAF1 299 113 - 1 GGUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCGACCAGCACCUGAAG-AGUGAGAUAAGCCAGUAAU-UAGUUUUGGGGCUCUAUA---CACAUU ((((((((((....)))))...((((((((..((....)).))))).)))..........))))).....-.(((..(((((((..(((.-.....))).)))).))).---)))... ( -31.30) >DroMoj_CAF1 317 104 - 1 GAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCAGUCAGAGGUCCAUCAACCAAAACCUGUAA--GAA--AUAAAU--A-AAU-UAGUUCAACUGCAAUACAA------UU ((((((((((....)))))))))).(((((.(((....)))))))).(((......)))......((((.--(((--.(((..--.-..)-)).)))...))))......------.. ( -22.40) >consensus GAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGAACACCAGUCAGAGGACCAUCAACCAAAACCUGCAA_AGUG__AUAAGC__AUAAU_UAGUUUAGAUGCUCUAUA___CACAUU ((((((((((....)))))))))).(((((.((.....)).))))).((........))........................................................... (-17.82 = -18.18 + 0.36)

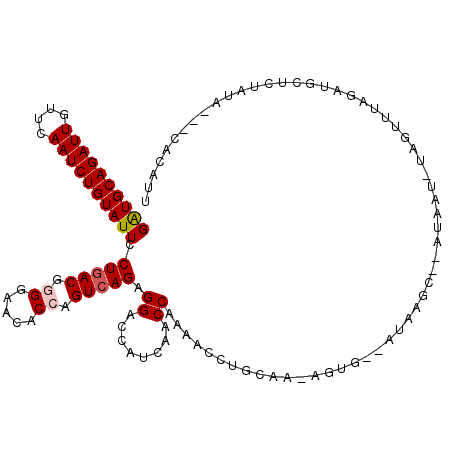

| Location | 8,575,581 – 8,575,698 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 84.93 |
| Mean single sequence MFE | -39.58 |
| Consensus MFE | -33.46 |
| Energy contribution | -34.35 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.599627 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
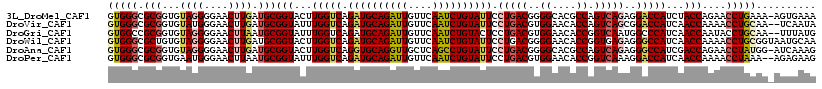
>3L_DroMel_CAF1 8575581 117 - 23771897 GUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGACCAUCUACCAGAACCUGAAA-AGUGAAA ..(((.(((...((((....)))).)))((((..(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))..))))....)))....-....... ( -43.40) >DroVir_CAF1 336 116 - 1 GUGGGCGCGGUGUAUGGGAACUUGAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCAGUCAGCGGACCAUCAACCAAAACCUGCAA--UCAAUA (..((.(((...((.(....).)).)))(((...(((((.((((((((((....))))))))))((((((.(((....)))))))).))))))...)))....))..)..--...... ( -39.60) >DroGri_CAF1 354 116 - 1 GUGGCCGCGGUGUAGGGGAACUUAAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUACUCCUGACGUGGAACACCGGUCAAUGGCCCAUCAACCAAUACCUGCAA--UUUAUG (((((((((...((((....)))).)))))).....(((((.((((((((....))))))))...)))))......))).(((...(((........)))..))).....--...... ( -35.30) >DroWil_CAF1 333 118 - 1 GUGGGCGCUGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGAACACCGGUGAGAGGGCCAUCAACCAAAACCUGCGGUAAUGCAA (..((.((....((((....))))..))(((...(((((.((((((((((....)))))))))).((.((.((.....)).)).))..)))))...)))....))..).......... ( -40.10) >DroAna_CAF1 316 117 - 1 GUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGGUGCAGGUUGCUCAGCCUGUAUUCCUGACGGGGCACGCCAGUCAGAGGGCCAUCGACCAGAACCUAUGG-AUCAAAG (((((.(((...((((....)))).)))(((...(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))...)))....)))))..-....... ( -45.00) >DroPer_CAF1 357 116 - 1 GUGGGCGCGGUGAAUGGGAACUUAAUGCGGUAUUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGUGGAACACCGGUCAAAGGACCAUCAACCAAAACCUAAA--AGAGAAG .((((...(((..((((...(((....((((.(((.((((((((((((((....)))))))))..)))))...))).))))....)))..))))..)))....))))..--....... ( -34.10) >consensus GUGGGCGCGGUGUAGGGGAACUUGAUGCGGUACUUGGUCAGAUGCAGAUUGUUCAAUCUGUAUUCCUGACGGGGAACACCAGUCAGAGGACCAUCAACCAAAACCUGCAA_AUUGAAA (((((.(((...((((....)))).)))(((...(((((.((((((((((....)))))))))).(((((..((....)).)))))..)))))...)))....))))).......... (-33.46 = -34.35 + 0.89)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:52 2006