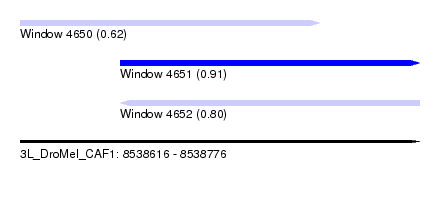
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 8,538,616 – 8,538,776 |
| Length | 160 |
| Max. P | 0.909540 |

| Location | 8,538,616 – 8,538,736 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.78 |
| Mean single sequence MFE | -50.57 |
| Consensus MFE | -38.49 |
| Energy contribution | -38.25 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.624741 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8538616 120 + 23771897 AUUGCCGCAAGCUGUGACGAUGAACAGGCCAGGAACCGUGUUAUUGGAGCUGCCAGUCAGUGUGCCCUGGCUACCAGUCAAUUGGUGGCUUGUGCCAAAGUUGUGGCACCAACGCUCCAC ..((((((((.((((........))))((((((...((..(((((((.....))))).))..)).)))))).(((((....)))))(((....)))....))))))))............ ( -40.20) >DroVir_CAF1 516 120 + 1 AUCGCCGCUAGCUGUGAGGACGAUCAGGCGCGCAAUCGCGUCAUUGGGGCAGCCAGCCAGUGUGCACUGGCCACCAGUCAGCUGGUGGCCUGCGCCAAGGUGGUGGCUCCCACCCUGCAC ...(((((.....)))(((.((((..((((((....))))))))))(((.(((((.(((.((((((..(((((((((....))))))))))))))...).)))))))))))..))))).. ( -58.30) >DroGri_CAF1 516 120 + 1 AUUGCCGCCAGCUGUGAGGAUGAUGAGGCACGCAAUCGUGUCAUUGGAGCAGCUAGCCAAUGUGCCCUGGCCACCAGUCAGUUGGUGGCAUGCGCCAAGGUAGUUGCACCCACUCUACAC .............(((((..((....((((((....))))))...((.(((((((.((...((((....((((((((....))))))))..))))...))))))))).))))..)).))) ( -44.90) >DroWil_CAF1 516 120 + 1 AUUGCUGCCAGCUGUGAGGAUGAGCAAGCCAGAAAUCGGGUAAUUGGAGCAGCCAGCCAAUGUGCCUUGGCCACGAGCCAGCUGGUGGCCUGUGCUAAGGUAGUGGCUCCCACUUUGCAC .(((((.((........))...))))).(((....).))((((.(((.(.((((((((...(..(...((((((.((....)).)))))).)..)...)))..)))))))))..)))).. ( -42.10) >DroMoj_CAF1 516 120 + 1 AUUGCCGCCAGCUGUGAGGAUGAUCAGGCGCGCAAUCGCGUCAUCGCUGCGGCUAGCCAAUGUGCACUGGCCACCAGUCAGCUGGUGGCCUGCGCCAAGGUGGUGGCACCCACUCUGCAU ..(((((((((((((..((.((((..((((((....)))))))))))))))))).(((...(((((..(((((((((....))))))))))))))...))))))))))............ ( -59.80) >DroAna_CAF1 516 120 + 1 AUCGCCGCCAGCUGCGAGGACGAGCAGGCGAGGAACCGGGUGAUCGGAGCGGCCAGCCAGUGCGCUCUGGCUACCAGCCAACUGGUGGCCUGUGCCAAGGUGGUGGCUCCCACGCUGCAU (((((((((.(((.((....))))).)))..(....).)))))).(((((.((((.((...((((...(((((((((....))))))))).))))...)))))).))))).......... ( -58.10) >consensus AUUGCCGCCAGCUGUGAGGAUGAUCAGGCGAGCAAUCGCGUCAUUGGAGCAGCCAGCCAAUGUGCCCUGGCCACCAGUCAGCUGGUGGCCUGCGCCAAGGUGGUGGCACCCACUCUGCAC ...((((((.(((((...(((((...((.......))...)))))...)))))..(((...((((...(((((((((....))))))))).))))...)))))))))............. (-38.49 = -38.25 + -0.24)



| Location | 8,538,656 – 8,538,776 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.94 |
| Mean single sequence MFE | -53.70 |
| Consensus MFE | -44.97 |
| Energy contribution | -45.28 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.40 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.909540 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8538656 120 + 23771897 UUAUUGGAGCUGCCAGUCAGUGUGCCCUGGCUACCAGUCAAUUGGUGGCUUGUGCCAAAGUUGUGGCACCAACGCUCCACAAUGCUGCCUGUCGUGAGCAACUAGAAGCGGCUGCCAAAA ...(((((((((((((.((.((((....(((((((((....))))))))).((((((......))))))........)))).)))))..((.(....))).......)))))).)))).. ( -43.50) >DroVir_CAF1 556 120 + 1 UCAUUGGGGCAGCCAGCCAGUGUGCACUGGCCACCAGUCAGCUGGUGGCCUGCGCCAAGGUGGUGGCUCCCACCCUGCACAAUGCUGCCUGUCGGGAGCAGCUGGAGGCGGCGGCCAGAA .......(((.(((..((((((((((..(((((((((....)))))))))........(((((......))))).)))))..((((.((....)).)))))))))....))).))).... ( -61.10) >DroGri_CAF1 556 120 + 1 UCAUUGGAGCAGCUAGCCAAUGUGCCCUGGCCACCAGUCAGUUGGUGGCAUGCGCCAAGGUAGUUGCACCCACUCUACACAAUGCUGCCUGCCGGGAACAAUUGGAGGCGGCGGCCAGAA ...((((.(((((((.((...((((....((((((((....))))))))..))))...)))))))))...............((((((((.((((......)))))))))))).)))).. ( -51.10) >DroWil_CAF1 556 120 + 1 UAAUUGGAGCAGCCAGCCAAUGUGCCUUGGCCACGAGCCAGCUGGUGGCCUGUGCUAAGGUAGUGGCUCCCACUUUGCACAAUGCUGCCUGUCGAGAACAAUUGGAGGCAGCGGCCCGAA ...((((.((((((((((...(..(...((((((.((....)).)))))).)..)...)))..)))))..............((((((((.((((......)))))))))))))))))). ( -51.00) >DroMoj_CAF1 556 120 + 1 UCAUCGCUGCGGCUAGCCAAUGUGCACUGGCCACCAGUCAGCUGGUGGCCUGCGCCAAGGUGGUGGCACCCACUCUGCAUAAUGCGGCCUGUCGCGAGCAAUUGGAGGCGGCGGCCCGGA .....(((((.(((..(((((.(((...(((((((((....)))))))))((((...(((((((((...))))).(((.....)))))))..)))).)))))))).))).)))))..... ( -57.60) >DroAna_CAF1 556 120 + 1 UGAUCGGAGCGGCCAGCCAGUGCGCUCUGGCUACCAGCCAACUGGUGGCCUGUGCCAAGGUGGUGGCUCCCACGCUGCAUAACGCUGCCUGCAGGGAACAACUCGAGGCGGCAGCCCGUA .....(((((.((((.((...((((...(((((((((....))))))))).))))...)))))).))))).............((((((.((.(((.....)))...))))))))..... ( -57.90) >consensus UCAUUGGAGCAGCCAGCCAAUGUGCCCUGGCCACCAGUCAGCUGGUGGCCUGCGCCAAGGUGGUGGCACCCACUCUGCACAAUGCUGCCUGUCGGGAACAACUGGAGGCGGCGGCCAGAA .....(((((.((((.((...((((...(((((((((....))))))))).))))...)))))).))))).............(((((((.((((......)))))))))))........ (-44.97 = -45.28 + 0.31)


| Location | 8,538,656 – 8,538,776 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.94 |
| Mean single sequence MFE | -52.75 |
| Consensus MFE | -35.69 |
| Energy contribution | -36.78 |
| Covariance contribution | 1.09 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.62 |
| SVM RNA-class probability | 0.802284 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8538656 120 - 23771897 UUUUGGCAGCCGCUUCUAGUUGCUCACGACAGGCAGCAUUGUGGAGCGUUGGUGCCACAACUUUGGCACAAGCCACCAAUUGACUGGUAGCCAGGGCACACUGACUGGCAGCUCCAAUAA ..((((.(((.(((((..((((((.......))))))...(((..((.(((((((((......))))))..((.((((......)))).))))).)).))).))..))).)))))))... ( -42.30) >DroVir_CAF1 556 120 - 1 UUCUGGCCGCCGCCUCCAGCUGCUCCCGACAGGCAGCAUUGUGCAGGGUGGGAGCCACCACCUUGGCGCAGGCCACCAGCUGACUGGUGGCCAGUGCACACUGGCUGGCUGCCCCAAUGA ....(((.((((((....((((((.......))))))...(((((((((((......))))))))((((.(((((((((....))))))))).)))).))).))).))).)))....... ( -66.20) >DroGri_CAF1 556 120 - 1 UUCUGGCCGCCGCCUCCAAUUGUUCCCGGCAGGCAGCAUUGUGUAGAGUGGGUGCAACUACCUUGGCGCAUGCCACCAACUGACUGGUGGCCAGGGCACAUUGGCUAGCUGCUCCAAUGA .((((((((((((((((..........)).)))).((((.((((.((((((......))).))).))))))))............))))))))))...((((((.........)))))). ( -45.90) >DroWil_CAF1 556 120 - 1 UUCGGGCCGCUGCCUCCAAUUGUUCUCGACAGGCAGCAUUGUGCAAAGUGGGAGCCACUACCUUAGCACAGGCCACCAGCUGGCUCGUGGCCAAGGCACAUUGGCUGGCUGCUCCAAUUA ....(((((((((((....(((....))).)))))))..(((((((.((((......)))).)).)))))))))..((((..(((.(((((....)).))).)))..))))......... ( -52.00) >DroMoj_CAF1 556 120 - 1 UCCGGGCCGCCGCCUCCAAUUGCUCGCGACAGGCCGCAUUAUGCAGAGUGGGUGCCACCACCUUGGCGCAGGCCACCAGCUGACUGGUGGCCAGUGCACAUUGGCUAGCCGCAGCGAUGA ...((((....))))...((((((.(((.(.((((((.....))...(((.((((((......)))))).(((((((((....)))))))))....)))...)))).).))))))))).. ( -55.40) >DroAna_CAF1 556 120 - 1 UACGGGCUGCCGCCUCGAGUUGUUCCCUGCAGGCAGCGUUAUGCAGCGUGGGAGCCACCACCUUGGCACAGGCCACCAGUUGGCUGGUAGCCAGAGCGCACUGGCUGGCCGCUCCGAUCA ..((((((((((....(.((.(((((((((..(((......))).))).)))))).)))....)))))..(((((((((....)))))((((((......)))))))))))).))).... ( -54.70) >consensus UUCGGGCCGCCGCCUCCAAUUGCUCCCGACAGGCAGCAUUGUGCAGAGUGGGAGCCACCACCUUGGCACAGGCCACCAGCUGACUGGUGGCCAGGGCACACUGGCUGGCUGCUCCAAUGA ....(((.((((...((((((((((..............(((((...((((......))))....)))))(((((((((....))))))))).))))).))))).)))).)))....... (-35.69 = -36.78 + 1.09)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:34 2006