
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
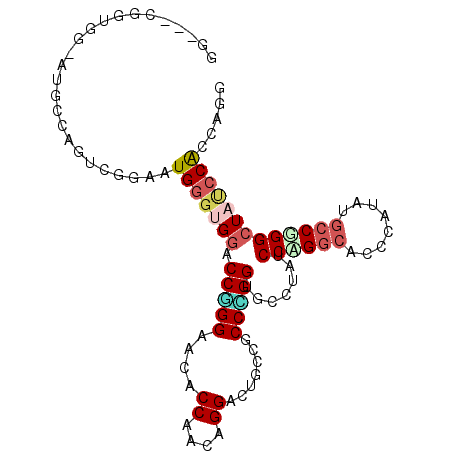
| Location | 8,441,780 – 8,441,878 |
| Length | 98 |
| Max. P | 0.875675 |

| Location | 8,441,780 – 8,441,878 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 69.57 |
| Mean single sequence MFE | -46.02 |
| Consensus MFE | -20.82 |
| Energy contribution | -21.43 |
| Covariance contribution | 0.61 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.45 |
| SVM decision value | 0.89 |
| SVM RNA-class probability | 0.875675 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8441780 98 + 23771897 GG---CGGUGGGAUGCCCCCAGGAAUGGGUGGACCGGGAACACCGACGGGACUGCCGCCAGGUGCCUAUCCUGGCAGCCAUAUGCCGGGAUAUCCACAAGG ((---(((..(....((((((....)))).))..(((.....)))......)..)))))..(((..((((((((((......))))))))))..))).... ( -48.40) >DroVir_CAF1 51458 92 + 1 GG---UGGC---AUGAGUGUCGGCAUGGGCGGGCCAGGCACGCCAACAGGUCUGCCACCUGGUGCCUAUCCGGGCUCUCACCUGGCCGGCUAUCCGCC--- ((---(((.---(((...((((((((((((..((((((((.(((....))).))))...)))))))))).((((......))))))))))))))))))--- ( -42.30) >DroGri_CAF1 40785 92 + 1 GG---UGUU---GUGGGUGUCGGCAUGGGUGGACCGGGUACGCCAACAGGAUUGCCGCCUGGUGCGUAUCCCGGCUCGCACAUGCCUGGCUACCCACC--- ..---....---(((((((((((((((.(((..((((((((((...((((.......))))..))))).)))))..))).)))))).))).)))))).--- ( -53.50) >DroEre_CAF1 23512 98 + 1 GG---CGGUGGAAUGCCCCCUGGAAUGGGUGGACCGGGAACACCGACGGGAUUGCCGCCCGGCGCUUAUCCAGGCAGCCAUAUGCCGGGCUAUCCACAAGG ..---..(((((..((((..((((.((((((...(((.....))).((((.......)))).))))))))))((((......))))))))..))))).... ( -42.40) >DroWil_CAF1 153201 95 + 1 AC---A---GGGGUACCAGUCGGCAUGACUGGACCGGGAACUCCAACUGGCCUGCCGCCCGGAGCCUAUCCAGGAUCUCAUAUGCCUGGCUAUCCGCCAGG ..---.---..(((.((((((.....)))))))))((((..(((....(((.....))).(((.....))).))))))).....((((((.....)))))) ( -36.80) >DroAna_CAF1 106926 101 + 1 GGACCCGGUCCUGGCCCAGUUGGAAUGGGCGGCCCCGGCACACCGACCGGCUUGCCGCCGGGUGCUUAUCCUGGCAGCCAUAUGCCGGGCUAUCCGCCGGG ...((((((..((((((....(((.((((((.((((((((..((....))..)))))..)))))))))))).((((......))))))))))...)))))) ( -52.70) >consensus GG___CGGUGG_AUGCCAGUCGGAAUGGGUGGACCGGGAACACCAACAGGACUGCCGCCCGGUGCCUAUCCAGGCACCCAUAUGCCGGGCUAUCCACCAGG .........................(((((((.(((((....((....)).......))))).......((((((........)))))))))))))..... (-20.82 = -21.43 + 0.61)


| Location | 8,441,780 – 8,441,878 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 69.57 |
| Mean single sequence MFE | -44.37 |
| Consensus MFE | -19.17 |
| Energy contribution | -19.95 |
| Covariance contribution | 0.78 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.43 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.742203 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8441780 98 - 23771897 CCUUGUGGAUAUCCCGGCAUAUGGCUGCCAGGAUAGGCACCUGGCGGCAGUCCCGUCGGUGUUCCCGGUCCACCCAUUCCUGGGGGCAUCCCACCG---CC ....((((.....(((((.....(((((((((.......)))))))))......)))))(((((((((...........)))))))))..))))..---.. ( -41.50) >DroVir_CAF1 51458 92 - 1 ---GGCGGAUAGCCGGCCAGGUGAGAGCCCGGAUAGGCACCAGGUGGCAGACCUGUUGGCGUGCCUGGCCCGCCCAUGCCGACACUCAU---GCCA---CC ---...((...(((..((.(((....))).))...))).)).(((((((....((((((((((...((....))))))))))))....)---))))---)) ( -45.10) >DroGri_CAF1 40785 92 - 1 ---GGUGGGUAGCCAGGCAUGUGCGAGCCGGGAUACGCACCAGGCGGCAAUCCUGUUGGCGUACCCGGUCCACCCAUGCCGACACCCAC---AACA---CC ---.((((((.(...((((((.(.(..(((((.(((((..((((.......))))...))))))))))..).).))))))..)))))))---....---.. ( -46.70) >DroEre_CAF1 23512 98 - 1 CCUUGUGGAUAGCCCGGCAUAUGGCUGCCUGGAUAAGCGCCGGGCGGCAAUCCCGUCGGUGUUCCCGGUCCACCCAUUCCAGGGGGCAUUCCACCG---CC ....(((((..(((((((.....))).((((((..((((((((.(((.....)))))))))))...((....))...))))))))))..)))))..---.. ( -42.50) >DroWil_CAF1 153201 95 - 1 CCUGGCGGAUAGCCAGGCAUAUGAGAUCCUGGAUAGGCUCCGGGCGGCAGGCCAGUUGGAGUUCCCGGUCCAGUCAUGCCGACUGGUACCCC---U---GU ((((((.....))))))..........((((((.....))))))..(((((......((.....))(((((((((.....)))))).)))))---)---)) ( -39.00) >DroAna_CAF1 106926 101 - 1 CCCGGCGGAUAGCCCGGCAUAUGGCUGCCAGGAUAAGCACCCGGCGGCAAGCCGGUCGGUGUGCCGGGGCCGCCCAUUCCAACUGGGCCAGGACCGGGUCC ((((((((....((((((((...((((((.((.......)).))))))..(((....))))))))))).))(((((.......)))))...).)))))... ( -51.40) >consensus CCUGGCGGAUAGCCCGGCAUAUGGCAGCCAGGAUAGGCACCAGGCGGCAAUCCCGUCGGUGUUCCCGGUCCACCCAUGCCGACAGGCAC_CCACCG___CC ...(((((((..((.(((........))).))....(((((.(((((.....)))))))))).....)))))))........................... (-19.17 = -19.95 + 0.78)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:46:08 2006