
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 8,394,803 – 8,394,963 |
| Length | 160 |
| Max. P | 0.969281 |

| Location | 8,394,803 – 8,394,923 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.50 |
| Mean single sequence MFE | -51.84 |
| Consensus MFE | -42.62 |
| Energy contribution | -44.62 |
| Covariance contribution | 2.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.64 |
| SVM RNA-class probability | 0.969281 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8394803 120 + 23771897 AAAUGCCGCCAACGUGAACAUGGCCAAGAGUGCGCCAAACAGCUGUCGCUGGCUCUUGGUGCCGCCAAAGCUGAGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUGGCGAUCUGGCU ....(((((((..(....).))))..(((...(((((..((((....))))......(((((.((((.(((((.(((((......))))).))))).)))).)))))))))).)))))). ( -53.30) >DroSec_CAF1 2010 120 + 1 AAAUGCCGCCAACGUGAACAUGGCCAAGAGUGCGCCAAACAGCUGUCGCUGGCUCUUGGUGCCGCCAAAGCUGAGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUGGCGAUCUGGCU ....(((((((..(....).))))..(((...(((((..((((....))))......(((((.((((.(((((.(((((......))))).))))).)))).)))))))))).)))))). ( -53.30) >DroSim_CAF1 1983 120 + 1 AAAUGCCGCCAACGUGAACAUGGCCAAGAGUGCGCCAAACAGCUGUCGCUGGCUCUUGGUGCCGCCAAAGCUGAGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUGGCGAUCUGGCU ....(((((((..(....).))))..(((...(((((..((((....))))......(((((.((((.(((((.(((((......))))).))))).)))).)))))))))).)))))). ( -53.30) >DroEre_CAF1 16 120 + 1 AAAUGCCGCCAACGUGAACAUGGCCAAGAGUGCGCCAAACAGCUGUCGCUGGCUCUUGGAGCCGCCAAAGCUGAGGCCCAGACUGGGCCUGUGGUUAUGGCUGCAUCUCUCGAUCUGGCU ....(((((((..(....).))))(.(((((((((((..(((((...((.(((((...)))))))...)))))((((((.....)))))).......)))).))).)))).)....))). ( -53.30) >DroYak_CAF1 16 120 + 1 AAAUGCCGCCAACAUGAACAUGGCCAAGAGUGCGCCAAACAGCUGUCGCUGGCUCUUGGAUCCUCCAAAGCUGUGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUCUGGAUCUGGCU .......((((.(((....))).(((.((((((((((.(((((((((((.((((.((((.....)))))))))))))((.....)))))))).....)))).))).))))))...)))). ( -46.00) >consensus AAAUGCCGCCAACGUGAACAUGGCCAAGAGUGCGCCAAACAGCUGUCGCUGGCUCUUGGUGCCGCCAAAGCUGAGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUGGCGAUCUGGCU ....(((((((.........))))..(((...(((((..((((....))))......(((((.((((.((((..(((((......)))))..)))).)))).)))))))))).)))))). (-42.62 = -44.62 + 2.00)



| Location | 8,394,803 – 8,394,923 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.50 |
| Mean single sequence MFE | -46.15 |
| Consensus MFE | -39.38 |
| Energy contribution | -39.78 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.62 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.840714 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8394803 120 - 23771897 AGCCAGAUCGCCAGAUGCAGCCAUAACCACAGCCCCAGUCUGGGCCUCAGCUUUGGCGGCACCAAGAGCCAGCGACAGCUGUUUGGCGCACUCUUGGCCAUGUUCACGUUGGCGGCAUUU .((((((.((((((((((.((((........((((......))))........)))).)))........((((....)))))))))))...)))..((((((....)).))))))).... ( -43.29) >DroSec_CAF1 2010 120 - 1 AGCCAGAUCGCCAGAUGCAGCCAUAACCACAGCCCCAGUCUGGGCCUCAGCUUUGGCGGCACCAAGAGCCAGCGACAGCUGUUUGGCGCACUCUUGGCCAUGUUCACGUUGGCGGCAUUU .((((((.((((((((((.((((........((((......))))........)))).)))........((((....)))))))))))...)))..((((((....)).))))))).... ( -43.29) >DroSim_CAF1 1983 120 - 1 AGCCAGAUCGCCAGAUGCAGCCAUAACCACAGCCCCAGUCUGGGCCUCAGCUUUGGCGGCACCAAGAGCCAGCGACAGCUGUUUGGCGCACUCUUGGCCAUGUUCACGUUGGCGGCAUUU .((((((.((((((((((.((((........((((......))))........)))).)))........((((....)))))))))))...)))..((((((....)).))))))).... ( -43.29) >DroEre_CAF1 16 120 - 1 AGCCAGAUCGAGAGAUGCAGCCAUAACCACAGGCCCAGUCUGGGCCUCAGCUUUGGCGGCUCCAAGAGCCAGCGACAGCUGUUUGGCGCACUCUUGGCCAUGUUCACGUUGGCGGCAUUU .(((....((((((.(((.((((.......((((((.....))))))(((((.(.(((((((...))))).)).).)))))..)))))))))))))((((((....)).))))))).... ( -58.20) >DroYak_CAF1 16 120 - 1 AGCCAGAUCCAGAGAUGCAGCCAUAACCACAGCCCCAGUCUGGGCCACAGCUUUGGAGGAUCCAAGAGCCAGCGACAGCUGUUUGGCGCACUCUUGGCCAUGUUCAUGUUGGCGGCAUUU .(((....(.((((.(((.((((........((((......))))....(((((((.....)).)))))((((....))))..))))))))))).)((((.........))))))).... ( -42.70) >consensus AGCCAGAUCGCCAGAUGCAGCCAUAACCACAGCCCCAGUCUGGGCCUCAGCUUUGGCGGCACCAAGAGCCAGCGACAGCUGUUUGGCGCACUCUUGGCCAUGUUCACGUUGGCGGCAUUU .(((..(((....)))...((((.....(((..((.((..((.(((.(((((.(.(((((.......))).)).).)))))...))).))..)).))...)))......))))))).... (-39.38 = -39.78 + 0.40)

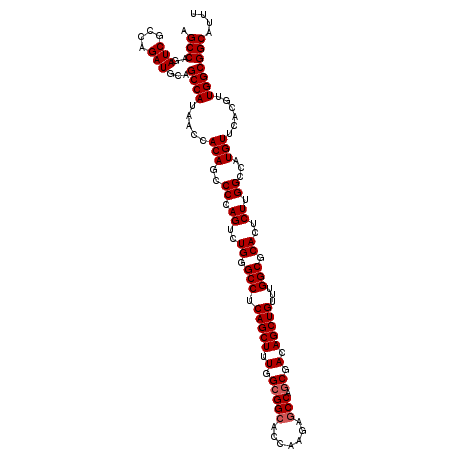

| Location | 8,394,843 – 8,394,963 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.67 |
| Mean single sequence MFE | -54.12 |
| Consensus MFE | -44.16 |
| Energy contribution | -45.20 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.810944 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8394843 120 + 23771897 AGCUGUCGCUGGCUCUUGGUGCCGCCAAAGCUGAGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUGGCGAUCUGGCUUGGAGUCUGGCCAAGUCCGUGGGCCUGUGUUUGGGUGGCG ((((......))))......((((((.((((..(((((((.....(((((.((((((.((((.((...(((......))))).))))))))))))))).)))))))..)))).)))))). ( -53.20) >DroSec_CAF1 2050 120 + 1 AGCUGUCGCUGGCUCUUGGUGCCGCCAAAGCUGAGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUGGCGAUCUGGCUUGGAGUCUGGCCAAGUCCGUGGGCCUGUGUUUGGGUGGCU ((((......))))......((((((.((((..(((((((.....(((((.((((((.((((.((...(((......))))).))))))))))))))).)))))))..)))).)))))). ( -52.70) >DroSim_CAF1 2023 120 + 1 AGCUGUCGCUGGCUCUUGGUGCCGCCAAAGCUGAGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUGGCGAUCUGGCUUGGAGUCUGGCCAAGUCCGUGGGCCUGUGUUUGGGUGGCU ((((......))))......((((((.((((..(((((((.....(((((.((((((.((((.((...(((......))))).))))))))))))))).)))))))..)))).)))))). ( -52.70) >DroEre_CAF1 56 120 + 1 AGCUGUCGCUGGCUCUUGGAGCCGCCAAAGCUGAGGCCCAGACUGGGCCUGUGGUUAUGGCUGCAUCUCUCGAUCUGGCUCGGAGUCUGGCCAAGACCGUGGGCCUGUGCUUGGGAGGCU (((....((.(((((...)))))((((.(((((((((((.....)))))).))))).)))).))..(((((((.(.((((((..((((.....))))..))))))...).)))))))))) ( -56.20) >DroYak_CAF1 56 120 + 1 AGCUGUCGCUGGCUCUUGGAUCCUCCAAAGCUGUGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUCUGGAUCUGGCUUGGAGUCUGGCCAAGACCGUGGGCCUCUGUUUGGGUGGCU ((((..(((.((((.((((.....)))))))))))((((((((.(((((..(((((.((((((...(((..(.......)..)))..)))))).)))))..).)))).)))))))))))) ( -55.80) >consensus AGCUGUCGCUGGCUCUUGGUGCCGCCAAAGCUGAGGCCCAGACUGGGGCUGUGGUUAUGGCUGCAUCUGGCGAUCUGGCUUGGAGUCUGGCCAAGUCCGUGGGCCUGUGUUUGGGUGGCU .((..((((.(((((..(((((.((((.((((..(((((......)))))..)))).)))).))))).........(((((((.......))))).))..)))))...))...))..)). (-44.16 = -45.20 + 1.04)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:45:38 2006