
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 8,189,650 – 8,189,780 |
| Length | 130 |
| Max. P | 0.886323 |

| Location | 8,189,650 – 8,189,745 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 83.46 |
| Mean single sequence MFE | -32.37 |
| Consensus MFE | -18.52 |
| Energy contribution | -18.38 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.81 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.586201 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8189650 95 + 23771897 CUCCG----CCGCUUACUAUCCGGCGUAUGCCAGCUAUCCGGCCUACGCGGCGUAUGCGUAUGCACCGGUUUAUGGCGCAUACGCCACGUAUCCAUACU ....(----(((.........))))(((((...(((....))).((((.(((((((((((..((....)).....))))))))))).))))..))))). ( -36.50) >DroPse_CAF1 18849 99 + 1 CUGCACCAUCGGCUUACUUUCCGACUUAUGCCAGCUAUCCGGCAUACGCGUCGUAUGCGUAUGCACCGGUUUAUGGCGCAUACGCCACGUAUCCAUACU ..........((..(((....((((.((((((........))))))...))))...((((((((.(((.....))).))))))))...))).))..... ( -32.30) >DroEre_CAF1 20518 95 + 1 CUCCG----CCGCCUACUAUCCGGCCUAUGCCAGCUAUCCGGCCUACGCGGCGUAUGCGUAUGCACCGGUUUAUGGCGCAUACGCCACGUAUCCCUACU ...((----((((.........(((....))).(((....)))....))))))...((((((((.(((.....))).)))))))).............. ( -34.50) >DroWil_CAF1 18325 99 + 1 CAGCUCCGUCAACCUAUUAUCCCGGAUAUUUCAGUUCGCCGGCCUAUGCGGCGUAUACGUAUACACCUGUUUAUGGUGCCUAUGCCACAUAUCCGUAUU ......................(((((((....((.(((((.......)))))...))(((((((((.......))))..)))))...))))))).... ( -24.30) >DroAna_CAF1 16597 99 + 1 CUACGCCCUCUGCCUAUUAUCCGGCGUAUGCCAGCUAUCCGGCCUACGCGUCGUAUGCGUAUUCUCCGGUUUAUGGCGCAUACGCCACGUAUCCCUACU .(((((.....)).........(((((((((..(((((((((..(((((((...)))))))....))))...))))))))))))))..)))........ ( -34.30) >DroPer_CAF1 19197 99 + 1 CUGCACCAUCGGCUUACUUUCCGACUUAUGCCAGCUAUCCGGCAUACGCGUCGUAUGCGUAUGCACCGGUUUAUGGCGCAUACGCCACGUAUCCAUACU ..........((..(((....((((.((((((........))))))...))))...((((((((.(((.....))).))))))))...))).))..... ( -32.30) >consensus CUGCGCC_UCCGCCUACUAUCCGGCGUAUGCCAGCUAUCCGGCCUACGCGGCGUAUGCGUAUGCACCGGUUUAUGGCGCAUACGCCACGUAUCCAUACU .............................(((........))).((((.(.(((((((((..((....)).....))))))))).).))))........ (-18.52 = -18.38 + -0.14)

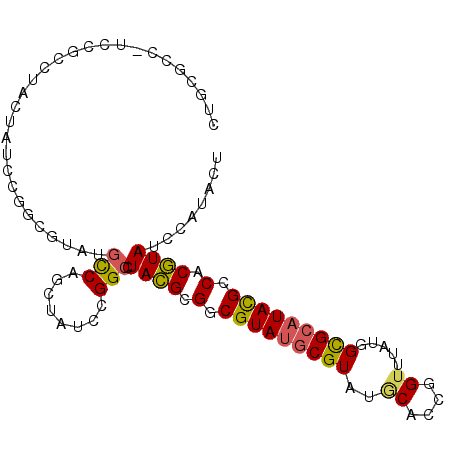
| Location | 8,189,650 – 8,189,745 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 83.46 |
| Mean single sequence MFE | -39.73 |
| Consensus MFE | -27.90 |
| Energy contribution | -28.27 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.21 |
| Mean z-score | -3.37 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.94 |
| SVM RNA-class probability | 0.886323 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8189650 95 - 23771897 AGUAUGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCAUACGCCGGAUAGUAAGCGG----CGGAG ..((((....))))(((((((((((.......)))))))))))...(((((((((.(((((....))))).)))((......))..))))----))... ( -44.40) >DroPse_CAF1 18849 99 - 1 AGUAUGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGACGCGUAUGCCGGAUAGCUGGCAUAAGUCGGAAAGUAAGCCGAUGGUGCAG ..........(((((((((((((((.......))))))))))).))))..(((((((((((....)))))))).(((((........)))))..))).. ( -43.30) >DroEre_CAF1 20518 95 - 1 AGUAGGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCAUAGGCCGGAUAGUAGGCGG----CGGAG ..........(((((((((((((((.......))))))))))).))))((((....(((((....)))))....(((........))).)----))).. ( -43.60) >DroWil_CAF1 18325 99 - 1 AAUACGGAUAUGUGGCAUAGGCACCAUAAACAGGUGUAUACGUAUACGCCGCAUAGGCCGGCGAACUGAAAUAUCCGGGAUAAUAGGUUGACGGAGCUG ....(((.((((((((....(((((.......)))))....(....)))))))))..))).............((((.(((.....)))..)))).... ( -28.20) >DroAna_CAF1 16597 99 - 1 AGUAGGGAUACGUGGCGUAUGCGCCAUAAACCGGAGAAUACGCAUACGACGCGUAGGCCGGAUAGCUGGCAUACGCCGGAUAAUAGGCAGAGGGCGUAG ..........((((((((((...((.......))...)))))).))))(((((((.(((((....))))).)))(((........))).....)))).. ( -35.60) >DroPer_CAF1 19197 99 - 1 AGUAUGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGACGCGUAUGCCGGAUAGCUGGCAUAAGUCGGAAAGUAAGCCGAUGGUGCAG ..........(((((((((((((((.......))))))))))).))))..(((((((((((....)))))))).(((((........)))))..))).. ( -43.30) >consensus AGUAUGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGACGCGUAGGCCGGAUAGCUGGCAUAAGCCGGAUAGUAAGCCGA_GGCGCAG .....((.(((((((((((((((((.......)))))))))))......))))))..))......(((((....))))).................... (-27.90 = -28.27 + 0.37)



| Location | 8,189,674 – 8,189,780 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 86.64 |
| Mean single sequence MFE | -40.74 |
| Consensus MFE | -30.68 |
| Energy contribution | -30.77 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.561614 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 8189674 106 - 23771897 CCGCAGCAGCAGGAUUCACCGCCCUAGAUACGAGUAGUAUGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCA ..(((((.((.((.....))))((...((((.....))))((.(((((((((((((((.(((......)))....)))))))))))))))..))))...))).)). ( -42.30) >DroPse_CAF1 18877 105 - 1 GGAUAUC-CUUGGAAUCAUCGUCGCAGAUAUGAGUAGUAUGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGACGCGUAUGCCGGAUAGCUGGCA ...((((-(......((((.(((...))))))).......)))))(((((((((((((((.......))))))))))).)))).......((((((....)))))) ( -39.12) >DroEre_CAF1 20542 106 - 1 CAACAGCAGCAGGACUCACCGCCCUAGAUACGAGUAGUAGGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCA .....((.((......(((..(((((..((....)).)))))....)))(((((((((((.......)))))))))))....)).))....(((((....))))). ( -42.40) >DroYak_CAF1 17897 106 - 1 CCACAGCGGCAGGAUUUAUCGUCCUAGAUAGGAGAAGUAGGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGCCGCGUAGGCCGGAUAGCUGGCA .....((((((((((.....)))))...........(((....)))((((((((((((((.......))))))))))).))))))))....(((((....))))). ( -46.30) >DroAna_CAF1 16625 102 - 1 CUAUAUC----CGCUUCAGCGCCUCAGGUAGGAGUAGUAGGGAUACGUGGCGUAUGCGCCAUAAACCGGAGAAUACGCAUACGACGCGUAGGCCGGAUAGCUGGCA ((((.((----(((....))(((...))).)))...))))...((((((.((((((((((.......))......)))))))).)))))).(((((....))))). ( -35.20) >DroPer_CAF1 19225 105 - 1 GGAUAUC-CUUGGAAUCAUCGUCGCAGAUAUGAGUAGUAUGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGACGCGUAUGCCGGAUAGCUGGCA ...((((-(......((((.(((...))))))).......)))))(((((((((((((((.......))))))))))).)))).......((((((....)))))) ( -39.12) >consensus CCACAGC_GCAGGAUUCAUCGCCCCAGAUACGAGUAGUAGGGAUACGUGGCGUAUGCGCCAUAAACCGGUGCAUACGCAUACGACGCGUAGGCCGGAUAGCUGGCA ...................(((..............(((....)))((((((((((((((.......))))))))))).)))...)))...(((((....))))). (-30.68 = -30.77 + 0.08)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:43:38 2006