
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 7,543,797 – 7,543,935 |
| Length | 138 |
| Max. P | 0.925194 |

| Location | 7,543,797 – 7,543,900 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.46 |
| Mean single sequence MFE | -30.60 |
| Consensus MFE | -24.25 |
| Energy contribution | -24.87 |
| Covariance contribution | 0.61 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.925194 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
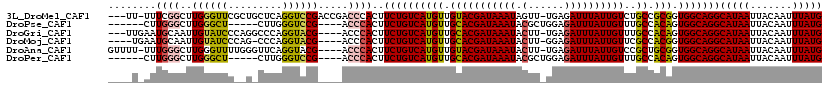
>3L_DroMel_CAF1 7543797 103 - 23771897 ACCCACUUCUGUCAUGUUGUACGAUAAAUAGUU-UGAGAUUUAUUGUCUGCCGCGGUGGCAGGCAUAAUUACAAUUUAUGCCCAAGGUGGU---------CCCCCCAAGAGCA------- ..((((((.((((((((.(((((((((((....-....)))))))))..)).)).))))))(((((((.......)))))))..)))))).---------.............------- ( -27.70) >DroPse_CAF1 7052 104 - 1 ACCCACUUCUGUCAUGUUGCACGAUAAAUACGCUGGAGAUUUAUUGUUUGCCACAGUGGCAGGCAUAAUUACAAUUUAUGCCCAAGGUGGU---------CUCCACACACACA------- ..((((((.((((((((.(((((((((((.(......))))))))))..)).))).)))))(((((((.......)))))))..)))))).---------.............------- ( -29.60) >DroGri_CAF1 7411 102 - 1 ACCCACUUCUGUCAUGUUGCACGAUAAAUACUU-UGAGAUUUAUUGUUUGCCACAGUGGCAGGCAUAAUUACAAUUUAUGCCCCUUU--GG---------GGCAGCA---GCAGCCA--- ..............((((((.......(((..(-((.((((...((((((((.....)))))))).)))).)))..)))(((((...--))---------))).)))---)))....--- ( -32.60) >DroYak_CAF1 13221 102 - 1 ACCCACUUCUGUCAUGUUGUACGAUAAAUACUU-UGAGAUUUAUUGUUCGCCGCGGUGGCAGGCAUAAUUACAAUUUAUGCCCAAGGUGGU---------CCC-CCAAGAGCA------- ..((((((.((((((((.(((((((((((....-....)))))))))..)).)).))))))(((((((.......)))))))..)))))).---------...-.........------- ( -27.70) >DroMoj_CAF1 8368 110 - 1 ACCCACUUCUGUCAUGUUGCACGAUAAAUACUU-GGAGAUUUAUUGUUCGCCACGGUGGCAGGCAUAAUUACAAUUUAUGCCCCAUUCGGG---------GGCAGCACAAACAGCCAACA ........((((..(((((((((((((((....-....)))))))))...((.((((((..(((((((.......)))))))))).))).)---------)))))))...))))...... ( -31.90) >DroAna_CAF1 5022 106 - 1 ACCCACUUCUGUCAUGUUGUACGAUAAAUACUU-UGAGAUUUAUUGUCCGCUGCGGUGGCAGGCAUAAUUACAAUUUAUGCCCAAGGUGGUGGUGGUGGUCCC-CUAC------------ ..(((((.(..((((.(((.(((((((((....-....)))))))))((((....))))..(((((((.......)))))))))).))))..).)))))....-....------------ ( -34.10) >consensus ACCCACUUCUGUCAUGUUGCACGAUAAAUACUU_UGAGAUUUAUUGUUCGCCACGGUGGCAGGCAUAAUUACAAUUUAUGCCCAAGGUGGU_________CCC_CCAC_AGCA_______ ..((((((.((((((((.(((((((((((.........)))))))))..)).))).)))))(((((((.......)))))))..)))))).............................. (-24.25 = -24.87 + 0.61)

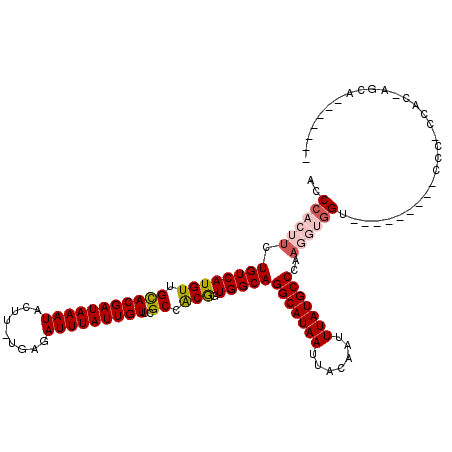
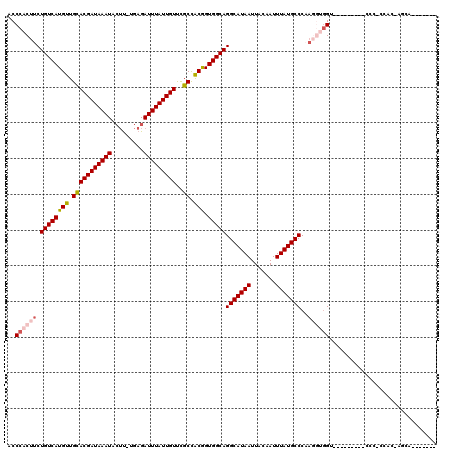
| Location | 7,543,821 – 7,543,935 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 81.09 |
| Mean single sequence MFE | -29.83 |
| Consensus MFE | -20.92 |
| Energy contribution | -20.40 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.23 |
| Structure conservation index | 0.70 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.519989 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
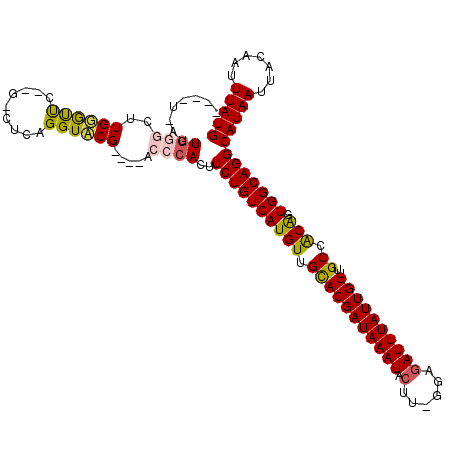
>3L_DroMel_CAF1 7543821 114 - 23771897 ---UU-UUUCGGCUUGGGUUCGCUGCUCAGGUCCGACCGACCCACUUCUGUCAUGUUGUACGAUAAAUAGUU-UGAGAUUUAUUGUCUGCCGCGGUGGCAGGCAUAAUUACAAUUUAUG ---..-..(((((((((((.....))))))).))))..(((........)))..((((((.(((((((....-....)))))))(((((((.....))))))).....))))))..... ( -33.30) >DroPse_CAF1 7076 104 - 1 ------CUUGGGCUUGGGCU-----CUUGGGUCCG----ACCCACUUCUGUCAUGUUGCACGAUAAAUACGCUGGAGAUUUAUUGUUUGCCACAGUGGCAGGCAUAAUUACAAUUUAUG ------..((((.(((((((-----....))))))----)))))..((((((((((.(((((((((((.(......))))))))))..)).))).)))))))(((((.......))))) ( -34.50) >DroGri_CAF1 7434 111 - 1 ---UUGAAUGCAAUUGUAUCCCAGGCCCAGGUACG----ACCCACUUCUGUCAUGUUGCACGAUAAAUACUU-UGAGAUUUAUUGUUUGCCACAGUGGCAGGCAUAAUUACAAUUUAUG ---....((((..(((((((.........))))))----).......(((((((((.(((((((((((....-....)))))))))..)).))).)))))))))).............. ( -25.70) >DroMoj_CAF1 8399 109 - 1 ----UGAAUGCAAUUGUAUCCCAG-CCCAGGUACG----ACCCACUUCUGUCAUGUUGCACGAUAAAUACUU-GGAGAUUUAUUGUUCGCCACGGUGGCAGGCAUAAUUACAAUUUAUG ----...((((..(((((((....-....))))))----).......(((((((((.(((((((((((....-....)))))))))..)).)).))))))))))).............. ( -26.10) >DroAna_CAF1 5049 113 - 1 GUUUU-UUUGGGCUUGGGUUUUGGGUUCAGGUACG----ACCCACUUCUGUCAUGUUGUACGAUAAAUACUU-UGAGAUUUAUUGUCCGCUGCGGUGGCAGGCAUAAUUACAAUUUAUG (((..-((((((((..(...)..))))))))...)----)).....((((((((((.(((((((((((....-....)))))))))..)).)).))))))))(((((.......))))) ( -24.90) >DroPer_CAF1 7117 104 - 1 ------CUUGGGCUUGGGCU-----CUUGGGUCCG----ACCCACUUCUGUCAUGUUGCACGAUAAAUACGCUGGAGAUUUAUUGUUUGCCACAGUGGCAGGCAUAAUUACAAUUUAUG ------..((((.(((((((-----....))))))----)))))..((((((((((.(((((((((((.(......))))))))))..)).))).)))))))(((((.......))))) ( -34.50) >consensus ____U_AUUGGGCUUGGGUUC__G_CUCAGGUACG____ACCCACUUCUGUCAUGUUGCACGAUAAAUACUU_GGAGAUUUAUUGUUUGCCACAGUGGCAGGCAUAAUUACAAUUUAUG ........((((..((((((.........)))))).....))))..((((((((((.(((((((((((.(......))))))))))..)).))).)))))))(((((.......))))) (-20.92 = -20.40 + -0.52)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:39:50 2006