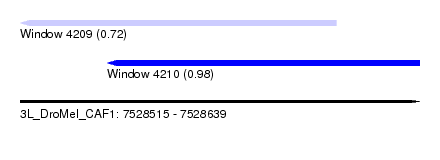
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 7,528,515 – 7,528,639 |
| Length | 124 |
| Max. P | 0.979414 |

| Location | 7,528,515 – 7,528,613 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 109 |
| Reading direction | reverse |
| Mean pairwise identity | 80.12 |
| Mean single sequence MFE | -22.29 |
| Consensus MFE | -14.23 |
| Energy contribution | -13.82 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.39 |
| SVM RNA-class probability | 0.716508 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 7528515 98 - 23771897 CACCGCUAAUGAUCUAUGAAGUGCAAGUGU--CUGUACUUUUACUUUCAUUUAUCAAAAGUGGAGGCGUCAUC-UCUUUUGGCAAAAAAAUAAA--------CGUGGAA ..((((.(((((.....((((((((.....--.)))))))).....)))))..(((((((.(((.......))-))))))))............--------.)))).. ( -22.80) >DroVir_CAF1 217146 92 - 1 UAACGCUAAUGAUCUAUGAAGUGUAAGUGUUUUUGUACUUUUACUUUCAUUUAUCAUAAAUGGAGGCGUCAUU-UGU---GCUCAAGC--C-----------AUUUGAA ........(((((..((((((((.(((((......))))).))).)))))..)))))((((((.((((.(...-.))---)))....)--)-----------))))... ( -20.70) >DroGri_CAF1 170216 92 - 1 CAACGCUAAUGAUCUGUGAAGUGUAAGUAUU-UUGUACUUUUACUUUCAUUUAUCAUAAAUGGAAGCGUCAUUUUGU---GCUCAAGA--C-----------AGUUGAA ((((.((.(((((..((((((((.((((((.-..)))))).))).)))))..)))))...(((..(((......)))---..))))).--.-----------.)))).. ( -19.90) >DroYak_CAF1 175248 104 - 1 CACCGCUAAUGAUCUAUGAAGUGCAAGUGU--CUGUACUUUUACUUUCAUUUAUCAAAAGUGGAGGCGUCAUC-UCUUUUGGCCAAAA--AAAACAAAAAAAUGUGGAA ..(((((..((((..((((((((.(((((.--...))))).))).)))))..))))..))))).(((......-.......)))....--................... ( -22.82) >DroMoj_CAF1 196242 91 - 1 UAACGCUAAUGAUCUAUGAAGUGUAAGUGCU-UUGUACUUUUACUUUCAUUUAUCAUAAAUGGAGGCGUCAUU-UGU---GCUCGACA--C-----------UGUUGAA ....((....(((..((((((((.((((((.-..)))))).))).)))))..)))((((((((.....)))))-)))---))(((((.--.-----------.))))). ( -20.50) >DroAna_CAF1 201377 91 - 1 CUCCGCUAAUGAUCUAUGAAGUGCAAGUGU--UUGUACUUUUACUUUCAUUUAUCAAAAGUGGAGGCGUCAUC-UCU--UGGCCGAGA--C-----------AGUGGAA (((((((..((((..((((((((.(((((.--...))))).))).)))))..))))..)))))))((....((-((.--.....))))--.-----------.)).... ( -27.00) >consensus CAACGCUAAUGAUCUAUGAAGUGCAAGUGU__UUGUACUUUUACUUUCAUUUAUCAAAAAUGGAGGCGUCAUC_UCU___GCCCAAGA__C___________AGUGGAA ...((((..((((..((((((((.(((((......))))).))).)))))..))))........))))......................................... (-14.23 = -13.82 + -0.41)


| Location | 7,528,542 – 7,528,639 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 73.86 |
| Mean single sequence MFE | -21.55 |
| Consensus MFE | -14.28 |
| Energy contribution | -13.90 |
| Covariance contribution | -0.39 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979414 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 7528542 97 - 23771897 -AAAUAAGAAGAA--AAACGAA--AAGUAA---C--CACCGCUAAUGAUCUAUGAAGUGCAAGUGU--CUGUACUUUUACUUUCAUUUAUCAA-------AAGUGGAGGCGUCAUC -............--.......--......---(--(.(((((..((((..((((((((.(((((.--...))))).))).)))))..)))).-------.))))).))....... ( -21.60) >DroVir_CAF1 217165 96 - 1 ------ACAAGCGG-CAGCAACAG-CACAA---C--UAACGCUAAUGAUCUAUGAAGUGUAAGUGUUUUUGUACUUUUACUUUCAUUUAUCAU-------AAAUGGAGGCGUCAUU ------......((-(.((..(((-(....---.--....))).(((((..((((((((.(((((......))))).))).)))))..)))))-------.....)..)))))... ( -19.30) >DroPse_CAF1 184790 103 - 1 -CAACCACCA---ACCACCAAC--CACCGA---C--CACCGCUAAUGAUCUAUGAAGUGUGAGUGU--UUGUACUUUUACUUUCAUUUAUCACAAAUCACAAAUGGAGGCGGCAUC -.........---.........--......---.--..(((((..((((..((((((((.(((((.--...))))).))).)))))..))))....(((....))).))))).... ( -18.70) >DroGri_CAF1 170236 102 - 1 AACAGCACAACCAA-CAACAAAAACAACAA---G--CAACGCUAAUGAUCUGUGAAGUGUAAGUAUU-UUGUACUUUUACUUUCAUUUAUCAU-------AAAUGGAAGCGUCAUU ....((....(((.-..............(---(--(...))).(((((..((((((((.((((((.-..)))))).))).)))))..)))))-------...)))..))...... ( -17.90) >DroMoj_CAF1 196261 96 - 1 ------ACAAGUGG-CAGCGGCGUCCACAA---G--UAACGCUAAUGAUCUAUGAAGUGUAAGUGCU-UUGUACUUUUACUUUCAUUUAUCAU-------AAAUGGAGGCGUCAUU ------...(((((-(.(((((((......---.--..))))).(((((..((((((((.((((((.-..)))))).))).)))))..)))))-------........)))))))) ( -26.30) >DroAna_CAF1 201397 104 - 1 -AAUAUACAAAAAAAAAACAAA--AACUAAAAGCCACUCCGCUAAUGAUCUAUGAAGUGCAAGUGU--UUGUACUUUUACUUUCAUUUAUCAA-------AAGUGGAGGCGUCAUC -.....................--........(((..((((((..((((..((((((((.(((((.--...))))).))).)))))..)))).-------.)))))))))...... ( -25.50) >consensus _AAA__ACAAGAAA_CAACAAA__CAACAA___C__CAACGCUAAUGAUCUAUGAAGUGUAAGUGU__UUGUACUUUUACUUUCAUUUAUCAU_______AAAUGGAGGCGUCAUC ......................................(((((..((((..((((((((.(((((......))))).))).)))))..))))...............))))).... (-14.28 = -13.90 + -0.39)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:39:24 2006