
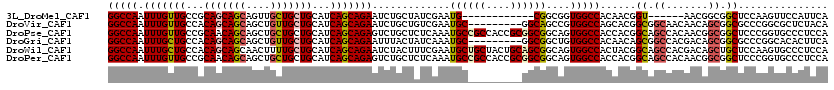
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 7,423,962 – 7,424,064 |
| Length | 102 |
| Max. P | 0.982207 |

| Location | 7,423,962 – 7,424,064 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.19 |
| Mean single sequence MFE | -50.19 |
| Consensus MFE | -32.71 |
| Energy contribution | -33.35 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.17 |
| Mean z-score | -3.24 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.91 |
| SVM RNA-class probability | 0.982207 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 7423962 102 + 23771897 GGCCAAUUUGUUGCCGCAGCAGCAGUUGCUGCUGCAUCAGCAGAAUCUGCUAUCGAAUG------------CGGCGGUGGCCACAACGGU------AACGGCGGCUCCAAGUUCCAUUCA ((..((((((..(((((.......((..(((((((((((((((...)))))...).)))------------))))))..)).....((..------..)))))))..))))))))..... ( -41.40) >DroVir_CAF1 84599 111 + 1 GGCCAAUUUGUUGCCACAGCAGCAGCUGUUGCUGCAUCAGCAGAAUCUGCUGUCGAAUGC---------GGCAGCCGUGGCCAGCACGGCGGCAACAACAGCGGCGCCCGGCGCUCUACA .((....(((((((((((((....)))))((((((((((((((...))))))....))))---------))))((((((.....))))))))))))))..))(((((...)))))..... ( -53.60) >DroPse_CAF1 72026 120 + 1 GGCCAAUUUGUUGCCGCAACAGCAGCUGCUGCUGCAUCAGCAGAGUCUGCUCUCAAAUGCCGCCACCGCGGCGGCAGUGGCCACCACGGCAGCCACAACGGCGGCUCCCGGUGCCCUCCA ((((.....((.(((((...(((((((.((((((...)))))))).)))))......(((((((.....)))))))))))).))..(((.((((.(....).)))).)))).)))..... ( -55.70) >DroGri_CAF1 67653 111 + 1 GGCCAAUUUGCUGCCACAGCAGCAGCUGUUGCUGCAUCAGCAGAAUUUACUAUCAAAUGC---------GGCGGCUGUGGCCACAACAGCGGCCACGACAGCGGCGCCCGGCACACUUCA .(((....((((((....))))))(((((((((((....(((...............)))---------.))))).((((((.(....).)))))))))))))))............... ( -46.06) >DroWil_CAF1 92248 120 + 1 GGCCAAUUUGCUGCCACAGCAGCAACUUUUGCUGCAUCAGCAGAAUCUACUUUCGAAUGCUGCUACUGCAGCGGCAGUGGCCACUACGGCAGCCACGACAGCUGCUCCAAGUGCCCUCCA .(((...(((((((....))))))).....((((((..(((((..((.......))...)))))..)))))))))((.(((.(((..((((((.......))))))...))))))))... ( -48.70) >DroPer_CAF1 72644 120 + 1 GGCCAAUUUGUUGCCGCAACAGCAGCUGCUGCUGCAUCAGCAGAGUCUGCUCUCAAAUGCCGCCACCGCGGCGGCAGUGGCCACCACGGCAGCCACAACGGCGGCUCCCGGUGCCCUCCA ((((.....((.(((((...(((((((.((((((...)))))))).)))))......(((((((.....)))))))))))).))..(((.((((.(....).)))).)))).)))..... ( -55.70) >consensus GGCCAAUUUGUUGCCACAGCAGCAGCUGCUGCUGCAUCAGCAGAAUCUGCUAUCAAAUGC_________GGCGGCAGUGGCCACCACGGCAGCCACAACAGCGGCUCCCGGUGCCCUCCA (((((.(((((((...(((((((....)))))))...))))))).............((((((....))))))....)))))......((.((.......)).))............... (-32.71 = -33.35 + 0.64)


| Location | 7,423,962 – 7,424,064 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.19 |
| Mean single sequence MFE | -54.15 |
| Consensus MFE | -34.58 |
| Energy contribution | -35.80 |
| Covariance contribution | 1.22 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.74 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.64 |
| SVM RNA-class probability | 0.969193 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 7423962 102 - 23771897 UGAAUGGAACUUGGAGCCGCCGUU------ACCGUUGUGGCCACCGCCG------------CAUUCGAUAGCAGAUUCUGCUGAUGCAGCAGCAACUGCUGCUGCGGCAACAAAUUGGCC ..........(((..(((......------..((.((((((....))))------------))..)).((((((...))))))..((((((((....)))))))))))..)))....... ( -39.30) >DroVir_CAF1 84599 111 - 1 UGUAGAGCGCCGGGCGCCGCUGUUGUUGCCGCCGUGCUGGCCACGGCUGCC---------GCAUUCGACAGCAGAUUCUGCUGAUGCAGCAACAGCUGCUGCUGUGGCAACAAAUUGGCC ....(.((((...)))))(((.((((((((((((((.....))))))(((.---------((((....((((((...)))))))))).)))(((((....)))))))))))))...))). ( -50.90) >DroPse_CAF1 72026 120 - 1 UGGAGGGCACCGGGAGCCGCCGUUGUGGCUGCCGUGGUGGCCACUGCCGCCGCGGUGGCGGCAUUUGAGAGCAGACUCUGCUGAUGCAGCAGCAGCUGCUGUUGCGGCAACAAAUUGGCC .....(((....(..(((((...(((.((..((((((((((....))))))))))..)).)))...((.(((((.((((((((...)))))).))))))).)))))))..)......))) ( -65.00) >DroGri_CAF1 67653 111 - 1 UGAAGUGUGCCGGGCGCCGCUGUCGUGGCCGCUGUUGUGGCCACAGCCGCC---------GCAUUUGAUAGUAAAUUCUGCUGAUGCAGCAACAGCUGCUGCUGUGGCAGCAAAUUGGCC ........(((((.....(((((.((((((((....))))))))....((.---------(((((.(.(((......)))).))))).)).)))))((((((....))))))..))))). ( -48.00) >DroWil_CAF1 92248 120 - 1 UGGAGGGCACUUGGAGCAGCUGUCGUGGCUGCCGUAGUGGCCACUGCCGCUGCAGUAGCAGCAUUCGAAAGUAGAUUCUGCUGAUGCAGCAAAAGUUGCUGCUGUGGCAGCAAAUUGGCC .((.((.((((..(.((((((.....)))))))..)))).)).))(((((((((.((((((.((((....)..))).)))))).)))))).....(((((((....)))))))...))). ( -56.70) >DroPer_CAF1 72644 120 - 1 UGGAGGGCACCGGGAGCCGCCGUUGUGGCUGCCGUGGUGGCCACUGCCGCCGCGGUGGCGGCAUUUGAGAGCAGACUCUGCUGAUGCAGCAGCAGCUGCUGUUGCGGCAACAAAUUGGCC .....(((....(..(((((...(((.((..((((((((((....))))))))))..)).)))...((.(((((.((((((((...)))))).))))))).)))))))..)......))) ( -65.00) >consensus UGGAGGGCACCGGGAGCCGCCGUUGUGGCUGCCGUGGUGGCCACUGCCGCC_________GCAUUCGAGAGCAGAUUCUGCUGAUGCAGCAACAGCUGCUGCUGCGGCAACAAAUUGGCC ...............(((((.((.((((((((....)))))))).)).(((((....))))).......(((((...)))))...((((((.....)))))).)))))............ (-34.58 = -35.80 + 1.22)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:38:55 2006