
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 7,409,217 – 7,409,313 |
| Length | 96 |
| Max. P | 0.996574 |

| Location | 7,409,217 – 7,409,313 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 70.99 |
| Mean single sequence MFE | -40.33 |
| Consensus MFE | -26.53 |
| Energy contribution | -27.57 |
| Covariance contribution | 1.03 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.66 |
| SVM decision value | 2.71 |
| SVM RNA-class probability | 0.996516 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
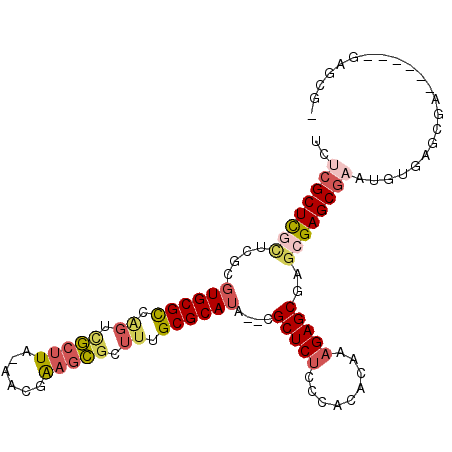
>3L_DroMel_CAF1 7409217 96 + 23771897 UCUCGCUCGUUCGCGUGCGUUAGUCGCUUA-AACGAAGCGCUGCGCGCAUA--CGCUCUCCCACACAAAGAGCGAGCGAGCGAACGUGAGCGA------GAGCGG ((((((((((((((((((((.((.(((((.-....)))))))))))))...--((((((.........)))))).....))))))..))))))------)).... ( -48.00) >DroPse_CAF1 58338 84 + 1 UCCCGCUUGCUCGAGUGCGCCAGCUACCUGCCGCUGAGUGUGUUGCGCA----CGCUCCCC-----A--GAGC--CCGAGCCAGUGCCAGAGA------GAGC-- ...((((.(((((.((((((((((........))))........)))))----)((((...-----.--))))--.))))).)))).......------....-- ( -31.20) >DroSec_CAF1 63800 102 + 1 UCUCGCUCGCUCGCGUGCGUCAGUCGAUUA-AAUGAAGCGCUUUGCGCAUA--CGCUCUCCCACACAAAGAGCGAGCGAGCGAAUGUGAGCGAGAGCGAGAGCGA ((((((((((((((((((.(((........-..))).))))....(((...--((((((.........)))))).))).))))..))))))))))((....)).. ( -44.90) >DroSim_CAF1 57511 96 + 1 UCUCGCUCGCUCGCGUGCGUCGGUCGCUUA-AAUGAAGCGCUUUGCGCAUA--CGCUCUCCCACACAAAGAGCGAGCGAGCGAAUAUGAGCGA------GAGCGA ((((((((((((((((((((..(.(((((.-....))))).)..)))))).--((((((.........)))))).))))))......))))))------)).... ( -46.20) >DroEre_CAF1 57170 87 + 1 UCUCGCUCGCACGCGUGCGCCAGUGGCUUA-AGCGAAGCACUUUGCGCAUAUGCGCUCUCCGACACAAAGAGCAUGAGAGCGC----------------G-GCGG ...(((.(((..((((((((.((((.(((.-....)))))))..))))))..))((((((...............))))))))----------------)-))). ( -33.96) >DroYak_CAF1 57602 82 + 1 UCUCGCUCGCACGCGUGCGCCAGUCGCUUA-AGCGAAGCGCUUUGCGCAUA--CGCUCUCCGAUACAAAGAGCAAGAGAGCGA----------------G-A--- ((((((((.(..((((((((...((((...-.)))).))))...))))...--.(((((.........)))))..).))))))----------------)-)--- ( -37.70) >consensus UCUCGCUCGCUCGCGUGCGCCAGUCGCUUA_AACGAAGCGCUUUGCGCAUA__CGCUCUCCCACACAAAGAGCGAGCGAGCGAAUGUGAGCGA______GAGCG_ ..((((((((....((((((.((.(((((......))))).)).))))))....(((((.........)))))..))))))))...................... (-26.53 = -27.57 + 1.03)

| Location | 7,409,217 – 7,409,313 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 70.99 |
| Mean single sequence MFE | -41.23 |
| Consensus MFE | -26.92 |
| Energy contribution | -28.98 |
| Covariance contribution | 2.06 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.83 |
| Structure conservation index | 0.65 |
| SVM decision value | 2.72 |
| SVM RNA-class probability | 0.996574 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 7409217 96 - 23771897 CCGCUC------UCGCUCACGUUCGCUCGCUCGCUCUUUGUGUGGGAGAGCG--UAUGCGCGCAGCGCUUCGUU-UAAGCGACUAACGCACGCGAACGAGCGAGA ....((------((((((..((((((..((.((((((((.....))))))))--...))(((.(((((((....-.))))).))..)))..)))))))))))))) ( -44.80) >DroPse_CAF1 58338 84 - 1 --GCUC------UCUCUGGCACUGGCUCGG--GCUC--U-----GGGGAGCG----UGCGCAACACACUCAGCGGCAGGUAGCUGGCGCACUCGAGCAAGCGGGA --....------..(((.((....((((((--((((--.-----...))))(----(((((........((((........))))))))))))))))..)).))) ( -34.50) >DroSec_CAF1 63800 102 - 1 UCGCUCUCGCUCUCGCUCACAUUCGCUCGCUCGCUCUUUGUGUGGGAGAGCG--UAUGCGCAAAGCGCUUCAUU-UAAUCGACUGACGCACGCGAGCGAGCGAGA ....((((((((..((((.(((.(((((.(((((.......))))).)))))--.))).((...((((.((...-.....))..).)))..)))))))))))))) ( -41.00) >DroSim_CAF1 57511 96 - 1 UCGCUC------UCGCUCAUAUUCGCUCGCUCGCUCUUUGUGUGGGAGAGCG--UAUGCGCAAAGCGCUUCAUU-UAAGCGACCGACGCACGCGAGCGAGCGAGA ..((..------..)).....(((((((((((((....(((((.((...(((--....)))....(((((....-.))))).)).))))).))))))))))))). ( -45.00) >DroEre_CAF1 57170 87 - 1 CCGC-C----------------GCGCUCUCAUGCUCUUUGUGUCGGAGAGCGCAUAUGCGCAAAGUGCUUCGCU-UAAGCCACUGGCGCACGCGUGCGAGCGAGA .(((-(----------------((((......((((((((...))))))))((...(((((..(((((((....-.))).)))).))))).))))))).)))... ( -36.70) >DroYak_CAF1 57602 82 - 1 ---U-C----------------UCGCUCUCUUGCUCUUUGUAUCGGAGAGCG--UAUGCGCAAAGCGCUUCGCU-UAAGCGACUGGCGCACGCGUGCGAGCGAGA ---(-(----------------((((((....((((((((...))))))))(--((((((....(((((((((.-...))))..))))).))))))))))))))) ( -45.40) >consensus _CGCUC______UCGCUCACAUUCGCUCGCUCGCUCUUUGUGUGGGAGAGCG__UAUGCGCAAAGCGCUUCGCU_UAAGCGACUGACGCACGCGAGCGAGCGAGA .....................((((((((((((((((((.....))))))).....((((.....(((((......))))).....))))...))))))))))). (-26.92 = -28.98 + 2.06)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:38:47 2006